版权声明:看我干嘛? 你又没打算转载我的博客~ https://blog.csdn.net/wjh2622075127/article/details/84190404
一. 使用PCA(主成分分析)进行降维实现数据可视化
降维的任务是要找到一个可以保留数据本质特征的低维矩阵来表示高维数据, 通常用于辅助数据可视化的工作.
下面我们使用主成分分析(principal component analysis, PCA)方法, 这是一种快速线性降维技术. 模型返回两个主成分, 用二维数据表示鸢尾花的4维数据.
1. 首先导入数据
import seaborn as sns
sns.set()
iris = sns.load_dataset('iris')
print(iris) # 输出查看数据
2. 划分数据, 特征和标签
X_iris = iris.drop('species', axis=1)
y_iris = iris['species']
3. 选择PCA模型, 进行拟合
from sklearn.decomposition import PCA
model = PCA(n_components=2) # 设置超参数, 初始化模型
model.fit(X_iris) # 进行拟合
X_2D = model.transform(X_iris) # 将数据转化为二维
4. 图形可视化
思路是将二维数据插入到DataFrame中, 然后用seaborn的lmplot方法绘制图形.
iris['PCA1'] = X_2D[:, 0]
iris['PCA2'] = X_2D[:, 1]
sns.lmplot("PCA1", "PCA2", hue='species', data=iris, fit_reg=False)
结果展示:
可以看到PCA降维成功地将鸢尾花的种类在视觉上进行划分.
原先的数据集是4维, 难以进行可视化, 降维后在2维上实现可视化.

二. 使用高斯混合模型对鸢尾花数据进行聚类
1. 首先导入数据
import seaborn as sns
sns.set()
iris = sns.load_dataset('iris')
print(iris) # 输出查看数据
2. 划分数据, 特征和标签
X_iris = iris.drop('species', axis=1)
y_iris = iris['species']
3. 使用主成分分析进行降维(为了可视化为2维数据)
from sklearn.decomposition import PCA
model = PCA(n_components=2) # 设置超参数, 初始化模型
model.fit(X_iris) # 进行拟合
X_2D = model.transform(X_iris) # 将数据转化为二维
iris['PCA1'] = X_2D[:, 0]
iris['PCA2'] = X_2D[:, 1]
4. 选择高斯混合模型
from sklearn.mixture import GMM
model = GMM(n_components=3, covariance_type='full') # 设置超参数
model.fit(X_iris) # 拟合数据
y_gmm = model.predict(X_iris) # 确定簇标签
5. 数据可视化
iris['cluster'] = y_gmm
sns.lmplot('PCA1', 'PCA2', data=iris, hue='species', col='cluster', fit_reg=False)
如图, 数据根据簇的不同被成三类.
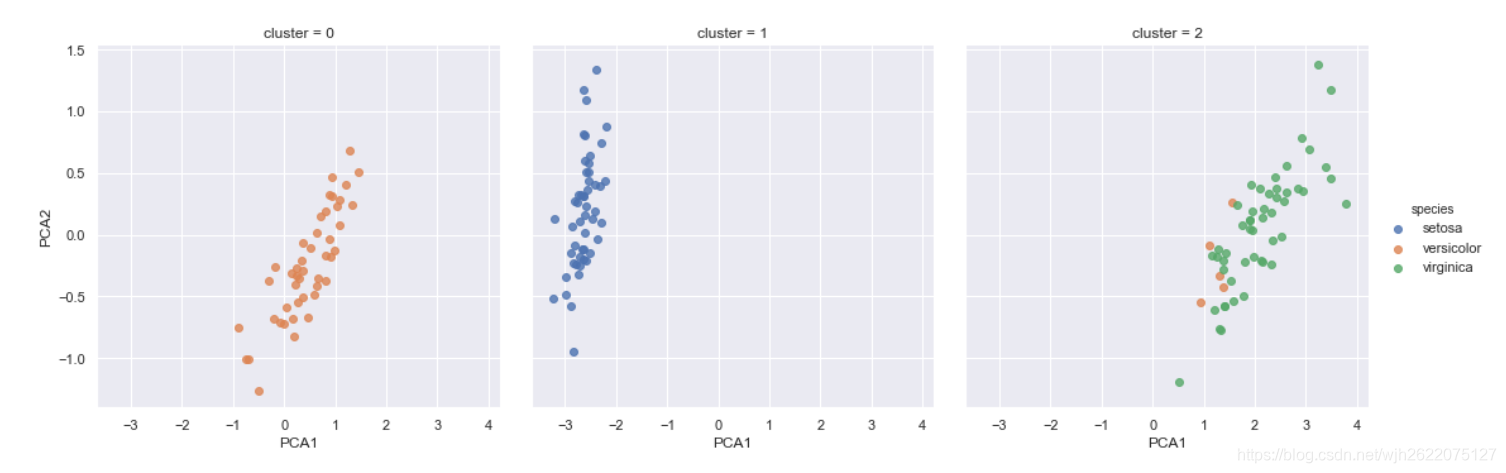
可以看到图一和图二的分类基本正确, 图三有少数的点是属于图二.