这一块的每一个小点都可以引申出很多的东西,所以先做一个大概,用以备忘,持续更新。
*一般过程:
(1)数据采集
数据采集是最基本也很耗时间的工作。比如对于具体的工程事件,需要考虑采集哪些类型的数据?需要哪些属性?需要多少数据支撑?然后再实际去采集这些数据,离线采集?在线获取?
(2)数据预处理
基于最后期望的目标结果,对于当前的数据,如何处理当前的数据类型,比如如何处理有序变量?如何处理无序变量?数据可信吗?有缺失值吗?应该正则化吗?离群值,噪音点怎么处理?特征工程?偏差检测?样本过滤?等等等等,数据清洗将会耗去大量的时间。
(3)解释模型结果和调整模型
根据数据,选用合适的模型,然后不断的根据结果微调模型,改进模型。或是在发现某些问题后,再返回第二步,重做第三步。不断的评估模型以完成最好的结果。
如何进行特征选择?
PCA:寻找表示数据分布的最优子空间,将原始数据降维,并提取不相关的部分。
LDA:寻找可分性判据最大的子空间。使得降维后类内散度最小,类间散度最大
ICA:将原始数据降维并提取出相互独立的属性,ICA理论的目标就是通过X求得一个分离矩阵W,使得W作用在X上所获得的信号Y是独立源S的最优逼近。ICA相比与PCA更能刻画变量的随机统计特性,且能抑制高斯噪声。
CCA,找到两组基,使得两组数据在这两组基上的投影相关性最大,用来描述两个高维变量之间的线性关系
特征选择:
包裹式:把最终机器学习模型的表现作为特征选择的重要依据,一步步筛选特征。目前比较常用的一种包裹式特征选择法为递归特征消除法,其原理是使用一个基模型(如:随机森林、逻辑回归等)进行多轮训练,每轮训练结束后,消除若干权值系数较低的特征,再基于新的特征集进行新的一轮训练。
过滤式:先对数据集进行特征选择,然后再进行模型的训练。常用的过滤式特征选择方法有Pearson相关系数法、方差选择法、假设检验、互信息法等。
嵌入式:与包裹式特征选择法最大的不同是,嵌入式方法是将特征选择过程与模型的训练过程结合为一体。
常用的嵌入式特征选择方法有基于正则化项的特征选择法(如:Lasso)和基于树模型的特征选择法(如:GBDT)。
高级特征运用加减乘除处理。
数据压缩–三大降维法则:
主成分分析,自编码,t-SNE等。
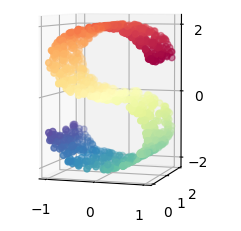
@@@PCA是一种线性算法。 它不能解释特征之间的复杂多项式关系。而t-SNE是基于在邻域图上随机游走的概率分布,可以在数据中找到其局部结构关系,最先出现在流型学习中,算法核心在于它的“距离”度量,即它将数据点之间的相似度转换为概率,以KL散度做评价标准。
import numpy as np
from sklearn.manifold import TSNE
X = np.array([[0, 0, 0], [0, 1, 1], [1, 0, 1], [1, 1, 1]])#简单的三维数据
tsne = TSNE(n_components=2)
tsne.fit_transform(X)
print(tsne.embedding_)
from time import time
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from matplotlib.ticker import NullFormatter
from sklearn import manifold, datasets
n_points = 1000
X, color = datasets.samples_generator.make_s_curve(n_points, random_state=0)
n_neighbors = 10
n_components = 2
ax = fig.add_subplot(211, projection='3d')
ax.scatter(X[:, 0], X[:, 1], X[:, 2], c=color, cmap=plt.cm.Spectral)
ax.view_init(4, -72) # 自定义3D显示视角
t0 = time()
tsne = manifold.TSNE(n_components=n_components, init='pca', random_state=0)#使用TSNE
Y = tsne.fit_transform(X)
t1 = time()#计算时间,可以看到号称最好降维技术的TNSE在使用时的最大弊病
print("t-SNE: %.2g sec" % (t1 - t0))
ax = fig.add_subplot(2, 1, 2)
plt.scatter(Y[:, 0], Y[:, 1], c=color, cmap=plt.cm.Spectral)
plt.title("t-SNE (%.2g sec)" % (t1 - t0))
ax.xaxis.set_major_formatter(NullFormatter()) # 设置标签显示为空
ax.yaxis.set_major_formatter(NullFormatter())
plt.show()
@@@PCA 和 t-SNE 是方法,而自编码器则是一系列的方法(中间层可以自由任我们设置与控制)。
如何数据预处理?
目标是:准确性,完整性,一致性,时效性,可信性,可解释性。
数据清洗:处理缺失值,光滑噪音数据,识别离群点,错误点,修正逻辑。
【多用聚类和异常点检测】
数据集成:多个数据源可能对同一属性有不同的名字而造成的冗余,冲突值,重复项等。
数据规约:降维,数据压缩,数量规约,以简化数据集。
数据变换:规范化,数据离散化,概念分层,粒度,数据类型变换,属性构造。
【不平衡采用采样法或者权重法】
具体方法有:
归一化:对数据集进行区间缩放,缩放到[0,1]的区间内,把有单位的数据转化为没有单位的数据,即统一数据的衡量标准,消除单位的影响。
from sklearn import preprocessing#使用preprocessing
import numpy as np
X = [[ 1., -1., 2.],
[ 2., 0., 0.],
[ 0., 1., -1.]]
X_normalized = preprocessing.normalize(X, norm='l2')
print(X_normalized)
normalizer = preprocessing.Normalizer().fit(X) #以便之后对测试集进行相同的转换
normalizer.transform([[-1., 1., 0.]])
输出为:
array([[ 0.40824829, -0.40824829, 0.81649658],
[ 1. , 0. , 0. ],
[ 0. , 0.70710678, -0.70710678]])
array([[-0.70710678, 0.70710678, 0. ]])
标准化:标准化是在不改变原数据分布的前提下,将数据按比例缩放,使之落入一个限定的区间,使数据之间具有可比性。如有:【但是实际上这样通常忽略分布的具体形态,数据转换仅指,减去每个特征的平均值,再除以他们的标准差。】
1.z-score标准化:这是最常见的特征预处理方式,基本所有的线性模型在拟合的时候都会做 z-score标准化。具体的方法是求出样本特征x的均值mean和标准差std,然后用(x-mean)/std来代替原特征。这样特征就变成了均值为0,方差为1了。在sklearn中,我们可以用StandardScaler来做z-score标准化。当然,如果我们是用pandas做数据预处理,可以自己在数据框里面减去均值,再除以方差,自己做z-score标准化。
2.max-min标准化:也称为离差标准化,预处理后使特征值映射到[0,1]之间。具体的方法是求出样本特征x的最大值max和最小值min,然后用(x-min)/(max-min)来代替原特征。如果我们希望将数据映射到任意一个区间[a,b],而不是[0,1],那么也很简单。用(x-min)(b-a)/(max-min)+a来代替原特征即可。在sklearn中,我们可以用MinMaxScaler来做max-min标准化。这种方法的问题就是如果测试集或者预测数据里的特征有小于min,或者大于max的数据,会导致max和min发生变化,需要重新计算。所以实际算法中, 除非你对特征的取值区间有需求,否则max-min标准化没有 z-score标准化好用。
3.L1/L2范数标准化:通常情况下,范数标准化首选L2范数标准化。在sklearn中,我们可以用Normalizer来做L1/L2范数标准化。
from sklearn import preprocessing#使用preprocessing
import numpy as np
X = np.array([[ 1., -1., 2.],
[ 2., 0., 0.],
[ 0., 1., -1.]])
X_scaled = preprocessing.scale(X)
print(X_scaled)
print(X_scaled.mean(axis=0))
print(X_scaled.std(axis=0))
scaler = preprocessing.StandardScaler().fit(X)#计算训练集的平均值和标准差,以便之后对测试集进行相同的转换。
print(scaler.transform([[-1., 1., 0.]]) )
输出结果为:
array([[ 0. , -1.22474487, 1.33630621],
[ 1.22474487, 0. , -0.26726124],
[-1.22474487, 1.22474487, -1.06904497]])
调整后的数据平均值为0,方差为1。
array([ 0., 0., 0.])
array([ 1., 1., 1.])
array([[-2.44948974, 1.22474487, -0.26726124]])
离散化:把连续的数值型特征进行分段,将落在每一个分段内的数据赋予一个新的统一的符号或数值。可采用相等步长或相等频率等方式进行离散化。
二值化:将数值型数据转换为0和1两个值,例如通过设定一个阈值,当特征的值大于该阈值时,转换为1,小于或等于该阈值时转换为0。二值化的目的在于简化数据,有些时候还可以消除数据中的“杂音”,例如图像数据。
from sklearn import preprocessing#使用preprocessing
import numpy as np
X = [[ 1., -1., 2.],
[ 2., 0., 0.],
[ 0., 1., -1.]]
binarizer = preprocessing.Binarizer().fit(X)
binarizer.transform(X)
binarizer = preprocessing.Binarizer(threshold=1.1)#默认阈值为0
binarizer.transform(X)
修改阈值可以看到:
array([[ 1., 0., 1.],
[ 1., 0., 0.],
[ 0., 1., 0.]])
array([[ 0., 0., 1.],
[ 1., 0., 0.],
[ 0., 0., 0.]])
哑编码:One-Hot Encoding,其作用是将特征进行量化。
from sklearn import preprocessing#使用preprocessing
import numpy as np
enc = preprocessing.OneHotEncoder()
enc.fit([[0, 0, 3], [1, 1, 0], [0, 2, 1], [1, 0, 2]])
enc.transform([[0, 1, 3]]).toarray()#自动提取
enc = preprocessing.OneHotEncoder(n_values=[2, 3, 4])#防止丢失值,明确设置value。如此处特征分别有2,3,4个值
enc.fit([[1, 2, 3], [0, 2, 0]])
enc.transform([[1, 0, 0]]).toarray()
array([[ 1., 0., 0., 1., 0., 0., 0., 0., 1.]])
array([[ 0., 1., 1., 0., 0., 1., 0., 0., 0.]])
**如何处理缺失数据? **
产生原因:数据无法获取,被遗漏,属性不可用,或者不重要等。
解决方法:
1.删除缺失行。从而得到一个完备的信息表。以数据量来换取信息的完备,会造成资源的大量浪费,而且丢弃了大量隐藏信息,甚至是某些很重要的信息
2.人工填写。特殊全局常量值,平均值,寻找最相似的对象补齐,K近邻平均补齐,回归,EM,寻找属性间的关系(决策树),所有的可能值填充。
import numpy as np
from sklearn.preprocessing import Imputer
imp = Imputer(missing_values='NaN', strategy='mean', axis=0)#缺失值由平均值处理
imp.fit([[1, 2], [np.nan, 3], [7, 6]])
Imputer(axis=0, copy=True, missing_values='NaN', strategy='mean', verbose=0)
X = [[np.nan, 2], [6, np.nan], [7, 6]]
print(imp.transform(X))
*如何评估模型?

性能度量:
正确率,错误率,查全率,查准率,F1
方差,误差,R平均值。
ROC曲线
AUC(ROC曲线下面积)
Lift(提升)和Gain(增益)
K-S图
基尼系数



*如何处理稀疏数据?
流型降维,数据平滑。
import scipy.sparse as sp
X = sp.csc_matrix([[1, 2], [0, 3], [7, 6]])
imp = Imputer(missing_values=0, strategy='mean', axis=0)#Imputer也支持稀疏矩阵
imp.fit(X)
X_test = sp.csc_matrix([[0, 2], [6, 0], [7, 6]])
print(imp.transform(X_test))
[[ 4. 2. ]
[ 6. 3.66666667]
[ 7. 6. ]]
但是这里的缺失值被编码为0,被隐性地储存在了矩阵中。当缺失值个数比观察值多很多的时候,这个形式才比较适用。