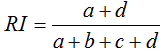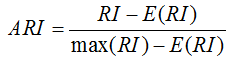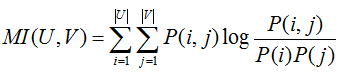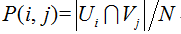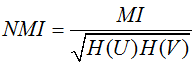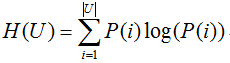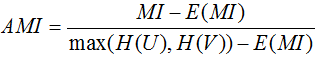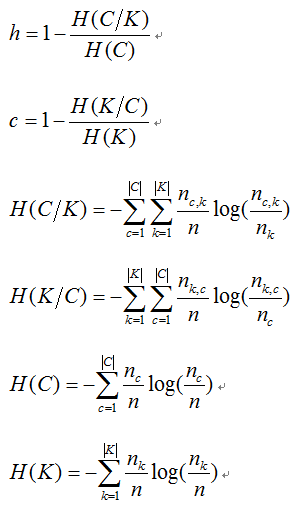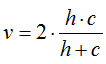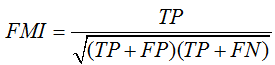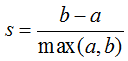1、调整兰德指数(Adjusted Rand Index)
兰德指数需要给定类别信息C,假设K是聚类结果,兰德指数表达式如下
a为在C中为同一类且在K中也为同一类别的数据点对数
b为在C中为同一类但在K中却隶属于不同类别的数据点对数
c为在C中不在同一类但在K中为同一类别的数据点对数
d为在C中不在同一类且在K中也不属于同一类别的数据点对数
RI的取值范围为[0,1],值越大意味着聚类结果与真实情况越匹配,调整兰德指数需要数据标记。
调整兰德指数解决对于两个随机的划分,其兰德系数值不是一个接近于0的常数, 表达式如下
ARI的取值范围为[-1,1],值越大意味着聚类结果与真实情况越匹配。可用于聚类算法之间的比较。
sklearn接口:metrics.adjusted_rand_score(labels_true, labels_pred)
2、调整互信息(Adjusted Mutual Information)
互信息的表达式如下
其中
标准化互信息
其中
调整互信息
MI和NMI的取值范围为[0,1],AMI的取值范围为[-1,1],值越大意味着聚类结果与真实情况越匹配,调整互信息需要数据标记。
sklearn接口:metrics.adjusted_mutual_info_score(labels_true, labels_pred)
3、同质性Homogeneity、完整性completeness、调和平均V-measure
其中,n是样本总数,nc和nk分别属于类c和类k的样本数,而nc,k是从类c划分到类k的样本数量。
取值范围为[0,1],完全随机标签并不总是产生相同的完整性和均匀性的值,所得调和平均值V-measure也不相同
sklearn接口:metrics.homogeneity_score(labels_true, labels_pred)
sklearn接口:metrics.completeness_score(labels_true, labels_pred)
sklearn接口:metrics.v_measure_score(labels_true, labels_pred)
4、Fowlkes-Mallows scores
sklearn接口:metrics.fowlkes_mallows_score(labels_true, labels_pred)
5、轮廓系数(Silhouette Coefficient)
轮廓系数适用于实际类别信息未知的情况。对于单个样本,设a是与它同类别中其他样本的平均距离,b是与它距离最近不同类别中样本的平均距离,其轮廓系数为:
对于一个样本集合,它的轮廓系数是所有样本轮廓系数的平均值。轮廓系数的取值范围是[-1,1],同类别样本距离越相近不同类别样本距离越远,分数越高。
sklearn接口:metrics.silhouette_score(X, labels, metric=‘euclidean’)
前四种方法要求数据有标记,轮廓系数不要求数据有标记
写博客的目的是学习的总结和知识的共享,如有侵权,请与我联系,我将尽快处理