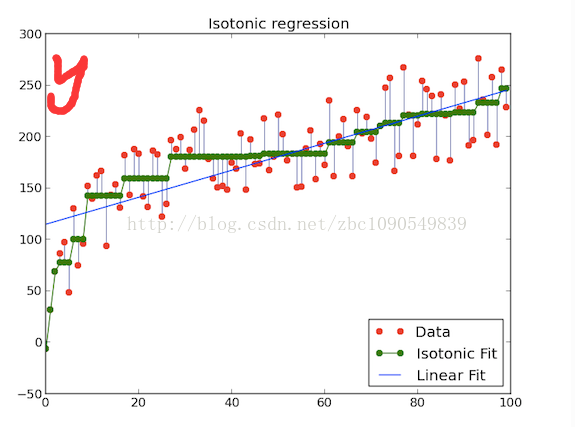
IsotonicRegression是Spark1.3版本引入的一个带约束的回归模型。IsotonicRegression又称保序回归,保序回归确保拟合得到一个非递减逼近函数的条件下 最小化均方误差,相关的介绍可以阅读http://fa.bianp.net/blog/2013/isotonic-regression/,借用该文的一篇图来说明一下
图中横轴为序号,纵轴y为输入数据,假设x为要拟合的数据,保序拟合的目标是序号越大的y,其拟合值x至少不比前面序号的x值小,即拟合后的值不递减。在满足这个条件的同时,拟合的均方误差应该尽可能小。上述优化目标用公式描述如下
上面的优化目标一般用 PAVA(pool adjacent violators algorithm)算法求解。Spark也正是采用的此算法进行模型求解的。
下面来看具体的实现
IsotonicRegressionModel的定义比较简单,主要是实现了predict方法。
predict方法主要用到了二分查找算法(查找预测数据的orderindex即序号值),然后采用线性插值算法来估计orderindex对应的值作为最终的预测值。
def predict(testData: Double): Double = {
def linearInterpolation(x1: Double, y1: Double, x2: Double, y2: Double, x: Double): Double = {
y1 + (y2 - y1) * (x - x1) / (x2 - x1)
}
//二分查找法获得预测数据的index
val foundIndex = binarySearch(boundaries, testData)
val insertIndex = -foundIndex - 1
//线性插值的方法来获得预测值
if (insertIndex == 0) {
predictions.head
} else if (insertIndex == boundaries.length) {
predictions.last
} else if (foundIndex < 0) {
linearInterpolation(
boundaries(insertIndex - 1),
predictions(insertIndex - 1),
boundaries(insertIndex),
predictions(insertIndex),
testData)
} else {
predictions(foundIndex)
}
}再来看模型求解过程 IsotonicRegression类,该类实现了递增保序回归和递减保序回归,通过setIsotonic(isotonic: Boolean)方法设定。
模型迭代求解的接口通过run方法对外暴露
def run(input: RDD[(Double, Double, Double)]): IsotonicRegressionModel = {
val preprocessedInput = if (isotonic) {
input
} else {
input.map(x => (-x._1, x._2, x._3))
}
val pooled = parallelPoolAdjacentViolators(preprocessedInput)
val predictions = if (isotonic) pooled.map(_._1) else pooled.map(-_._1)
val boundaries = pooled.map(_._2)
new IsotonicRegressionModel(boundaries, predictions, isotonic)
}parallelPoolAdjacentViolators来实现PAVA算法的并行化迭代计算
private def parallelPoolAdjacentViolators(
input: RDD[(Double, Double, Double)]): Array[(Double, Double, Double)] = {
val keyedInput = input.keyBy(_._2)
val parallelStepResult = keyedInput
.partitionBy(new RangePartitioner(keyedInput.getNumPartitions, keyedInput))
.values
.mapPartitions(p => Iterator(p.toArray.sortBy(x => (x._2, x._1))))
.flatMap(poolAdjacentViolators)
.collect()
.sortBy(x => (x._2, x._1)) // Sort again because collect() doesn't promise ordering.
poolAdjacentViolators(parallelStepResult)
}这里实现了PAVA算法的并行化计算
Spark的PAVA算法分成了两个步骤:
1、按序号分区运行PAVA算法,得到每个分区上的保序回归模型:
具体来看,Spark默认使用HashPartitioner进行数据分区。由于需要分区与分区之间的序号是有序的,这样便于后面flatMap(poolAdjacentViolators)对各个分区分别进行PAVA算法,使用了RangePartitioner进行分区。有序分区完成后,调用flatMap(poolAdjacentViolators)对每个分区拟合一个保序回归模型。 poolAdjacentViolators是一个PAVA算法的具体实现,这个和分布式无关了。
2、对各个分区的保序回归模型合并:
由于各个分区之间的序号是有序的,因此每个分区使用PAVA算法求解的模型直接按区间顺序合并,合并得到的结果是序号值有序、值无序的一个数组parallelStepResult,再调用一次poolAdjacentViolators方法对parallelStepResult拟合就得到了最终的保列回归模型。
并行化的PAVA算法是map-reduce思想的典型体现。