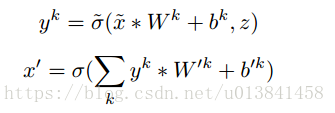Deep Supervised and Convolutional Generative Stochastic Network for Protein Secondary Structure Prediction论文笔记
用于蛋白质二级结构预测的深度监督和卷积生成随机网络
Abstract
预测蛋白质二级结构是蛋白质结构预测中的基本问题。在这里,我们提出了一种新的监督生成随机网络(GSN)的方法来预测具有深层次表示的局部二级结构。 GSN是最近提出的深度学习技术(Bengio&Thibodeau-Laufer,2013),用于全球培养深度生成模型。我们提出GSN的监督扩展,它学习马尔可夫链从条件分布中采样,并将其应用于蛋白质结构预测。为了将模型扩展到全尺寸,高维数据,如具有数百个氨基酸的蛋白质序列,我们引入了卷积体系结构,允许跨多层次表示的有效学习。我们的架构专注于预测结构化的低级标签,通过模型学习的低级和高级表示。在我们的申请中,这对应于标记每个氨基酸残基的二级结构状态。我们在不同的非同源蛋白质组上训练和测试该模型,所述非同源蛋白质具有小于30%的序列同一性。我们的模型在CB513数据集上实现了66.4%的Q8准确度,优于之前报告的这一具有挑战性的二级结构预测问题的最佳性能64.9%(Wang等,2011)。
1. Introduction
理解蛋白质序列和结构之间的复杂依赖性是计算生物学中最大的挑战之一(Cheng等人,2008)。 虽然人们普遍认为氨基酸序列含有足够的信息来确定蛋白质的三维结构,但基于序列预测蛋白质结构是极其困难的。 了解蛋白质结构对于分析蛋白质功能和药物设计等应用至关重要。 蛋白质二级结构预测决定了氨基酸残基的局部区段的结构状态,如α-螺旋和“β-链”,它为进一步阐明蛋白质的三维结构提供了重要信息。因此它也被用作 由许多其他蛋白质序列和结构分析算法输入。
蛋白质二级结构预测已经用机器学习方法进行了广泛的研究(Singh,2005)。该领域的关键挑战是预测没有已知3D结构的紧密同源物的蛋白质序列。自(Qian&Sejnowski,1988)的早期工作以来,神经网络已被广泛应用,并且是许多最成功方法的核心组成部分(Rost&Sander,1993; Jones,1999; Baldi等,1999)。通过使用来自多序列比对的序列谱(Rost&Sander,1993)或来自PSI-BLAST的位置特异性评分矩阵(Jones,1999),利用进化信息实现了对二级结构预测的最显着改进。其他发展包括使用双向递归神经网络(BRNN)更好地捕获空间依赖性(Baldiet al。,1999; Pollastri等,2002),概率图形模型(Schmidler等,2000; Chu等,2004; van der Maaten等,2011),或神经网络和图形模型的组合,如条件神经域(CNF),它将基于窗口的神经网络与条件随机场(CRF)相结合(Peng et al。,2009; Wang et al 。,2011)。二级结构通常分为8个州(Kabsch&Sander,1983)或进一步合并为3个州(Singh,2005)。大多数二级结构预测研究都集中在粗粒度三态二级结构预测上。实现细粒度8态二次二次预测,虽然它揭示了比三态预测更多的结构细节,但却是一个更具挑战性和更少解决的问题(Pollastri等,2002; Wang等,2011)。在这里,我们解决蛋白质的8状态分类问题,没有已知结构的紧密同系物。
人们普遍认为,从整个蛋白质序列引入全局信息对于进一步改善局部二级结构标记的预测性能至关重要,因为二级结构形成依赖于局部和长程相互作用。例如,二级结构状态“β链通过与其他”β链形成的氢键稳定,所述β链可以在蛋白质序列中彼此远离。诸如BRNN之类的许多现代方法可以捕获某种形式的空间依赖性,但它们在捕获复杂的空间依赖性方面仍然受到限制。据我们所知,这些方法都没有学习和利用深层次表示,这使得构建更好的智能系统能够解释各种类型的其他复杂结构化自然数据,如图像,语音和文本。具有深度代表性的模型在捕获这些数据中的复杂依赖性方面非常成功(Bengio等,2013a; Salakhutdinov&Hinton,2009)。自动学习功能对蛋白质特别有用,因为我们缺乏手工制作涉及多种氨基酸的中高级特征的必要直觉或知识。
在这项工作中,我们引入了一系列技术来应对深度学习中蛋白质二级结构预测的挑战,并且我们证明了它们在8态蛋白质二级结构预测方面优于先前方法的准确性。虽然我们在这里关注二级结构预测应用,但我们的方法是通用的,可以应用于计算生物学内外的广泛问题。首先,我们将最近开发的生成随机网络(GSN)技术扩展到监督结构化输出预测(Bengio等,2013a)。受监督的GSN学习马尔可夫链以从输入数据条件下的输出分布中进行抽样,并且像GSN一样,在学习具有隐藏层的深层模型时,它避免了对隐藏变量的难以处理的显式边缘化。受监督的GSN相对于GSN的优势类似于条件随机场(CRF)优于马尔可夫随机场(MRF)或判别式与生成性分类器的优势,即,我们将资源集中在对我们期望的预测很重要的依赖性上。 ,或以数据为条件的标签分发。受监督的GSN可用于产生每种蛋白质的所有氨基酸残基的二级结构状态的结构化输出。
我们的方法的另一个重要元素是,能够学习全尺寸数据的分层表示,如含有数百种氨基酸的蛋白质序列,正在为GSN引入卷积结构,允许学习多层次的层次表示。 卷积体系结构还允许在不同空间位置处的高级和低级特征与特征之间的有效通信。 卷积GSN非常适合于对低级或本地信息敏感的预测,同时通过高级或全局信息进行通知。
2. Preliminaries
2.1. Generative Stochastic Networks
生成随机网络(GSN)是最近提出的模型,其利用新的非常规方法来学习数据分布的生成模型,而无需明确指定概率图形模型,并允许通过反向传播的全局训练来学习深度生成模型。 生成随机网络学习马尔可夫链以从数据分布P(X)中进行采样。 在训练期间,GSN训练一个随机计算图来重建输入X,它推广了广义去噪自动编码器(Bengio et al。,2013b)。 GSN的不同之处在于它允许计算图具有潜在状态,并且不仅将噪声注入到输入中,而且还将噪声注入到中间计算中。
与其他深度生成模型(如深Boltzmann机器)相比,GSN的一个主要优点是它避免了对潜在变量的难以推断的推理和显式边缘化,因为它没有明确地学习概率图形模型的参数,而是学会直接从数据中抽样 分配。 GSN还通过反向传播重建成本享受可行的全局训练。
以下证明(Bengio et al。,2013b)和(Bengio et al。,2013a)作为广义去噪自动编码器和生成随机网络的理论基础:
理论1.如果Pθ(X | X~)是真实条件分布P(X | X~)的一致估计,而Tn定义了不可约和遍历的马尔可夫链,那么作为n - > infity,它的平稳分布 生成的样本π(X)收敛于数据生成分布P(X)。
在(Bengio et al。,2013a)中,该结果扩展为引入潜在状态Ht并用Ht +1~1Pθ1(H | Ht,Xt)和Xt +1~Pθ2(X | Ht)代替加法噪声和去噪算子。 分别为+1)。
定理2.让训练数据X~P(X)和独立噪声Z~P(Z)并引入一系列潜在变量H,通过函数f迭代定义Ht =fθ1(Xt-1,Zt-1,Ht- 1)对于给定的Xt序列。 考虑训练的模型Pθ2(X |fθ1(X,Zt-1,Ht-1))(在θ1和θ2上),使得对于给定的θ1,它是真实P(X | H)的一致估计量。 考虑上面定义的马尔可夫链并假设它收敛于X和H上的平稳分布πn并且具有边际πn(X),即使在训练样本数n - >∞的极限内也是如此。 然后πn(X) - > P(X)为n - >∞。
(Bengio et al。,2013a)建议将GSN扩展到解决结构化输出问题。 在关注条件分布的同时,分享GSN的优势似乎提供了一种有吸引力的新方法,即利用深度表示来进行结构化输出预测。我们在下文中探讨了监督生成随机网络的这种思想。
2.2. Supervised Generative Stochastic Network
我们将监督的生成随机网络用于如下。 我们使用X来表示输入随机变量,使用Y来表示输出随机变量。 监督的GSN旨在捕获给定X的Y的条件依赖结构。类似于GSN,它学习从P(Y | X)采样的马尔可夫链。
设C(Y〜| Y)是将Y变换为随机变量Y的腐败过程。 设Pθ(Y | Y,X~)是一个去噪自动编码器,它计算Y给定Y和X的概率。然后我们得到以下结果。
推论1.如果Pθ(Y | Y,X~)是真实条件分布P(Y | Y,X~)的一致估计,并且Tn定义了不可约和遍历的马尔可夫链,则n - > 1, 生成的样本的静止分布π(Y | X)收敛于数据生成分布P(Y | X)。
证明。 设P(X 0)= P(Y | X); C(X~0 | X 0)= X(Y〜| Y);P(X0 | X〜0)= P(Y | Y,X~); 那么这个推论相当于定理1。
在训练期间,我们学习了估计Pθ(Y | Y,X~)的计算图。 因此,我们提供Y和X~作为输入,训练程序应尽量减少重建成本。 在给定重构分布的情况下,应该选择重建成本以将其解释为(正则化的)未损坏标签的对数似然性。
为了将监督训练推广到生成随机网络,类似于推论1,我们考虑独立噪声Z~P(Z)。 腐蚀过程现在是Ht =fθ1(Yt-1,Zt-1,Ht-1,X),其中fθ1是任意可微函数。 重构函数Pθ2(Y |fθ1(Yt-1,Zt-1,Ht-1,X))通过具有(Y,Z,X)三元组的示例的正则化条件最大似然来训练。
推论2.假设训练模型Pθ2(Y |fθ1(Yt-1,Zt-1,Ht-1,X))是真P(Y | H)和马尔可夫链Y~Pθ2的一致估计量 (Y |fθ1(Yt-1,Zt-1,Ht-1,X))收敛于静止分布π。 然后我们在定理2之后得到π(Y | X)= P(Y | X)。
为了从训练有素的监督GSN模型进行预测,我们可以通过从任意Y初始化从Pθ1,θ2(Y | X)进行采样,然后通过马尔可夫链Y->Pθ2运行采样(Y |fθ1(Yt-1,Zt-) 1,Ht-1,X))。
受监督的GSN允许灵活的网络架构和噪声形式,就像GSN一样。 它具有避免在训练和推理中隐藏变量边缘化的相同优点。 监督GSN和GSN都可以将捕获P(X)或P(Y | X)的困难学习任务转换为更容易学习重建分布的任务。
3. Algorithms
3.1. Convolutional Generative Stochastic Network
图2.显示具有2个卷积GSN层的卷积GSN架构的空间连通性架构。 每个卷积层(转换)特征连接到下层的所有特征映射,而池化层(池)特征仅连接到相应特征映射中的特征。在卷积GSN(图1)的计算图中,卷积GSN层可以包含单个卷积层(h1)或者包含池和卷积层(h2),并且池化层被认为是用于计算卷积输入的中间计算。层。
(Bengio et al。,2013a)中提出的类似DBM的原始GSN架构没有利用数据的空间结构,因此每个特征都连接到每个位置的数据,因此很难扩展到实际大小的数据。 因此,我们引入卷积GSN(图1,2)来学习层次表示,在更高层次上逐渐具有更多的全局表示。 鉴于蛋白质数据的连续性,我们在这里使用1D卷积,但是该架构可以很容易地推广到更高维的卷积。 该架构适用于GSN和受监督的GSN。
最简单的卷积GSN仅包括输入层和卷积层。 假设输入层由Nv⇥Cv可见单元组成,其中Nv是输入的大小,Cv是输入通道的数量,卷积层包括K个特征映射,并且每个映射与大小为Nw⇥Cv滤波器相关联,其中 Nw是卷积窗口大小。 因此卷积层由(Nv-Nw + 1)个K个隐藏单元组成,每个隐藏单元与Nw⇥Cv可见单元连接。 过滤器权重在地图上共享。 每个地图还具有隐藏单位偏差b和可见单位偏差b0。 我们使用*来表示1D卷积,然后可见层和卷积层之间的向上和向下传递计算如下:
通过具有噪声激活函数σ~的独立随机变量z~P(Z)注入噪声。 在这项工作中,我们使用了与(Bengio et al。,2013a)中使用的相同的噪声激活函数:
通过仅考虑可微分部分或“直通估计器”来计算随机神经元的梯度,如(Bengio和Thibodeau-Laufer,2013)。
可以堆叠卷积层以创建深度表示。 为了允许学习高级表示,通常希望通过合并来减小上层中的特征映射的大小。 因此,我们应用平均池,在特征映射中汇集多个相邻要素并输出它们的平均值。 平均池的替代方法是max-pooling,它输出max而不是average值; 因为最大操作是不可逆的,我们可以进行向下传递,就像向上传递使用平均汇集一样。 汇集也有效地硬编码更高级别特征中的平移不变性。
在卷积GSN的计算图中,我们使用类似于(Bengio等人,2013a)中提出的深Boltzmann机器类网络架构的分层采样。 由于池化层包含与下层的冗余信息,因此将其与其上层卷积层一起视为卷积GSN层。 直观地,在分层采样中,池化层可以被认为仅提供卷积层输入的中间计算,并且存储卷积层激活并用于下一个采样步骤。
注意,其计算图的循环结构使得卷积GSN与前馈卷积网络显着不同。 在卷积GSN中,来自不同层和不同空间位置的表示可以通过双向连接彼此通信。 通过足够数量的向上和向下交替传递,来自输入/特征图的任何位置的信息可以通过多个不同的路径传播到任何其他位置(所需的传递次数直接取决于架构;如果顶层特征是 连接到所有输入位置,然后2N层轮就够了)。
虽然表示深度可以被认为是卷积GSN中的层数,但计算图的深度随着“采样”迭代次数的增加而增加,这也称为回溯次数(Bengio et al。,2013a), 所以计算图可以任意深度复制参数。
当使用这种卷积GSN架构进行蛋白质二级结构预测等监督训练时(图1),我们将考虑两种类型的输入通道,即特征通道(X)和标签通道(Y)。 在监督卷积GSN的计算图中(图1),我们只破坏和重建标签通道,并且特征通道不被破坏并作为输入的一部分提供以计算第一隐藏层的激活。
4. Experimental Results
4.1. Features and dataset
我们的目标是正确预测任何给定蛋白质序列的每个氨基酸的二级结构标签。 我们关注8状态二级结构预测,因为这是一个比3状态预测更具挑战性的问题,并揭示了更多的结构信息。 我们使用多任务学习同时预测二级结构和氨基酸溶剂可及性,因为学习使用相同网络预测其他结构属性允许共享特征表示并且因此可以改进特征学习。
作为位置特异性评分矩阵(PSSM)的进化信息被认为是预测来自先前研究的二级结构的最具信息性的特征(Jones,1999)。 为了产生PSSM,它们是n⇥b基质,其中n是蛋白质长度,b是氨基酸类型的数量,我们针对UniRef90数据库运行PSI-BLAST,包含阈值0.001和3次迭代。 我们使用来自PSI-PRED软件包的pfilt程序对数据库进行预过滤,以删除低信息量内容和卷曲线圈区域(Jones,1999)。 为了使用PSSM作为监督GSN模型的输入,我们通过sigmoid函数将PSSM分数转换为0-1范围。 我们还提供由n⇥b二元矩阵编码的氨基酸残基的原始蛋白质序列作为输入特征。 另外两个输入特征编码蛋白质序列的起始和终止位置。 虽然可以考虑其他功能以进一步提高性能,但我们将重点放在评估模型架构上。
我们使用了具有6128种蛋白质的大型非同源序列和结构数据集(在所有过滤后),并将其随机分成训练(5600),验证(256)和测试(272)组。 该数据集由PISCES Cull PDB服务器(Wang&Dunbrack,2003)生成,该服务器通常用于评估结构预测算法。 我们检索了一组解析的蛋白质结构,其分辨率优于2.5A,同时共享的同一性低于30%,这与(Wang et al。,2011)中使用的设置相同。 我们还去除了少于50或超过700个氨基酸或不连续链的蛋白质链。
通过DSSP程序从3D PDB结构推断出8态二级结构标记和溶剂可及性得分(Kabsch&Sander,1983)。 我们将溶剂可及性评分离散化为绝对溶剂可及性和相对溶剂可及性(Qi等,2012)。
得到的训练数据包括特征和标签,共有57个通道(PSSM 22个,序列22个,终端2个,二级结构标签8个,溶剂可及性标签2个),整个通道大小为700个.700个氨基酸通道 选择酸长度截止值以提供效率和覆盖率之间的良好平衡,因为大多数蛋白质链短于700AA。 短于700AA的蛋白质填充全零特征。
我们评估了含有272种蛋白质的测试数据集的二级结构预测的性能。 我们还对CB513数据集进行了单独评估,同时对Cull PDB数据集进行了进一步过滤以去除与CB513数据集具有> 25%同一性的序列。 通过Q8精确度或正确标记的氨基酸的比例来测量二级结构预测性能。 我们运行了5条平行马尔可夫链,每条链从模型中生成5000个连续的重建分布,并通过对来自每个马尔可夫链的最后500个重建分布求平均来评估性能。
4.2. Training setup
要向标签注入噪声,输入标签会被一半的值随机设置为零而损坏。 除了第一卷积GSN层之外,在激活之前和之后添加具有标准偏差2的高斯噪声。 通过从重建分布中取样获得每个yt。 我们将重构分布视为二级结构的多项分布和溶剂可及性的二项分布。 回溯号码设置为12,以确保整个网络的信息流。
当一个任意标签Y 0远离真实P(Y | X)的概率密度所在的位置启动时,有助于更好地学习重建分布,这对于监督预测任务非常重要,在培训一半的培训期间 我们通过与用于预测的相同的任意启动而不是其真实值来使我们初始化Y. 我们称这个技巧为“启动”。
Tanh激活函数用于除可见层之外的所有情况,其使用sigmoid激活函数进行重建。 所有图层都使用无条件权重,因此W和W'不相等。 我们通过约束每个权重向量的范数来规范网络。
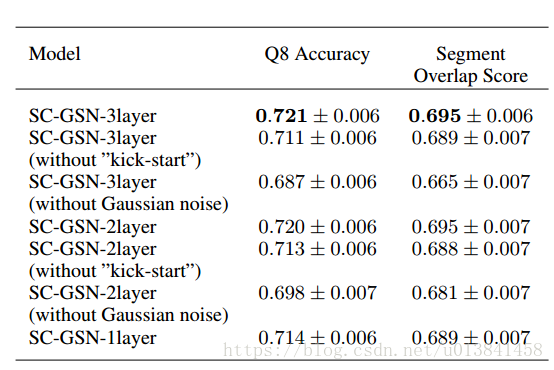
表1.不同体系结构的比较。
表2.公共基准数据集CB513的性能
通过反向传播重建误差来全局训练所有层的参数。 具有学习率0.25和动量0.5的小批量随机梯度下降用于训练模型参数。 所有比较模型都经过300个时期的训练。 该模型基于Theano和Pylearn2库实现,并在Tesla K20m GPU上进行了培训。
4.3. Performance
我们的Cull PDB测试集序列的Q8准确度达到了72.1±0.6%(表2)。 我们测试的最佳单一模型具有3层卷积结构。 我们将结构表示为{80 * conv5} - {pool5 - 80 * conv5} - {pool5-80 * conv4}。 'conv'或'pool'表示图层类型是卷积层还是池图层,图层类型后面的数字表示过滤器或池窗口的大小。 平均池用于此体系结构中的所有池层。 我们在所有卷积层中使用了80个通道。
图3显示了根据来自测试组蛋白的输入特征从训练模型生成的连续样品的实例。 我们观察到,当链条运行时,样本逐渐接近实验测量的二级结构,并且在后续的采样步骤中修正了一些不正确的预测。 后来的样本似乎也有更多空间一致的组织。
各个二级结构状态的预测性能如(表2)所示。 我们实现了高精度个体二级结构状态预测灵敏度,精确度和我们的Cull PDB测试集中所有蛋白质的氨基酸残基计算的频率如下所示。 状态我无法评估,因为它甚至不会出现在测试集中。
表3.各个二级结构状态的分类准确度。
对于四个主要状态H,E,L和T,已知对较不频繁的状态G和S的预测是困难的,因为它们具有有限数量的训练标准,导致显着不平衡的标签。 我们没有专门尝试解决这里的不平衡标签问题。 今后应该做出更多努力来更好地识别这些状态。 状态I非常罕见,因此我们甚至在测试集中没有遇到过,因此它可能会限制其预测的改进。
我们还在公共标准基准数据集CB513上进行了验证。 对于该验证,我们在过滤Cull PDB数据集之后训练模型以去除与CB513序列具有同源性的序列(即> 25%同一性)。 我们的模型达到了0.664的Q8精度,优于CNF / RaptorSS8(0.649)之前的最新结果(Wang et al。,2011)。
4.4. Analysis of architecture
为了发现我们架构成功的重要因素,我们尝试了具有不同层数的替代模型架构,并尝试去除“启动”训练或高斯噪声注入(表2,图4)。
对于具有不同层数的模型架构,我们从最初的最佳3层架构中删除了顶部1和2卷积GSN层。 2层模型优于1层模型,3层模型的性能略高于2层模型。
在训练期间不使用“启动”会大大降低采样速度,以便在采样期间获得最佳性能和预测精度。 这可能是因为“启动”模型在链从任意状态初始化时学习了更好的重建分布。

图3.来自训练模型的测试集蛋白的二级结构预测示例。 子面板中的每一行代表二级结构状态,从上到下分别为H,E,L,T,S,G,B和I. 有关二级结构状态的描述,请参阅表3。 每列对应于该蛋白质序列中的一个氨基酸残基,X轴显示该蛋白质中氨基酸残基的指数。 暗色与浅色表示预测的强烈对弱信度。
在计算上层时注入的噪声似乎是深度模型的最佳性能所必需的。 我们还注意到,在训练期间验证数据的较低重建误差并不总是对应于更好的预测性能。 对于没有高斯噪声注入到中间计算的3层模型,尽管验证重建误差低于具有噪声的模型,但来自具有噪声的模型的样本给出了更好的预测(0.721对0.687)。
5. Conclusions
为了将深度表示应用于蛋白质二级结构预测,我们实施了监督生成随机网络(GSN)并引入了卷积体系结构,以允许其学习全尺寸数据的分层表示。 虽然我们证明了其在二级结构预测方面的成功,但这种架构可能潜在地应用于其他蛋白质结构预测任务。 我们的实验表明,监督生成随机网络是一种有效的结构化预测算法,扩展了生成随机网络在捕获数据中复杂依赖性方面的成功。
我们应用的卷积和监督生成随机网络的组合非常适合于对本地信息敏感的低级结构化预测,同时被告知高级和远程特征。 因此,这种架构还可以应用于生物信息学之外的各种结构化预测问题,例如场景解析和图像分割。
对于蛋白质序列和结构建模的进一步发展,当前结构的一个限制是卷积结构是硬编码的,因此在某些情况下捕获蛋白质序列的空间组织可能不是最佳的,特别是对于由 远程互动。 为了更好地模拟蛋白质中的长程相互作用,自适应和动态架构根据输入自适应地改变连接,类似于展开递归自动编码器(Socher et al。,2011),可以进一步提高未来的表示质量。
Acknowledgments
我们要感谢Robert E. Schapire的有益讨论。 我们还承认普林斯顿大学的TIGRESS高性能计算机中心提供计算资源支持。 这项工作得到了美国国家科学基金会(NSF)职业奖(DBI-0546275),美国国立卫生研究院(NIH)(R01 GM071966,R01 HG005998和T32 HG003284)和国家普通医学研究所(NIGMS)卓越中心的支持( P50 GM071508)。O.G.T. 是加拿大高级研究院的高级研究员。
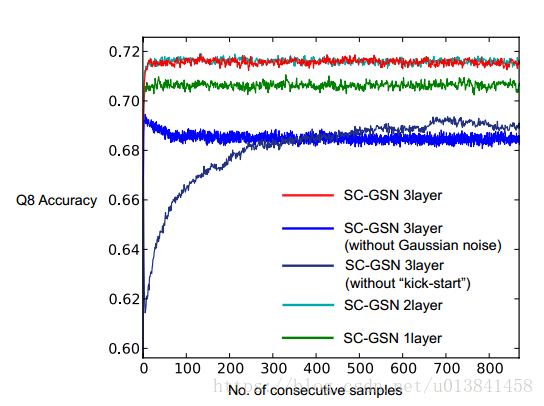
图4.来自具有不同架构的训练模型的各个连续样本的预测性能。 在每个模型的每个采样迭代中计算我们的Cull PDB测试集蛋白的Q8准确度。 显示了272种测试蛋白的平均性能。