简单介绍
sklearn中的LinearRegression类拟合了一个带有系数
的线性模型,使得数据集实际观测数据和预测数据之间的残差平方和最小:
其中向量
为coef_,定义
为intercept_
coef为系数、intercept为截距;
sklearn在coef和intercept后加上的下划线是为了表明这个是模型更新的属性。
数学解释
普通最小二乘法是解决线性回归问题最为普通的一种方法。
约定:
-
表示第
个样本点
-
表示第
个样本点的第
个特征
-
表示样本点
的第
个特征
维平面里有 个点 , 其中
假设函数 :
的向量形式:
其中:
点 的残差:
损失函数(残差平方和):
损失函数的向量形式:
最小二乘法推导过程:
由 得出:
使用矩阵求解回归系数的方法不需要对数据进行归一化处理, 而用梯度下降法则需要
当维度未上万, 用最小二乘法, 维度上万了, 用梯度下降法
对于普通最小二乘的系数估计问题,其依赖于模型各项的相互独立性。当各项是相关的,且设计矩阵 X 的各列近似线性相关,那么,设计矩阵会趋向于奇异矩阵,这会导致最小二乘估计对于随机误差非常敏感,产生很大的方差。例如,在没有实验设计的情况下收集到的数据,这种多重共线性(multicollinearity)的情况可能真的会出现。
sklearn中的最小二乘
1、首先从sklearn的linear_model(即线性模型)模块中引用LinearRegression类并实例化一个对象(该对象即为一个模型)。该类实现了普通最小二乘法。
from sklearn.linear_model import LinearRegression
model = LinearRegression()该方法使用 的奇异值分解来计算最小二乘解。如果 是一个 size 为 的矩阵,设 ,则该方法的复杂度为
LinearRegression类有4个参数:
| 参数 | 解释 | 默认值 | 说明 |
|---|---|---|---|
| fit_intercept | 是否为该模型计算截距 | True | 如果设为False,计算的时候将不会考虑截距(如当你的数据已经中心化了可以设为False,否则不是特殊情况不建议修改改参数)。 |
| normalize | 是否在训练之前对X做归一化处理 | False | 如果设置为False,训练之前可以使用sklearn.preprocessing.StandardScaler来做归一化处理;如果设置为True,X将减去平均值然后除以
范数 |
| copy_X | 是否保存一个X的副本 | True | 如果为True,保存一个副本,否则会被重写 |
| n_jobs | 计算机并行数 | 1 | 如果设为-1,所有cpu均会启用。只有当标签数大于1或者处理大型数据时这个选项才会起作用,一般情况不建议修改。 |
看到这么多参数讲真有点头疼,庆幸的是LinearRegression将这几个参数都设置了默认值,而且sklearn会尽量保证所设置的默认值最合理最高效(特别是在一些带超参数的模型如svm中,这点尤为重要)
2、然后使用model的fit方法来训练该模型
X = [[0, 0], [1, 1], [2, 2]] # features
y = [0, 1, 2] # labels
model.fit(X, y) # 训练fit方法有3个参数
| 参数 | 解释 | 默认值 | 说明 |
|---|---|---|---|
| X | 训练数据 | 无 | [n_samples, n_features]形状的numpy数组或稀疏矩阵。如果是别的形式(如列表),将被自动转成numpy数组 |
| y | 目标值 | 无 | [n_samples, n_targets]形状的numpy数组。如果是一维的,会自动转成[n_samples, 1]的形状 |
| sample_weight | 样本的权重 | None | [n_samples]形状的numpy数组,是否计算样本的权重,若为None,则所有样本权重相等 |
3、调用model的属性来查看训练结果
print(model.coef_)
print(model.intercept_)运行结果为
[0.5, 0.5]
2.220446049250313e-16我们看到截距项约为
,近乎为0。其实手动计算一下我们会发现截距项就是0,之所以会出现intercept_非常接近于0但不为0这种情况,是因为计算机计算误差,不能保证完全一样。当然这个误差可以忽略不计。
4、利用predict方法进行预测
X2 = [[3, 4], [5, 6]]
print(model.predict(X2))运行结果为
[3.5, 5.5]predict方法只有X这一个参数
| 参数 | 解释 | 默认值 | 说明 |
|---|---|---|---|
| X | 需要预测的样本 | 无 | [n_samples, n_features]形状的numpy数组 |
5、利用score方法计算
系数
关于 系数的数学解释,请查看这里
X_test = [[1.5, 2.5], [3.5, 4.5]]
y_test = [2.1, 3.9]
print(model.score(X_test, y_test))运行结果为
0.9876543209876545这个结果可以说是相当高了,说明数据拟合得非常好。
score方法有三个参数
| 参数 | 解释 | 默认值 | 说明 |
|---|---|---|---|
| X | 测试样本 | 无 | [n_samples, n_features] |
| y | X的真实值 | 无 | [n_samples]或[n_samples, n_outputs] |
| sample_weight | 样本权重 | None | [n_samples] |
sklearn的api设计得十分合理,这5个方法在别的大部分算法里也可以套用,这一点对新人还是很友好的。
举个栗子
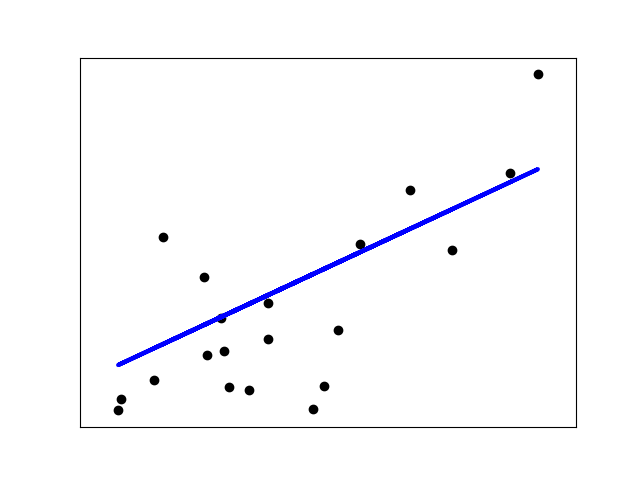
此示例使用糖尿病数据集的第一个特征。 在图中可以看到直线,显示线性回归如何尝试绘制直线,这将最好地最小化残差平方。 然后还计算了模型的系数,残差平方和 系数。
import matplotlib.pyplot as plt
import numpy as np
from sklearn import datasets, linear_model
from sklearn.metrics import mean_squared_error, r2_score
# 读取糖尿病数据
diabetes = datasets.load_diabetes()
# 仅使用一个特征
diabetes_X = diabetes.data[:, np.newaxis, 2]
# 将features分为训练集和测试集
diabetes_X_train = diabetes_X[:-20]
diabetes_X_test = diabetes_X[-20:]
# 将labels分为训练集和测试集
diabetes_y_train = diabetes.target[:-20]
diabetes_y_test = diabetes.target[-20:]
# 创建线性回归模型对象
regr = linear_model.LinearRegression()
# 训练
regr.fit(diabetes_X_train, diabetes_y_train)
# 预测
diabetes_y_pred = regr.predict(diabetes_X_test)
# 模型的系数
print('Coefficients: \n', regr.coef_)
# 均方误差
print("Mean squared error: %.2f"
% mean_squared_error(diabetes_y_test, diabetes_y_pred))
# R^2系数
print('Variance score: %.2f' % r2_score(diabetes_y_test, diabetes_y_pred))
# 绘图
plt.scatter(diabetes_X_test, diabetes_y_test, color='black')
plt.plot(diabetes_X_test, diabetes_y_pred, color='blue', linewidth=3)
plt.xticks(())
plt.yticks(())
plt.show()运行结果为
Coefficients:
[ 938.23786125]
Mean squared error: 2548.07
Variance score: 0.47