目录
旅行商问题(Traveling Salesman Problem, TSP)是计算机科学和运筹学中的经典问题。给定一系列城市和每对城市之间的距离,TSP要求找出访问每个城市一次并返回起点的最短路径。这是一个NP-hard问题,意味着没有已知的多项式时间算法可以求解所有实例。因此,研究者经常使用启发式方法来寻找近似解,其中遗传算法(Genetic Algorithm, GA)是一种广泛应用的方法。
TSP问题(Traveling Salesman Problem),是数学领域中著名问题之一。假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路径的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。路径的选择目标是要求所选的路径路程为所有路径中的最小值。
从图论的角度来看,TSP问题的输入是一个边带权的完全图,目标是找一个权值和最小的哈密顿回路。TSP问题可大致分为对称TSP问题和非对称TSP问题。所谓对称指的是在模型中,城市u 到城市 v的距离与城市u到城市 v的距离是一样的,其在图中的体现就是对称TSP问题的输入一般是无向图,而非对称TSP问题的输入往往是有向图。本文主要讨论的是对称TSP问题。
一、遗传算法概述
遗传算法是受自然选择和遗传学启发的搜索算法。它模拟了自然选择中的“适者生存”原则,通过迭代地改进一个解决方案的种群来寻找问题的最优解。GA的基本步骤包括初始化、选择、交叉(杂交)、变异和终止。
二、遗传算法在TSP中的应用
扫描二维码关注公众号,回复:
17326328 查看本文章


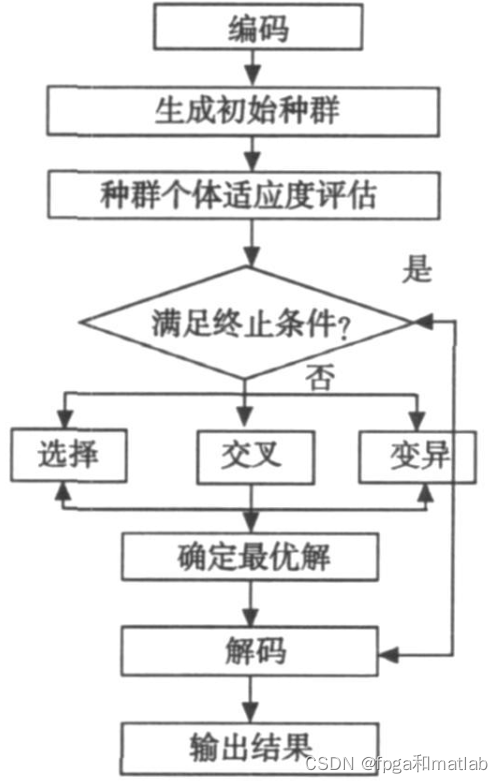
根据算法的流程图可知,遗传算法的基本步骤如下:
- 编码:在TSP中,每个解都可以表示为一个城市的排列。例如,对于5个城市A, B, C, D, E,路径ABCED是一个可能的解。这种编码方式称为排列编码。
- 初始化:随机生成一个初始种群,其中包含N个个体(解)。每个个体代表TSP问题的一个可能路径。
- 适应度函数:为了评估每个个体的“适应度”,我们使用路径长度的倒数作为适应度函数。路径越短,适应度越高。
(fitness = \frac{1}{total\ distance})
其中,总距离是路径中所有城市间距离的和。 - 选择:选择操作根据每个个体的适应度来选择父母,用于产生下一代。常用的选择方法有轮盘赌选择、锦标赛选择等。
- 交叉(杂交):交叉操作是GA中的关键步骤,它模拟了生物进化中的基因重组过程。在TSP中,常用的交叉方法有PMX (Partially Mapped Crossover)、OX (Order Crossover)和LOX (Large Order Crossover)等。以PMX为例,首先随机选择两个交叉点,然后交换两个父代个体在这两点之间的子串,并通过映射修复可能产生的重复城市。
- 变异:变异操作模拟了生物进化中的基因突变过程,有助于保持种群的多样性。在TSP中,常见的变异方法有交换变异(随机交换路径中的两个城市)、倒置变异(随机选择一段子路径并反转)等。
- 终止条件:当达到预设的最大迭代次数或满足某个停止准则(如适应度达到某个阈值)时,算法终止。
三、MATLAB程序
clc;
clear;
close all;
warning off;
addpath(genpath(pwd));
rng('default')
data = 20000*rand(100,2);
count = length(data);
population_size = 200;
iterations = 5000;
show_iterations = true;
show_iteration_every = 100;
text_x = max(data(:,1)) * 0.15;
text_y1 = max(data(:,2)) * 0.2;
text_y2 = max(data(:,2)) * 0.25;
text_y3 = max(data(:,2)) * 0.3;
%% Initianl population
initial_population = zeros(population_size, count);
for i = 1: population_size
initial_population(i,:) = randperm(count);
end
fitnesses(population_size) = 0;
population = initial_population;
%% Evolving loop
for round = 1 : iterations
%% Fitness calculation
for i = 1: population_size
fitnesses(i) = tsp_ga_fitness(data, population(i,:));
end
% disp(fitnesses);
%% Parents Selection
[selected, old_fittest] = tsp_ga_selection(population_size/2, population, fitnesses);
% disp(selected);
old_population = [];
for i = length(selected) : -1 : 1
old_population(i,:) = population(selected(i),:);
old_fitnesses(i) = fitnesses(selected(i));
end
% disp(old_fitnesses);
%% Recombination and Mutation
new_population = tsp_ga_recombination(old_population);
for i = 1 : size(new_population, 1)
new_population(i,:) = tsp_ga_mutation(new_population(i,:));
end
%% %% Fitness calculation
new_fitnesses = fitnesses(1:population_size/2);
for i = 1 : population_size/2
new_fitnesses(i) = tsp_ga_fitness(data, new_population(i,:));
end
% disp(new_fitnesses);
all_population = [old_population; new_population];
all_fitnesses = [old_fitnesses, new_fitnesses];
% disp(all_fitnesses);
%% Natural Selection
[next_generation, new_fittest] = tsp_ga_selection(population_size, all_population, all_fitnesses);
% disp(size(population));
% disp(size(old_population));
% disp(size(new_population));
% disp(size(all_population));
% disp(size(next_generation));
for i = 1 : population_size
population(i,:) = all_population(next_generation(i), :);
end
%% Display the generation
if show_iterations == true && rem(round, show_iteration_every)==1
clf;
tsp_ga_display(data, new_population(new_fittest,:), 'r');
tsp_ga_display(data, old_population(old_fittest,:), 'b');
text(text_x, text_y1, num2str(old_fitnesses(old_fittest)));
text(text_x, text_y2, num2str(new_fitnesses(new_fittest)));
text(text_x, text_y3, num2str(round));
drawnow;
end
end
% disp(all_fitnesses);
clf;
tsp_ga_display(data, old_population(old_fittest,:), 'b');
text(text_x, text_y1, num2str(old_fitnesses(old_fittest)));
text(text_x, text_y3, num2str(round));
disp(min(all_fitnesses));
四、仿真结论
