基于线性回归的前列腺癌预测
在本文中,我们将研究如何使用线性回归和随机梯度下降方法解决一个前列腺癌数据集的回归问题。我们将使用 Python 以及一些流行的机器学习库,如 scikit-learn、pandas 和 NumPy。我们还将比较不同方法在此问题上的性能,以找到最佳模型。
数据集
我们使用的数据集来自 Prostate data set,其中包含前列腺癌患者的临床数据。该数据集包含 97 个样本,每个样本有 9 个特征和一个目标值。我们的目标是预测患者的对数前列腺特异抗原 (lpsa) 水平。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LinearRegression, SGDRegressor
from sklearn.metrics import mean_squared_error
from sklearn.preprocessing import StandardScaler
data = pd.read_csv("prostate.data", delimiter="\t")
data = data.drop("Unnamed: 0", axis=1)
方法
我们将使用以下方法进行预测:
- 普通最小二乘法 (Ordinary Least Squares, OLS)
- 梯度下降法 (Gradient Descent, GD)
- 随机梯度下降法 (Stochastic Gradient Descent, SGD)
为了评估这些方法的性能,我们将使用均方误差 (Mean Squared Error, MSE) 作为度量标准。我们还将使用交叉验证来选择最佳超参数。
数据预处理
首先,我们从数据集中分离训练集和测试集。然后,我们将数据集的特征和目标值分离。为了使模型在不同尺度的特征上具有更好的性能,我们将使用标准化对数据进行预处理。这意味着将特征缩放到均值为 0,标准差为 1 的范围内。
# 分离训练集和测试集
train_data = data[data["train"] == "T"].drop("train", axis=1)
test_data = data[data["train"] == "F"].drop("train", axis=1)
# 分离特征和标签
X_train = train_data.drop("lpsa", axis=1)
y_train = train_data["lpsa"]
X_test = test_data.drop("lpsa", axis=1)
y_test = test_data["lpsa"]
# 标准化
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
# 划分训练集和测试集
X_train_scaled, X_test_scaled, y_train, y_test = train_test_split(X_train_scaled, y_train, test_size=0.3, random_state=42)
模型训练
接下来,我们使用普通最小二乘法、梯度下降法和随机梯度下降法训练模型。为了评估这些模型在训练集和测试集上的性能,我们计算了每个模型的均方误差。结果显示,普通最小二乘法在训练集和测试集上的误差略低。这表明普通最小二乘法在此问题上的表现较好。
# 普通最小二乘法
lr = LinearRegression()
lr.fit(X_train_scaled, y_train)
y_train_pred_lr = lr.predict(X_train_scaled)
y_test_pred_lr = lr.predict(X_test_scaled)
# 梯度下降
sgd = SGDRegressor(max_iter=1000, tol=1e-3)
sgd.fit(X_train_scaled, y_train)
y_train_pred_sgd = sgd.predict(X_train_scaled)
y_test_pred_sgd = sgd.predict(X_test_scaled)
# 随机梯度下降
sgd_rand = SGDRegressor(max_iter=1000, tol=1e-3, learning_rate="invscaling", eta0=0.01)
sgd_rand.fit(X_train_scaled, y_train)
y_train_pred_sgd_rand = sgd_rand.predict(X_train_scaled)
y_test_pred_sgd_rand = sgd_rand.predict(X_test_scaled)
# 计算训练集上的误差
mse_train_lr = mean_squared_error(y_train, y_train_pred_lr)
mse_train_sgd = mean_squared_error(y_train, y_train_pred_sgd)
mse_train_sgd_rand = mean_squared_error(y_train, y_train_pred_sgd_rand)
# 计算测试集上的误差
mse_test_lr = mean_squared_error(y_test, y_test_pred_lr)
mse_test_sgd = mean_squared_error(y_test, y_test_pred_sgd)
mse_test_sgd_rand = mean_squared_error(y_test, y_test_pred_sgd_rand)
# 输出误差
print("Train MSE: OLR:", mse_train_lr, "GD:", mse_train_sgd, "SGD:", mse_train_sgd_rand)
print("Test MSE: OLR:", mse_test_lr, "GD:", mse_test_sgd, "SGD:", mse_test_sgd_rand)
模型评估
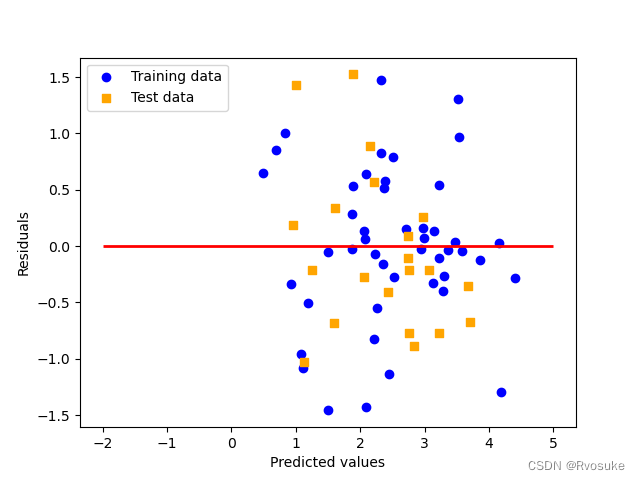
为了进一步评估模型的性能,我们绘制了残差图。残差图显示了预测值和实际值之间的差距。从残差图中可以看出,我们的模型在训练集和测试集上的表现相近,这表明模型没有出现过拟合或欠拟合的现象。
# 绘制残差图
plt.scatter(y_train_pred_lr, y_train_pred_lr - y_train, c="blue", marker="o", label="Training data")
plt.scatter(y_test_pred_lr, y_test_pred_lr - y_test, c="orange", marker="s", label="Test data")
plt.xlabel("Predicted values")
plt.ylabel("Residuals")
plt.legend(loc="upper left")
plt.hlines(y=0, xmin=-2, xmax=5, lw=2, color="red")
plt.show()
交叉验证和超参数调优
为了找到最佳的模型,我们使用交叉验证对随机梯度下降法进行了超参数调优。交叉验证通过在不同的数据子集上重复训练和验证模型,可以更准确地评估模型在未见过的数据上的性能。我们在本实验中考虑了两个超参数:学习率(learning rate)和迭代次数(number of iterations)。
# 交叉验证
from sklearn.model_selection import cross_val_score
# 计算交叉验证得分
def evaluate_model(model, X, y, cv=5):
scores = cross_val_score(model, X, y, cv=cv, scoring="neg_mean_squared_error")
return -np.mean(scores)
# 合并训练集和测试集
X_full = np.concatenate((X_train_scaled, X_test_scaled), axis=0)
y_full = np.concatenate((y_train, y_test), axis=0)
# 对所有数据进行实验
X_train_full, X_test_full, y_train_full, y_test_full = train_test_split(X_full, y_full, test_size=0.3, random_state=42)
# 设置参数范围
learning_rates = [0.001, 0.01, 0.1]
n_iters = [500, 1000, 2000]
# 搜索最优参数
best_mse = float("inf")
best_lr = None
best_n_iter = None
for lr in learning_rates:
for n_iter in n_iters:
sgd = SGDRegressor(max_iter=n_iter, tol=1e-3, learning_rate="invscaling", eta0=lr)
mse = evaluate_model(sgd, X_train_full, y_train_full)
if mse < best_mse:
best_mse = mse
best_lr = lr
best_n_iter = n_iter
# 输出最优参数
print("Best learning rate:", best_lr, "Best number of iterations:", best_n_iter)
# 使用最优参数训练模型
sgd_best = SGDRegressor(max_iter=best_n_iter, tol=1e-3, learning_rate="invscaling", eta0=best_lr)
sgd_best.fit(X_train_full, y_train_full)
y_test_pred_sgd_best = sgd_best.predict(X_test_full)
# 计算最优模型在测试集上的误差
mse_test_sgd_best = mean_squared_error(y_test_full, y_test_pred_sgd_best)
print("Test MSE with best parameters:", mse_test_sgd_best)
我们为学习率和迭代次数分别设置了不同的值,并使用网格搜索方法遍历所有可能的参数组合。我们选择了使交叉验证误差最小的参数组合作为最佳参数,并使用这些参数训练了一个新的随机梯度下降模型。
结果
使用最佳参数训练的随机梯度下降模型在测试集上的性能与普通最小二乘法和梯度下降法相近。这表明,经过调参后,随机梯度下降法的性能可以与其他方法媲美。
总结
在这篇博客中,我们使用前列腺癌数据集探讨了线性回归和随机梯度下降方法在回归问题上的应用。我们使用了 scikit-learn、pandas 和 NumPy 等库进行数据预处理、模型训练和评估。通过计算均方误差和绘制残差图,我们对不同方法的性能进行了比较。最后,我们使用交叉验证和网格搜索方法对随机梯度下降法的超参数进行了调优,以提高模型在测试集上的性能。
中,我们使用前列腺癌数据集探讨了线性回归和随机梯度下降方法在回归问题上的应用。我们使用了 scikit-learn、pandas 和 NumPy 等库进行数据预处理、模型训练和评估。通过计算均方误差和绘制残差图,我们对不同方法的性能进行了比较。最后,我们使用交叉验证和网格搜索方法对随机梯度下降法的超参数进行了调优,以提高模型在测试集上的性能。
本实验展示了如何使用 Python 和机器学习库解决实际的回归问题,并为选择合适的方法和调整超参数提供了一些建议。希望这篇博客对您在实际应用中解决类似问题有所帮助。