名词解释
MRF:
磁共振指纹打印技术( MR Fingerprinting)是近几年发展起来的最新磁共振技术,以一种全新的方法对数据进行采集、后处理和实现可视化。
MRF使用一种伪随机采集方法,取代了过去为获得个体感兴趣的参数特征而使用重复系列数据的采集方法,并使之具有唯一的信号演变或“指纹”,即同时获得所研究的不同物质特性的功能。
数据采集后的处理过程涉及一个模式识别算法,将“指纹”与预测信号演变的预定义资料库匹配。然后这些“指纹”被转换成定量的兴趣区的磁性参数图。
MRF提供高准确度的T1、T2、质子密度及扩散定量图。这为在影像生物标志物方面应用磁共振成像提供了一种全新方法,可以应用于神经、肿瘤、肌骨、心血管、胸部、代谢。
与传统的定性MR扫描相比,MRF在单次成像时间内可以获得完整的定量结果,且没有在许多其他快速检查方法中存在的对测量误差高度敏感的特点。
最重要的是,只要给予足够的扫描时间,MRF有潜力同时定量检测很多MR参数,而当前的MR技术一次只能检测有限的参数。
因而,MRF打开了计算机辅助多参数MR分析的大门,类似于基因组或蛋白组分析,可检测从大量MR参数中同时获得的重要且复杂的数据变化。新的研究表明,有可能通过MRF获得扩散数据和灌注信息。MRF可提供高度可重复性的多参数图像,在多中心/多机型研究中有很大的潜能。
T1 &T2
MRI(磁共振成像是利用原子核在磁场内共振产生的信号经重建成像的成像技术)里面的T1和T2加权成像[2]:
-
T1加权成像(T1WI)----突出组织T1弛豫(纵向弛豫)差别
-
T2加权成像(T2WI)----突出组织T2弛豫(横向弛豫)差别。
在任何序列图像上,信号采集时刻横向的磁化矢量越大,MR信号越强。
T1加权像高信号的产生机制
一般认为,T1加权像上的高信号多由于出血或脂肪组织引起。但近年来的研究表明,T1加权高信号尚可见于多种颅内病变中,包括肿瘤、脑血管病、代谢性疾病以及某些正常的生理状态下。
在射频脉冲的激发下,人体组织内氢质子吸收能量处于激发状态。在弛豫过程中,氢质子将其吸收的能量释放到周围环境中,若质子及所处晶格中的质子也以与Larmor频率相似的频率进动,那么氢质子的能量释放就较快,组织的T1弛豫时间越短,T1加权像其信号强度就越高。T1弛豫时间缩短者有3种情况:其一为结合水效应;其二为顺磁性物质;其三为脂类分子。
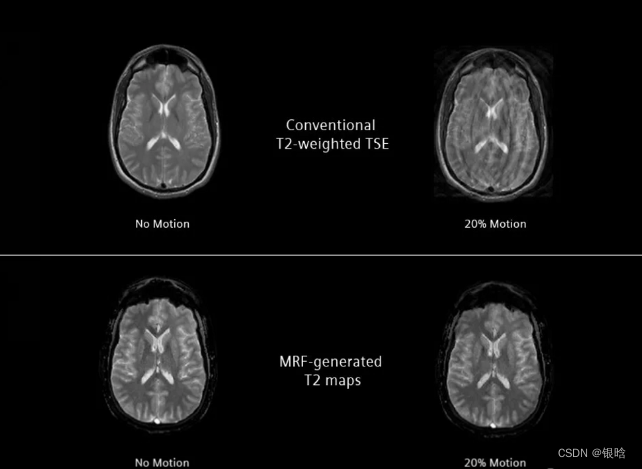
Quantitative prostate imaging——前列腺定量成像
挑战&可做的点
前列腺癌面临的挑战和问题包括:
(1)由于无法预测特定癌症的侵袭性和风险而导致的过度诊断和过度治疗;
(2)活检取样不足导致的误诊和看似低风险前列腺癌患者的疾病进展。
可做的点:
1)而在前列腺癌中,AI可在肿瘤分子诊断的基础上,加强临床医生对前列腺癌检测、定位、确定分期及监测的能力;
(2)对于前列腺癌的诊断可使用不同模式的卷积神经网络。
①例如一些研究人员使用卷积神经网络,通过自动开窗机制对MRI的结果进行分类,克服了MRI图像的高动态范围和归一化。还有一些研究者使用不同的mpMRI图像组合,将每个模态作为RGB图像的二维通道进行训练。
(3)而三维卷积神经网络可以用来设计特殊MRI参数,比如表观扩散系数、高b值、体积传输常数(Ktrans)模态。
论文1(2020):
影像组学特征 + 病理学特征 --> 机器学习模型 / 统计学分析相关性
- 探索MR图像和数字化病理图像在癌症预测中的相关性
方法:病理图像中分割组织,利用病理组学提取特征;MRI中分割组织,利用影像组学提取特征;相关性分析 - 结合MRI图像和病理图像中,用影像组学的方法提取特征,用机器学习方法对局部晚期直肠癌进行分期预测
- 《Identifying Cross-Scale Associations between Radiomic and Pathomic Signatures of Non-Small Cell Lung Cancer Subtypes: Preliminary Results》
- 探索CT图像和相应的数字化病理图像之间的跨尺度关联,使用多分辨率细胞密度量化以识别判别病理模式
方法:病理组学+影像组学+统计分析
- 探索CT图像和相应的数字化病理图像之间的跨尺度关联,使用多分辨率细胞密度量化以识别判别病理模式
- 将MRI图像的影像组学与从深度学习模型获得的病理学特征以及临床因素相结合,以构建鼻咽癌的多尺度预后列线图
论文2:
2021 << European Radiology >> : T1 and T2 MR fingerprinting measurements of prostate cancer and prostatitis correlate with deep learning–derived estimates of epithelium, lumen, and stromal composition on corresponding whole mount histopathology
方法流程
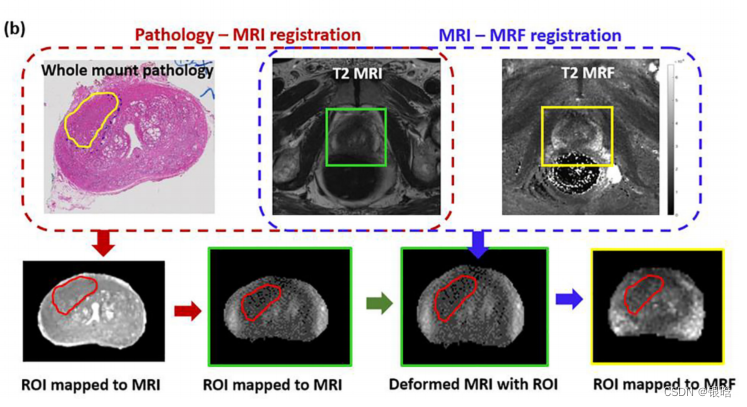
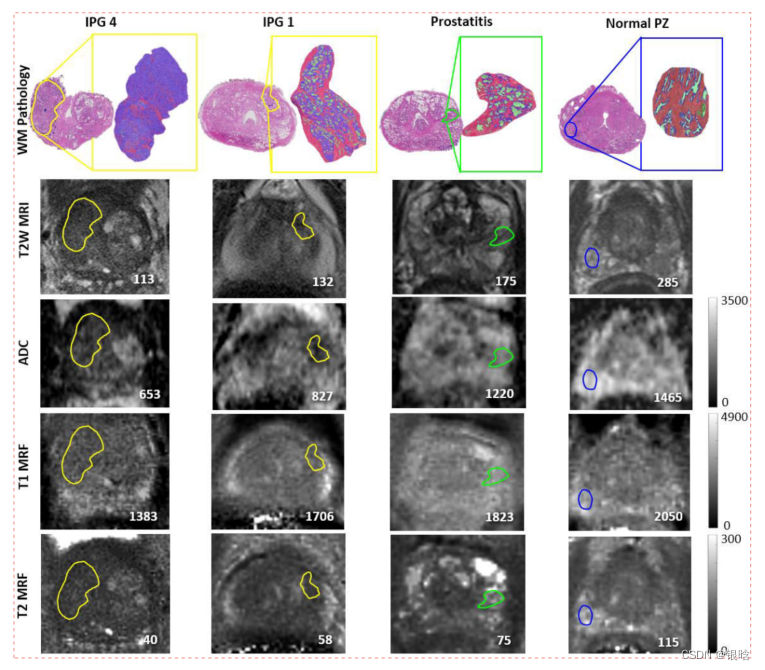
14例PCa患者,他们在根治性前列腺切除术前接受了3T多参数MRI和T1、T2 MRF图。人工建立整个标本与MRI、MRF之间的对应关系。
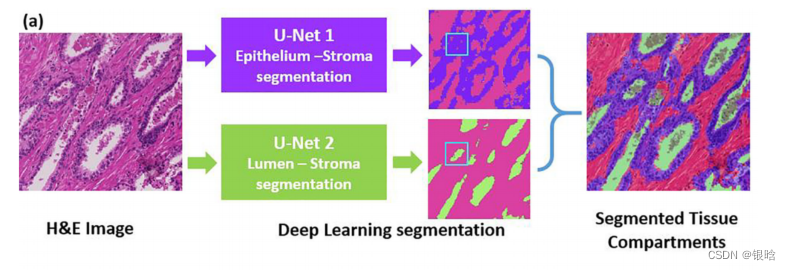
文章用两种U-net深度学习模型对前列腺炎、前列腺癌和病理上的正常外周区(PZ)感兴趣区域(ROIs)进行图像分割,以获取上皮、管腔和间质的组织间比率(TCRs)。
- ROIs映射到T2加权MRI图像上面 ;
- T1和T2的MRF图像表现扩散系数(ADC):用来区分癌变和正常的
- TCRs之间的相关性通过皮尔逊相关性分析,通过方差分析来分析统计学的差异
前列腺癌和前列腺炎的病理ROI映射到MRI和MRF上
出发点:
-
MRF的形态学基础对于理解驱动这一过程的病理特性是非常重要的
-
之前的研究:ADC来自WI(扩散加权成像),T2加权,DCE(图像动态对比度加强),上皮、管腔和间质都被研究了,目前还没有研究通过配对组织病理学来评估T1和T2弛豫时间之间的关系
-
定量MRF可能有助于区分前列腺炎和前列腺癌, 但是有必要评估这些发现的可解释性,特别是在相应的组织病理学特征方面。
本文是基于MRF的T1和T2弛豫测量的组织形态基础 ,探讨前列腺癌和前列腺炎定量T1和T2图与组织病理学之间的差异相关性。
目标是建立一个组织形态测量基础来利用MRF松弛测量来表征前列腺癌和前列腺炎。
我们观察到病理上的组织隔间与T1、T2 MRF图之间的显著相关性。
IPG对上皮细胞密度的增加反映在较低的T2 MRF测量值中,并且与管腔密度呈反比关系。
通过T1 MRF测量,基质含量随着IPG的升高而降低。
通过T2 MRF测量显示前列腺癌和前列腺炎之间存在差异,这些趋势在低级别前列腺癌和前列腺炎中也有观察到。
结果
- T1、T2的MRF和上皮细胞之间是负相关
- T1与前列腺癌是正相关,T1与前列腺间质是负相关
- T2和前列腺管腔是正相关,1与前列腺炎是正相关
- 前列腺炎和前列腺癌在TZ(过渡区域)和PZ(正常周边区域)存在显著性差异
综上所述:
- T1和T2的MRF之间有显著性差异(T1是过渡区域,T2是正常周边区域),
- T2 MRF在外周区相关性较强,而T1 MRF在过渡区与前列腺癌的组织病理学相关性较强
-
- 不同ipg、前列腺炎和正常外周区(NPZ)的平均T1和T2 MRF测量值有显著性差异(p < 0.001)
- 随着ISUP预后组(IPG)的增加,癌变感兴趣区域内的平均T2 MRF测量值和ADC下降。
- T1和T2的MRF图像与之对应的组织病理学上的差异也被观察到了;
如何利用病理知识:
手术前,前列腺标本取5毫米厚切片,每个切片用标准的数字显微镜进行数字化,分辨率是x20。然后病理学家在数字化后的病理切片上做标记,包括前列腺癌、前列腺炎和正常外周区(NPZ)。
使用到的深度学习模型:U-Net
组织间室分割包括识别WM上ROIs内的上皮、管腔和间质区域。采用了两种不同的基于u -net的深度学习模型。
- 一种是上皮-间质分割模型,另一种是管腔分割模型。
两个模型分开训练。因为第一个模型使用的图像分辨率(x20)高于第二个模型(x5),结合两个不同模型的结果对三个组织间室进行了分割。
每个图像的右下角提供了ROI内的平均值。
结论:在相应的组织病理学上,我们观察到MRF (T1在转移区中,T2在外围区中)与组织间的显著相关性
论文3
Nature新子刊《Nature Machine Intelligence》发表的一篇“High-accuracy prostate cancer pathology using deep learning”的文章
亮点:
- 通过基于DL(深度学习)的精确模型分析前列腺癌患者的组织学图像,达到并超越人类水平精度的潜力,
即从原来的切片级别 --> ptach 级别。 - 通过基于DL的模型可将Gleason评分集成到数字病理学流程中,从而开发前列腺腺癌的Gleason分级开发了一种新的生物学算法。
检测模型
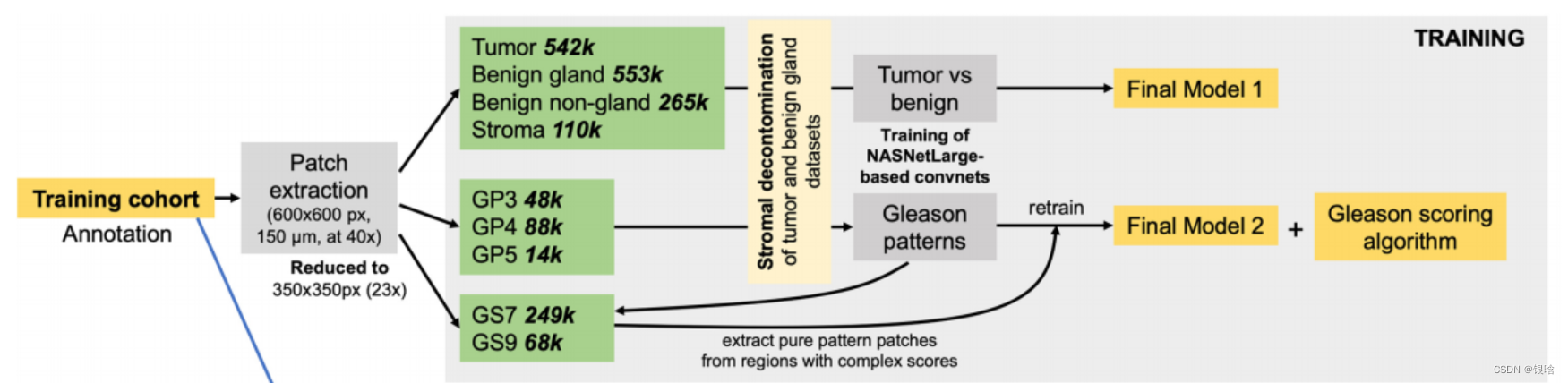
首先,作者通过一个训练队列和两个验证队列对肿瘤组织和良性组织进行检测分类,训练队列由389个WSI组成,两个验证队列由苏木精和曙红染色的TMAs(组织微阵列)组成。
结果发现NASNetLarge卷积网络体系结构在肿瘤组织检测中达到很高的准确性。初始卷积在训练队列中整体分类精度为97.8%(图1a),验证队列1和2的精度96.2%(图1b)和96.7%(图1c)。
接下来作者通过基于DL的策略增强了初始卷积精度,其中P8策略将整体准确性提高了0.7%,而ENS策略也可提高总体准确性,其主要是通过提高肿瘤组织分类的准确性(图1c)。
这几种策略提高总体准确性的原因是可提供肿瘤斑块属于某个类别的概率的定量信息
在大多数患有癌的斑块中,成为肿瘤斑块的概率为0.9-1.0,但是如果斑块中还存在良性组织,癌灶很小或分化良好或存在假阳性结果,则这种可能性可能会更低。
- 该系统将单个斑块进行扩展分析的必要触发条件为肿瘤概率达到0.2-0.8,并通过后续P8和ENS策略提高其精准度,从而使得肿瘤组织中的假阳性结果显著改善。
深度卷积网络不是一成不变的,可以从新信息中学习。作者将来自验证队列1的标记数据集整合到训练数据集中,并对卷积进行了一个时期的后训练。观察到在验证队列2上测试的总体准确性有显着提高,这在很大程度上是由于良性肿瘤中斑块的假阳性分类减少所致。
加强策略
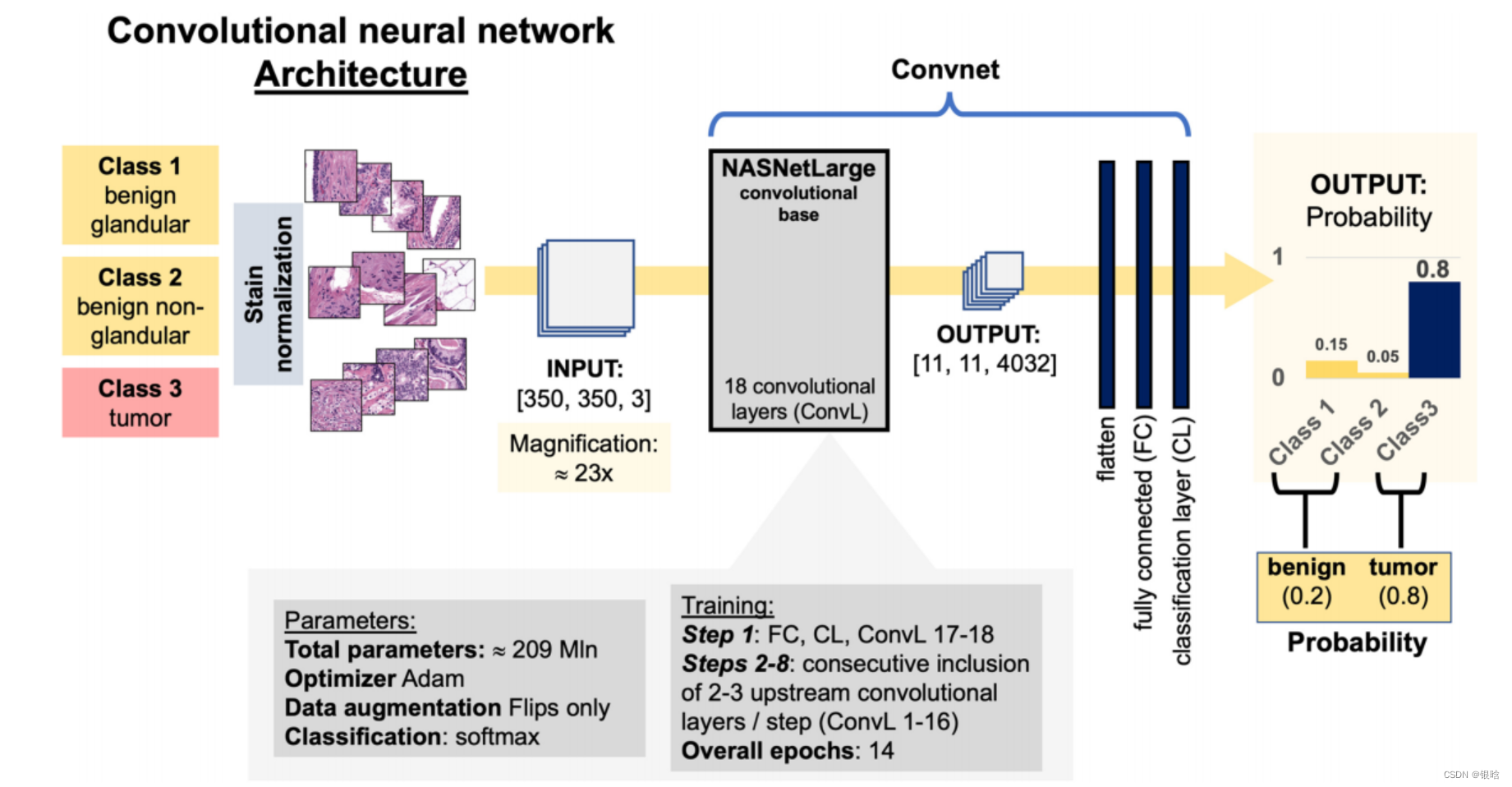
整个流程:先用卷积网络进行分类,网络输出结果后,如果判断为恶性肿瘤的概率是0.2 - 0.8 ,就使用再判断的策略,P8和ENS,完成判断
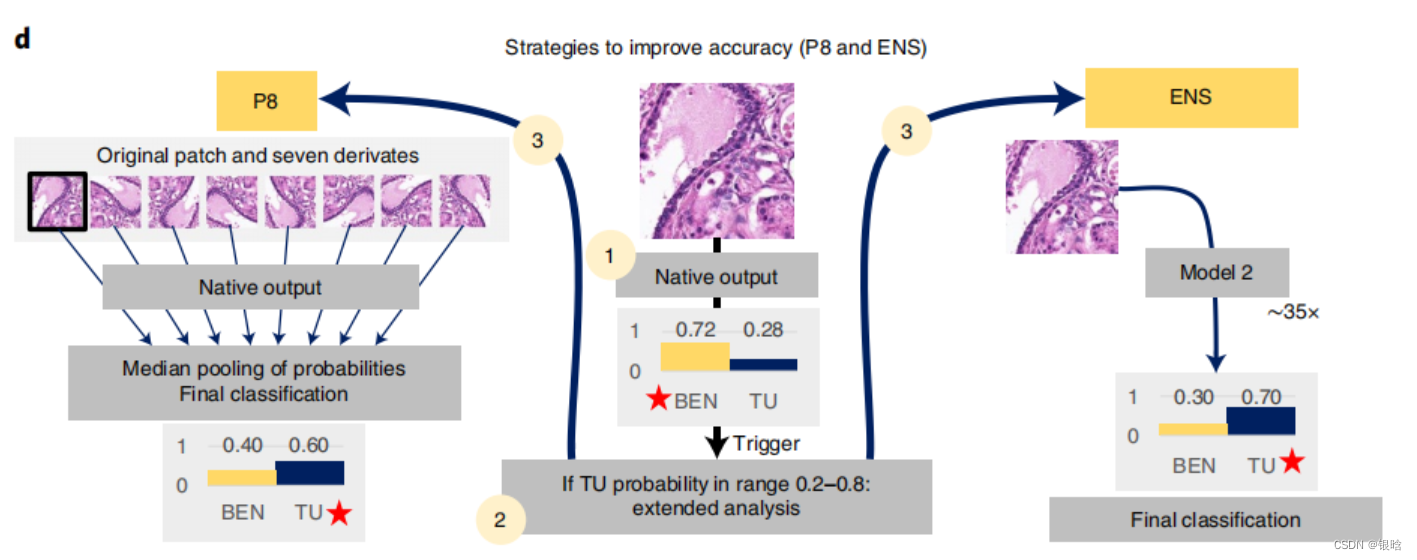
P8: analysis and pooling of probabilities from eight derivates of initial patch 我的理解是分析8个子图的是恶性肿瘤的概率,投票得到最后的结果
ENS: using ensemble of two models, the second ENS convnet was trained using similar principles but at a magnification of approximately ×35 (patch size 500 px). 看另外一个网络的结果,另外一个网络的数据的patch size大小不一样
接下来作者前列腺切片WSI进行分析,发现通过P8和ENS(ensemble network )两种策略,肿瘤组织中的假阳性结果显著改善,组织平方,肿瘤平方和肿瘤体积等数据组织分类结果中生成,生成完整的病例报告(图2)。
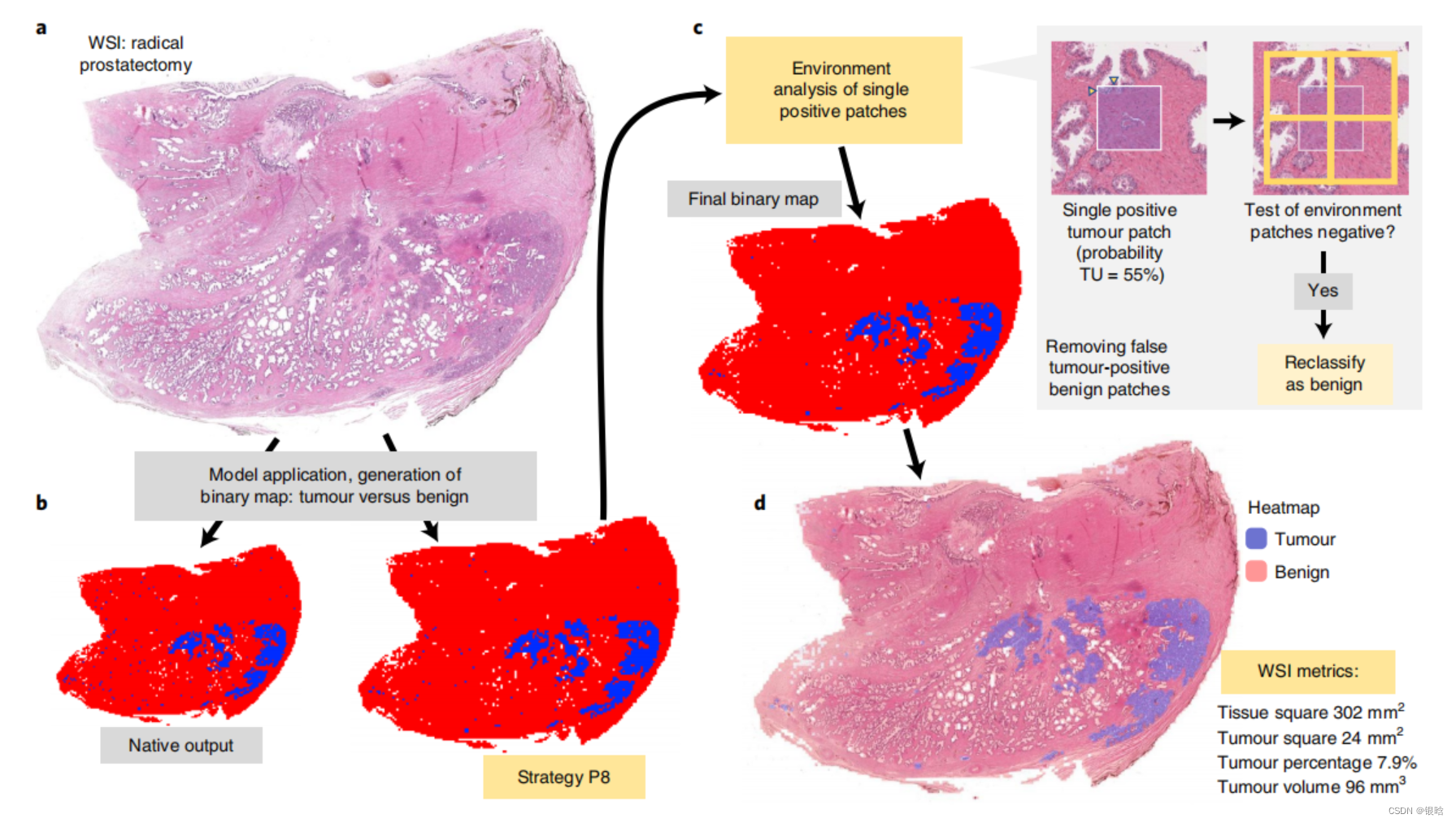
- 这个红蓝图,是个二值化处理后的图像 模型输出后 P8策略改善,然后ptach level的环境分析,即离群点检测
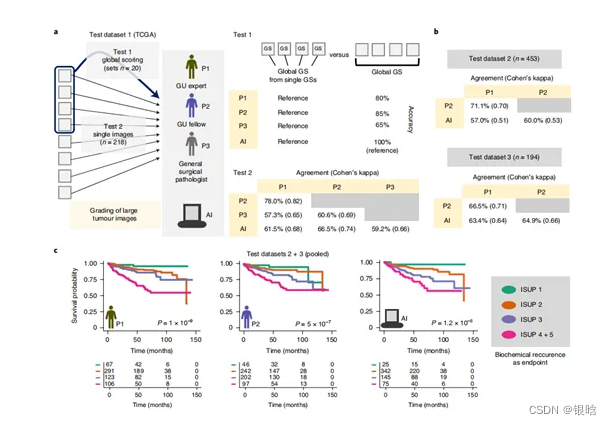
接下来作者考察三位前列腺癌专家和基于Dl的模型对前列腺癌组织进行分级准确性测试(图a)。使用两个测试数据集,分别为癌症基因组图谱(TCGA)队列的218张大肿瘤图像的总体Gleason评分(测试数据集1),对单个肿瘤图像进行Gleason分级,比较总体和单个图像的得分,从而获得整体得分的准确性(测试数据集2)。
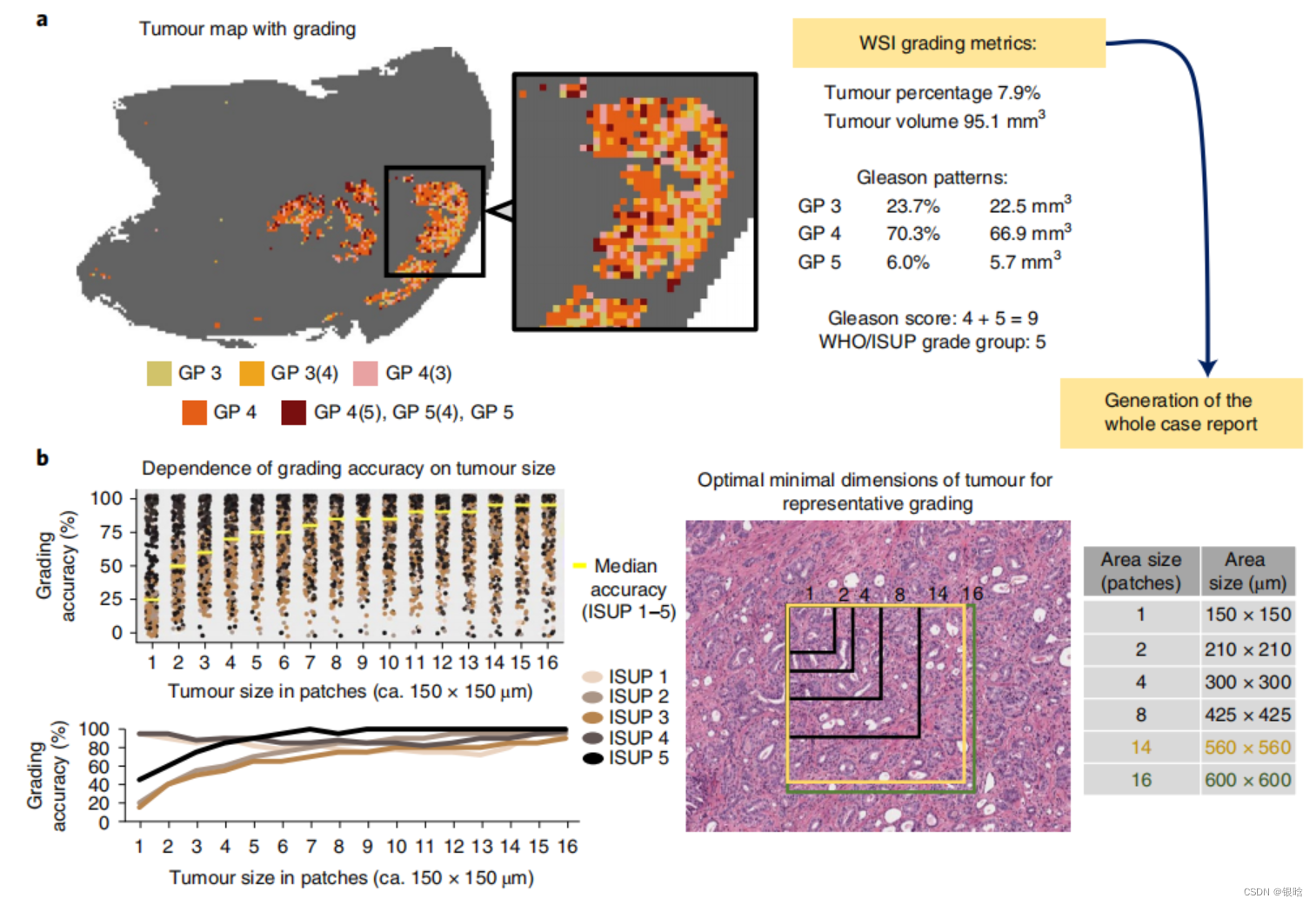
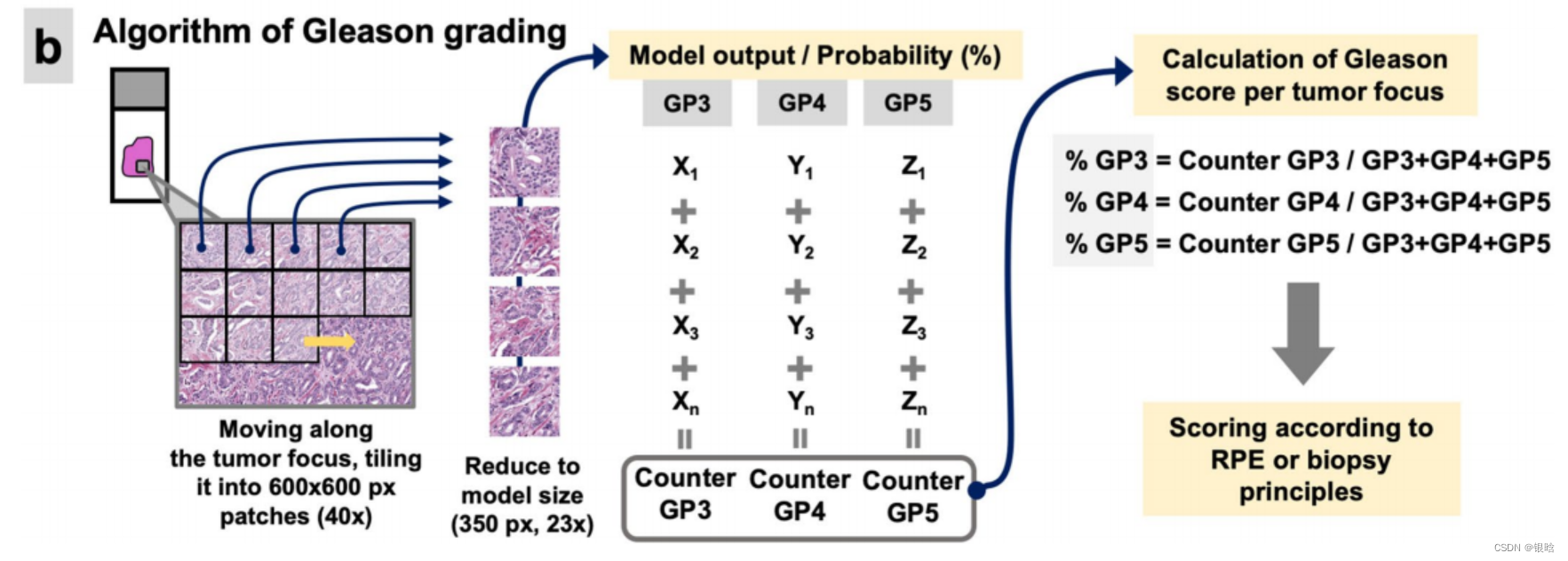
格里森分数评级模型:
评分依据:来自于卷积网络输出的概率(每种类别对应有概率)
在肿瘤区域内,检测最小最佳的肿瘤(patch level)分析肿瘤的体积,把原来的1 个 patch 随机抽取 16个更小的图。
- 综上:两个模型,一个分类,一个打分
结果发现,人类病理学家给至少15–20%的病例提供了不精准的总体Gleason评分,而使用人工智能(AI)根本没有发生这种观察者差异。此外,基于DL分析的单个肿瘤图像进行Gleason分级,其与总体一致性可达到60%并且高于病理学家所分析的数据(图3b)。
但在某些情况下,如定量变量Gleason分级,人类评分与基于AI评分之间的差异存在分歧只是微不足道的。例如对预后分级中,病理学家和基于DL的算法都表现出较高的预后评分(图3c).
- 关键点:可通过基于DL的模型可将Gleason评分集成到数字病理学流程中(图4a),
从而获取前列腺癌患者切片中所有与评分相关的指标,而人类无法准确计算这些指标,但是其也受到肿瘤大小的影响。
前列腺肿瘤表现出高水平的形态异质性,这可能会影响Gleason分级,作者通过将图像中不同斑块获取的Gleason评分与整体图像评分进行比较(图4b),发现肿瘤大小在560×560μm时可达到95%的准确度,表明在基于DL的模型分析中依旧存在肿瘤大小影响Gleason评分功能。
网络结构:
格里森打分算法
前列腺癌(PCA)的结构不仅仅是一个具有Gleason特征(GP)3、4和5的腺体的粗略混合。
在三维空间中,癌是一种树,其中不存在单一的格里森特征,它是一个连续体
-
我们的格里森分级算法是基于对格里森特征之上的一个连续体的理解。在处理过程中,一个肿瘤区域被切割成patch,然后由convnet分别进行分析
-
对每个patch的不同类别(GP3、GP4、GP5)的概率进行统计,并对整个肿瘤区域进行总结,而不是用于将每个patch表示为一个离散的GP
作者表示尽管数据集很大且质量很高,但仍需要通过结合新的训练数据集和预期的多机构验证来进一步不断发展。进一步的研究应包括对整个前列腺切片的分析,包括所有肿瘤部位,因为基于TMAs的材料可能不是最佳的。目前,完全数字化队列的可用性是一个瓶颈,有望在不久的将来克服。
数据集及代码
论文4
Nature: Biologically informed deep neural network for prostate cancer discovery
- 这篇文章构建了名为P-NET、以生物信息学为基础的深度机器学习模型。通过这一模型,可以为处于治疗耐受状态的前列腺癌病人预测新的治疗靶标。
重点:可解释性机器学习 -》解释神经网络输出的信息 ,然后去验证- 权衡准确率和可解释性
对比机器学习
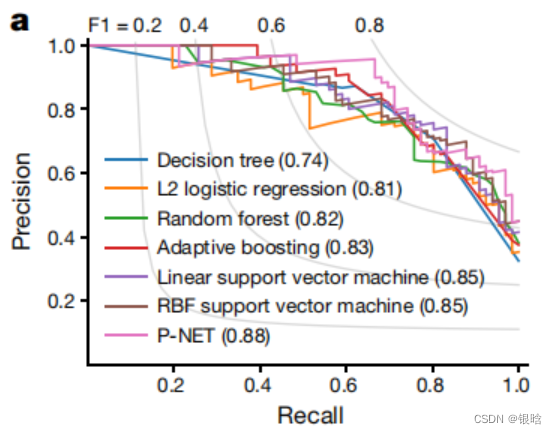
- 看出来,5层的多层感知机模型在效果上其实不占很大优势,原因是神经元太少,层数太少了
- 所以说,acc 和 interpretability 二者权衡!!!
- 另外,作者用了神经网络的可解释性的工具及思想,emmm…
网络结构
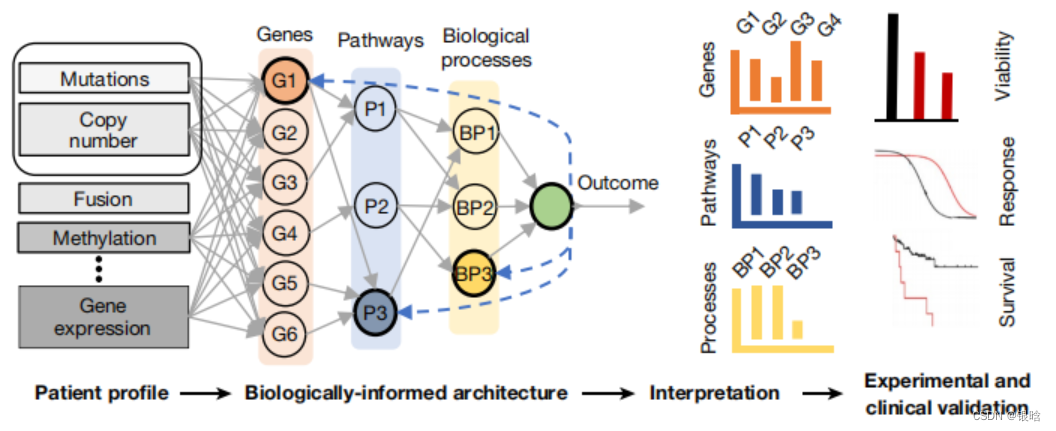
- 这篇文章预测的是前列腺癌的基因组数据,基因组的数据是一个图数据
具体来说,作者基于生物神经网络的层级概念,整合了3007组生物网络图谱,构建了一个深度学习预测模型,输入前列腺癌病人的基因组,输入基因组进行计算
第一层可以输入一系列基因簇,这些基因簇之间可以通过带有权重的线相连接;
第二层则是相关的生物学通路;
接下来是整合这些通路所预测的可能生物现象及结论,依此类推,最终可以预测这一病人的病情。
- 所以作者在这个5层全连接网络里面,逐层逐节点的进行解释,每个节点是干什么的
- 我觉得是正则化的思想,每一个正则化的力度很大,所以表现出不同的功能
- 对节点的度施加了强约束,例如:节点的入度和出度
数据来源:
训练集:作者采集了1013位前列腺癌病人的资料,其中80%用于模型训练,10%用于模型校正,剩余10%用于检测这一模型的正确率。从结果来看,在输入相同体量的样本情况下,作者所构建的P-NET模型准确率要优于已知的深度机器学习模型。
测试集:作者招募了两组前列腺癌病人,一组是处于原发状态,另一组癌症病灶已经发生转移。
结果,P-NET模型可以准确预测73%的原发状态和80%的已转移状态。
作者猜测,之所以有几例处于原发状态的病人被P-NET模型归为已转移状态,很可能是因为这几例病人病情较为严重,P-NET评分较高所致。从另
- 这一模型在预测病人所处状态之余,还可以通过P-NET评分来显示病情严重程度。
可解释性:
再下来,为了研究不同的基因、信号通路和生物学网络之间的相互作用以及对模型预测结果的影响,作者用示意图的形式显示了这一模型的输出的整体结构(如下图所示)。
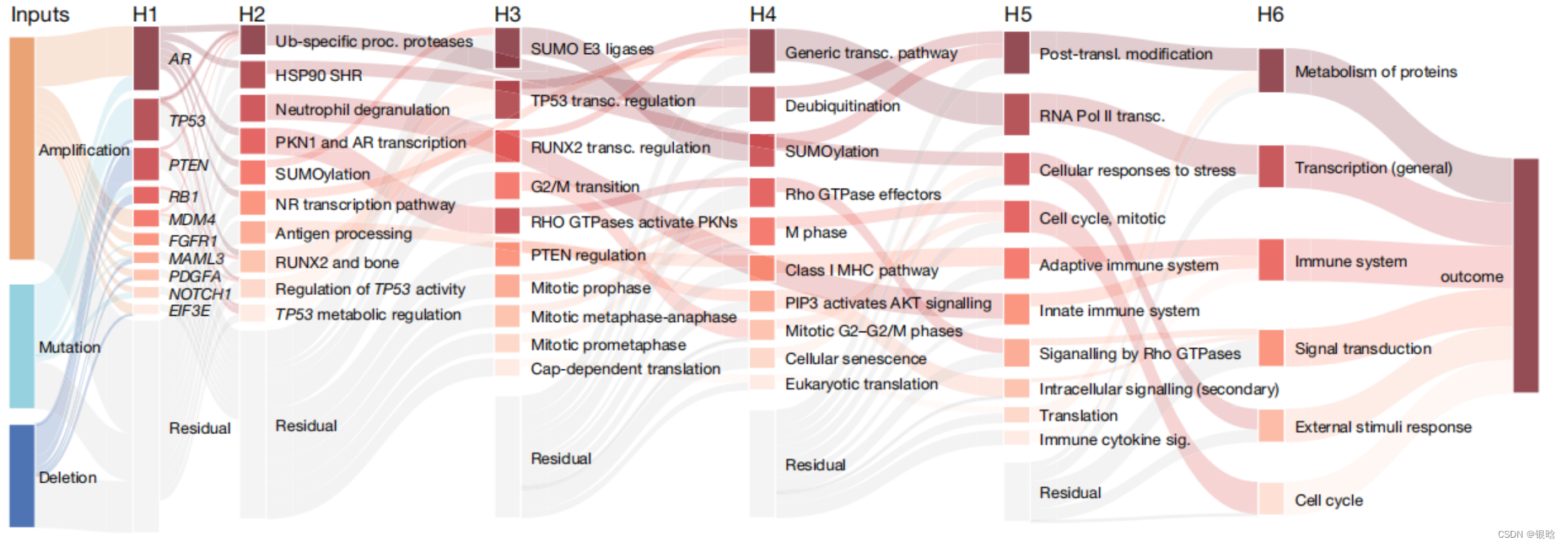
- 在前列腺癌病人众多的基因组层面变化中,基因拷贝数的变化对病情影响要高于基因突变本身。
另外,在P-NET所涵盖的3007组信号通路中,多条细胞周期相关通路与已转移状态相关,而泛素化和SUMO化信号通路则与多种肿瘤抑制因子相关,包括AR,这些通路异常很可能会导致前列腺癌发病。例如,骨源转录因子RUNX2可以调控细胞周期,且与前列腺癌转移相关。
再下来作者通过P-NET模型预测出一系列与癌症进程相关的基因。重点关注的基因包括AR, PTEN, RB1 和TP53 ,都是已知的前列腺癌驱动基因,之前也被报道与肿瘤转移相关。另外,有一些基因,比如MDM4, FGFR1, NOTCH1 和PDGFA ,与预测结果的准确性相关。
最后,作者深入分析P-NET模型输出的各级信息,发现TP53相关通路与摘除性器官治疗效果有限的前列腺癌相关。有报道指出,TP53以及其通路重要基因MDM2与前列腺癌病程相关,作者同样发现,MDM4这一基因很可能也有类似的作用。而在此前,已知MDM4可以通过与TP53的转录区域相结合来抑制TP53的表达,但与前列腺癌疾病进程之间的关系仍旧不够清楚。
为了进一步研究MDM4这一基因的具体作用,作者通过CRISPR–Cas9 的方法,在多种前列腺癌细胞系中特异性敲除了MDM4。结果显示,与对照组细胞相比,特异敲除MDM4的细胞增殖水平明显降低。另外,含有野生型TP53的前列腺癌细胞系,对MDM4抑制剂RO-5963与TP53突变的细胞系相比更为敏感。
总结
综上所述:
- 作者给出了通过神经网络模式,基于深度机器学习建立模型,从而解决生物学问题的新方法。
- 作者证实,通过这一模型,可以由前列腺癌病人的基因测序结果预测病人所属的疾病状态。
- 通过模型的输出信息,作者还预测了一些新的靶标,比如MDM4,很可能与前列腺癌病程相关,并通过体外敲除的方法证实,MDM4缺失可以抑制前列腺癌细胞的增殖。更进一步说,这一模型以及相关研究方法,也有希望推广至多种癌症类型。
P-Net设计:
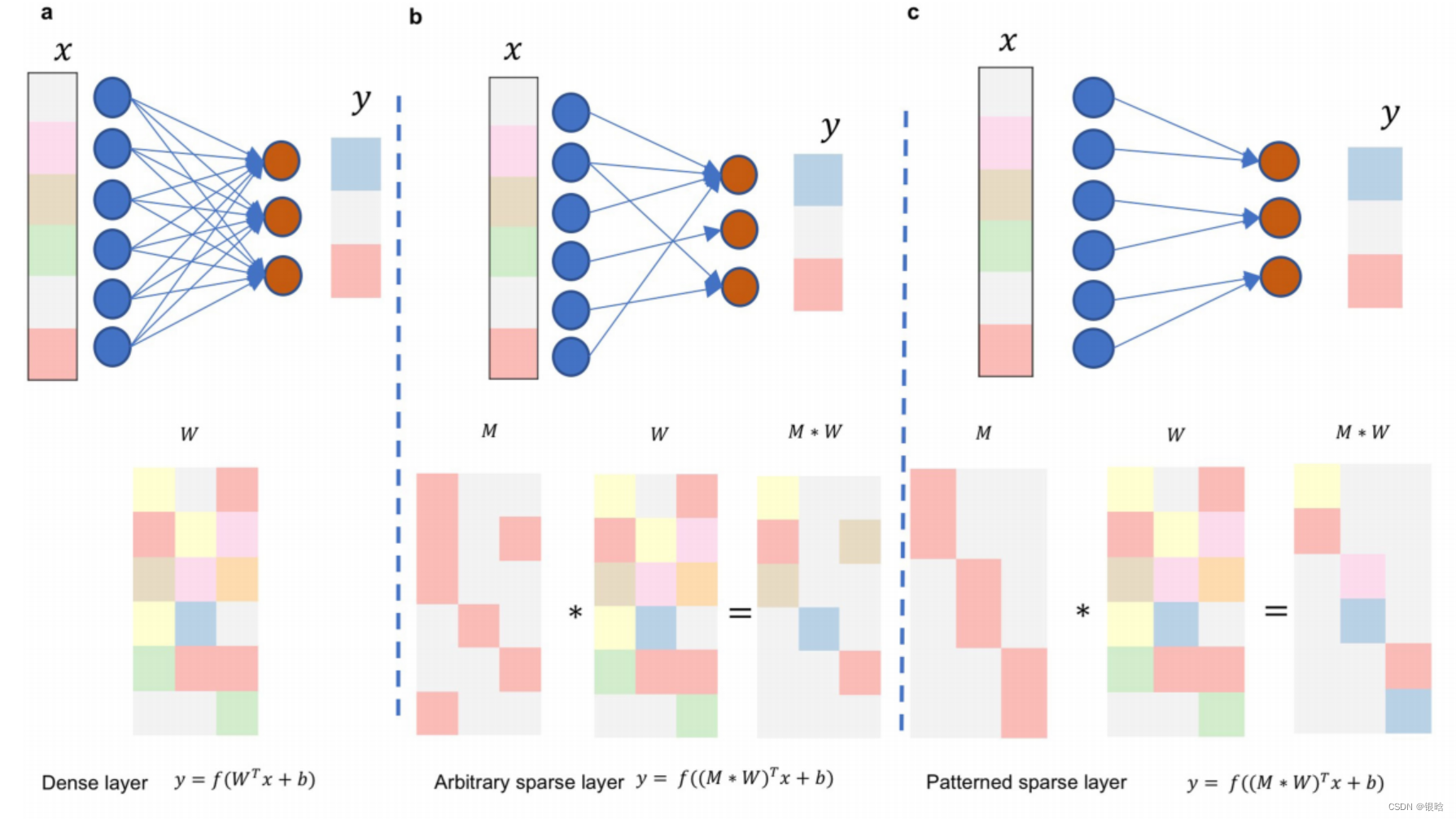
论文:Development and validation of a deep learning algorithm for improving Gleason scoring of prostate cancer(2019)
- 用深度学习系统来进行格里森评分,该系统由两部分构成:CNN( InceptionV3 网络)进行GP特征提取,KNN作分类
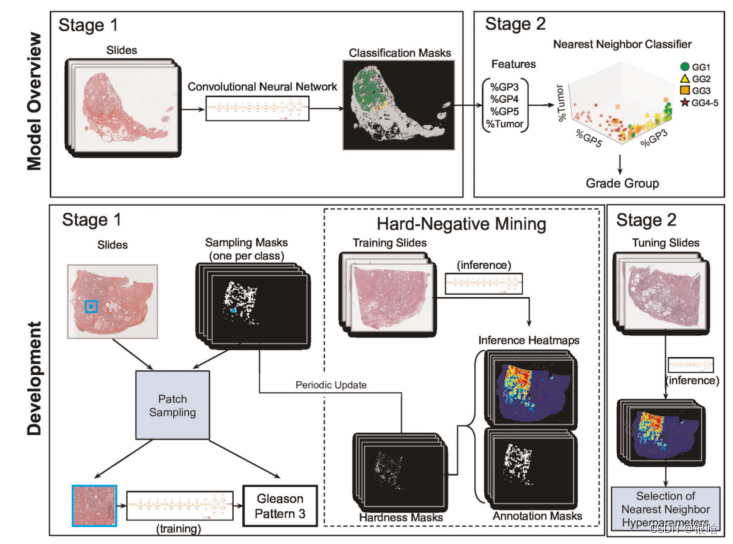
- 每个区域的标签都来自于病理学家提供的区域级标注
- 使用了Ensembling and hard-negative mining方法进行调优,包括了颜色归一化55和交替卷积神经网络结构,但没有显示出任何好处
论文:Clinical-grade computational pathology using weakly supervised deep learning on whole slide images(2020)
-
采用弱监督的多实例算法,避免大量标注数据集;
-
对前列腺癌、基底细胞癌和乳腺癌转移到腋窝淋巴结的检测显示,所有癌症类型的曲线下面积都在0.98以上
-
大多都是用监督学习进行分类(需要大量数据),病理学的数据集都是小而精的
-
数据集构成:
1)前列腺核心活检数据集,共24859张切片;
(2)9962张载玻片的皮肤数据集;
(3)9894张切片,向淋巴结的乳腺转移数据集。
前列腺癌在观察者间和观察者内的高变异性,基于非常小的病变,占整个组织表面积的1%
步骤:
MIL思想:如果在特定包中至少有一个贴片被预测为阳性,则整个包被预测为阳性。
负包内都是负样本;正包内需要进行分类,是正还是负,至少一个是正
- 多实例假设,也就是最大池化
- MIL训练一个基分类器(ResNet18/CNNs),用分类器去进行预测打分,然后结合多种策略或者集成模型进行投票
- 用MIL训练一个深度神经网络表示出title-level的特征,然后用RNN来合并全部的信息,得出最终的结果,即用RNN代替max-pooling
即,MIL-RNN
MIL分类算法的完整管道(图1c)包括以下步骤:
(1)在数据集中放置每张切片(每个epoch,包括整个训练数据);
(2)对所有数据进行完整的推理;
(3)包的切片进行分类和排名;
4)基于每张幻灯片排名靠前的实例进行模型学习。
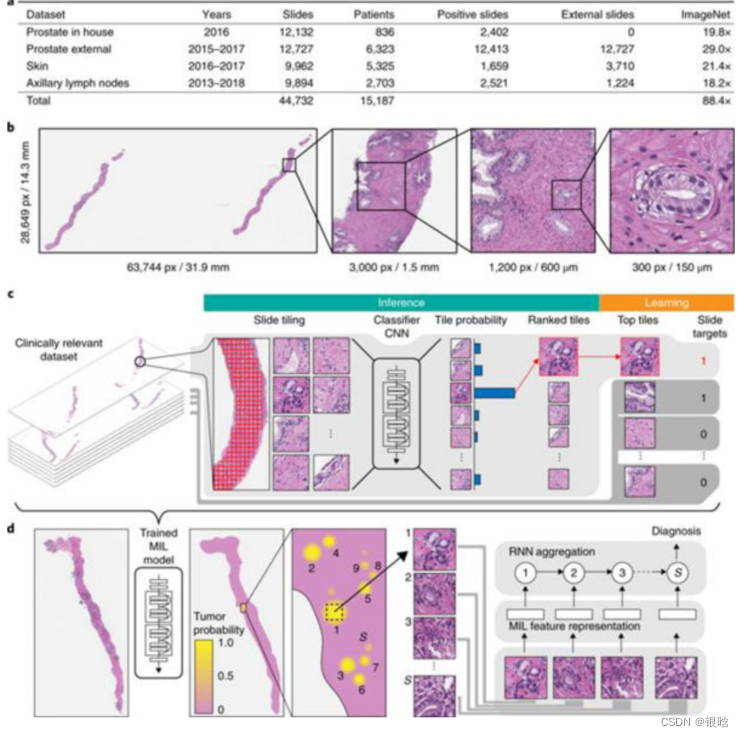
结果
-
用MIL训练ResNet34进行分类
-
数据处理:切片缩略图图像进行阈值处理,以有效地丢弃所有的背景贴图,从而大大减少了每张切片的计算量
-
模型训练:
- 正负样本不平衡,给分配权重
- SGD,Adam ,batchsize : 512 for AlexNet, 256 for
ResNets and 128 for VGGs and DenseNet201. - All models were initialized with ImageNet pretrained weights.
- Early stopping was used to avoid overfitting.
-
通过将每个模型的预测与一个操作符合并,可以创建一个多尺度的集成。我们使用平均池化和最大池化来获得朴素的多尺度模型。
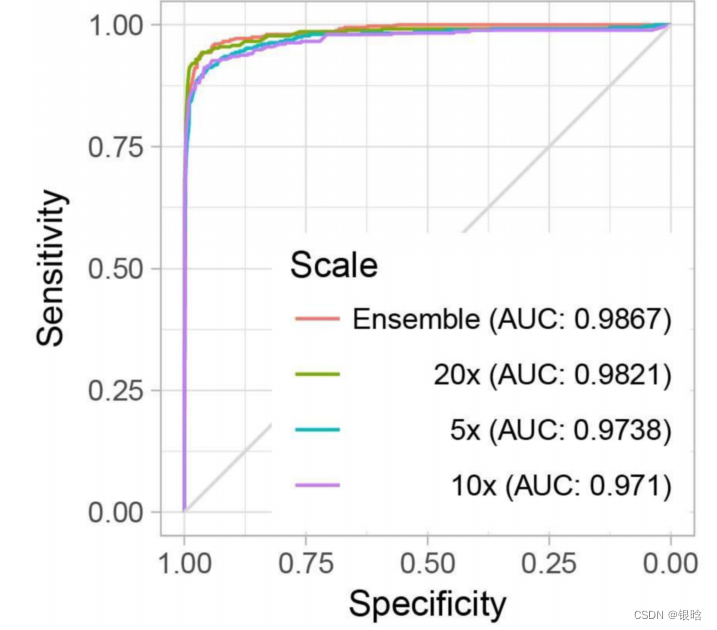
论文:Prostate Cancer Risk Stratification via Nondestructive 3D Pathology with Deep Learning–Assisted Gland Analysis(2022)
前列腺癌的治疗计划在很大程度上依赖于对芯针活检的检查。前列腺的微观结构构成了病理学家进行预后分级的基础。通过对有限数量的二维(2D)组织学切片的视觉检查来解释这些复杂的三维(3D)腺体结构往往是不可靠的,这有助于患者的治疗不足和过度治疗。
为了改进风险评估和治疗决策,我们开发了一个工作流程,用于无损三维病理和计算分析,用快速和廉价的标准苏木精和伊红(H&E)染色荧光类似物标记。这种分析是基于可解释的腺体特征,并通过三维图像翻译辅助分割(ITAS3D)的发展而得到促进。
ITAS3D是一种可推广的基于深度学习的策略,它使组织微观结构能够在注释中进行体积分割,自由和客观的(基于生物标志物)的方式,不需要免疫标记。作为初步证明计算3D与计算2D病理方法的翻译价值的初步证明,我们对从50个存档的根治性前列腺切除术标本中提取的300个体外活检进行了成像,其中118个活检包含癌症。根据低至中危前列腺癌患者的临床生化复发结果,肿瘤活检中的3D腺体特征优于相应的2D特征。本研究的结果支持使用计算三维病理学来指导前列腺癌的临床管理。
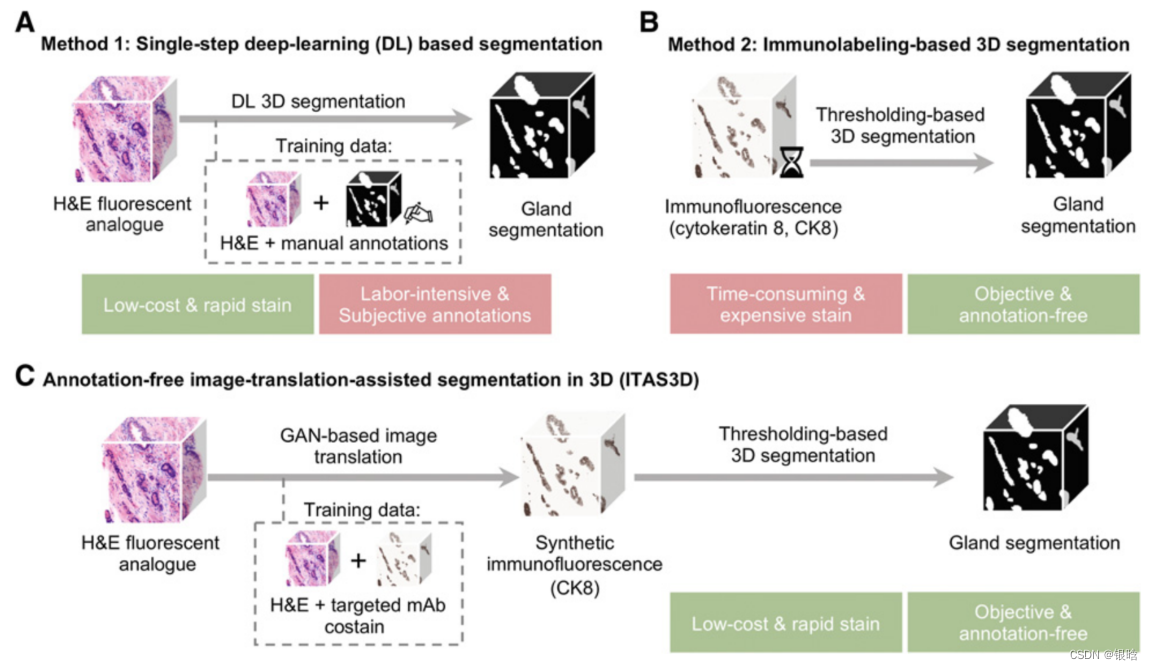
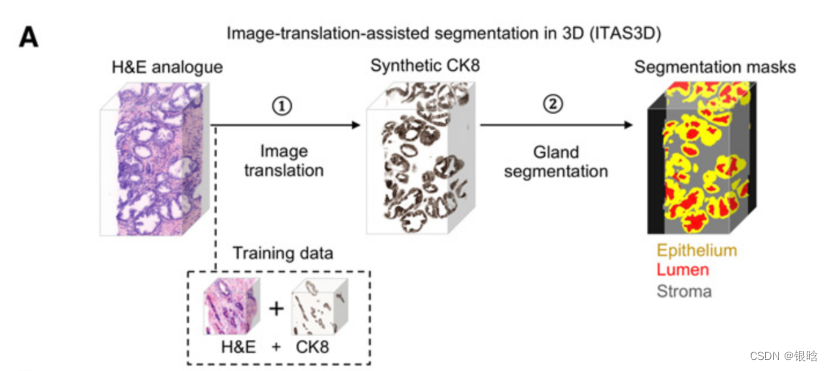
流程
ITAS3D:无需打标签的前列腺3D分割模型
- 一个标本的3D显微镜数据集,用快速和廉价的H&E荧光模拟物染色,通过使用配对的图像序列翻译模型和真实CK8免疫荧光数据集(三标记组织)训练,转换成一个合成的CK8免疫荧光数据集。
CK8生物标志物用于标准护理泌尿生殖系统病理实践,在所有前列腺的管腔上皮细胞中广泛表达。 - 使用图像分割算法对上皮细胞、管腔和周围的基质区域分割
-
image-translation:
- 三维前列腺活检被细分为重叠的块,每个块都被视为二维图像的深度序列。
- 经过gan训练的生成器在图像块的每个二维级别上依次执行图像转换
- 每个级别的图像平移都是基于该级别的H&E模拟输入,同时利用之前两个级别的H&E模拟和CK8图像来加强级别之间的空间连续性(即“2.5D”平移方法)。
- 然后将合成的CK8图像块输出进行拼接,生成整个活检的3D CK8数据集,以协助腺体分割。
-
Gland segement: 使用基于阈值的算法从合成的ck8数据集中分割出上皮细胞层(上皮细胞)。腺体管腔空间通过填充被上皮细胞包围的区域而被分割,并根据细胞质通道(伊红荧光)进行细化
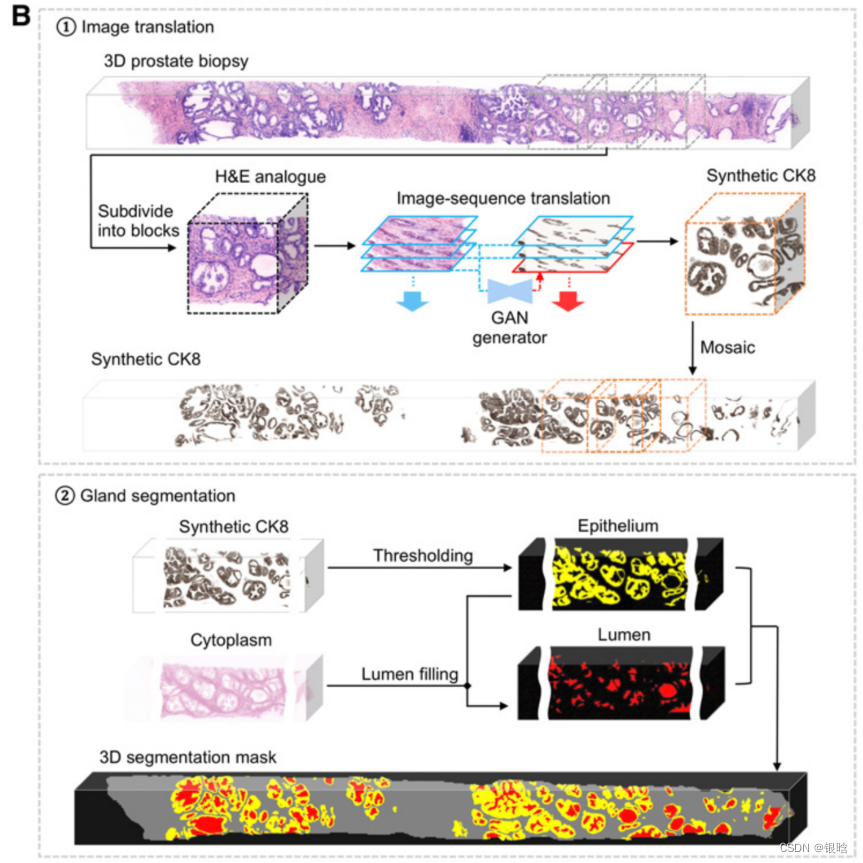
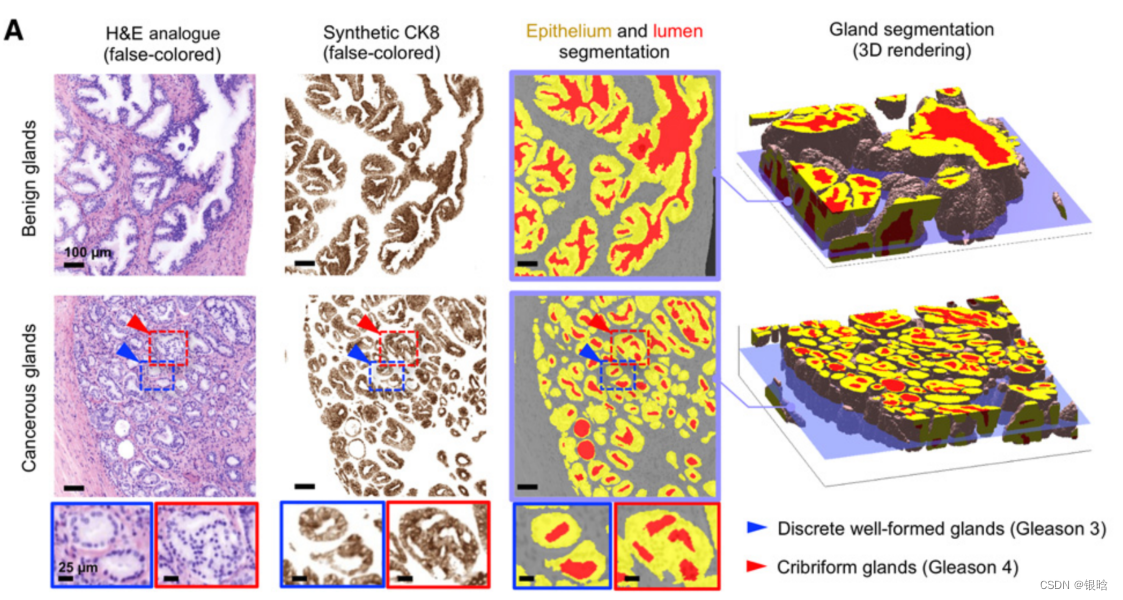
- H&E模拟图像,基于合成ck8图像,最右边显示了一个良性和癌变区域的腺体分割的三维效果图
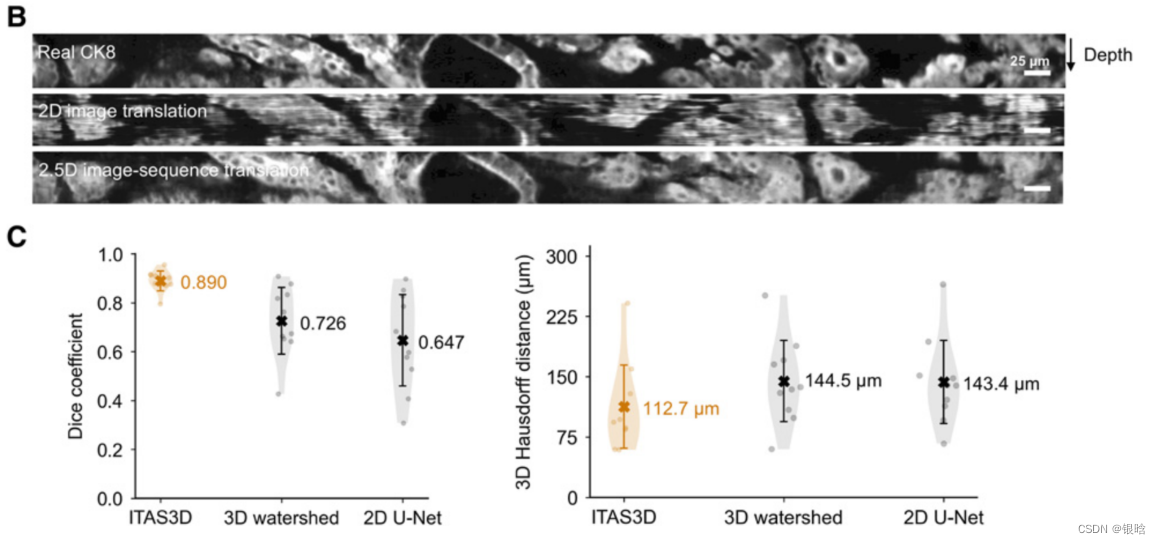
- 真实的和合成的ck8免疫荧光图像的图像序列的侧视图(深度方向向下定向)。与二维图像翻译结果相比,2.5维图像翻译结果显示出显著改善的深度连续性
Application of a novel machine learning framework for predicting non-metastatic prostate cancer-specific mortality in men using the Surveillance, Epidemiology, and End Results (SEER) database
准确的预后对于非转移性前列腺癌患者的治疗决定是至关重要的。目前的模型依赖于预先指定的变量,这限制了它们的性能。我们的目的是研究一种新的机器学习方法,以开发一种改进的预后模型,用于预测10年前列腺癌特异性死亡率,并将其性能与现有的已验证的模型进行比较。
- 利用数据库的大量数据
- 四个模型组合而成:Cox比例风险、随机生存森林、条件推理生存森林和DeepHit模型
Artificial intelligence and pathology: From principles to practice and future applications in histomorphology and molecular profiling(综述文章)
人工智能和病理学:从原则到实践和未来在组织形态学和分子图谱分析中的应用
- 侧重点是病理学的角度,以定量和标准化的方式分析复杂数据,通过组织学图像的分析,以进一步提高诊断的范围和精度
- 可解释性AI,LRP:归因方法如何工作提供了一个很好的直觉:神经网络受到大脑结构的启发,信息通过神经元以一层的方式传播神经元,从输入到最终输出神经元的值决定决策。
- LRP回溯了这个过程,以确定最相关的特性。更详细地说,它从输出神经元开始,通过向后行走神经元图,以递归的方式计算每个神经元所连接神经元的输入的相关性
- 这样做,直到达到输入神经元,这些最终值表示个体输入特征对神经网络决策的“相关性”程度。其他方法也同样基于传播规则,或者采用基于对输入数据的修改的互补方法
Artificial intelligence for diagnosis and grading of prostate
cancer in biopsies: a population-based, diagnostic study
- DNNs进行格里森活检中进行识别、定位和分级,从而提供一个安全网,以防止活检中的潜在错误分类
Artificial intelligence neuropathologist for glioma classification using deep learning on hematoxylin and eosin stained slide images and molecular markers(2020)
- 人工智能神经病理学家使用苏木精和伊红深度学习染色载玻片图像和分子标记进行胶质瘤分类
- 包括切片扫描仪和深度卷积神经网络(CNNs),以对胶质瘤的5种主要组织学亚型进行分类,以协助病理学家。在267例患者的79990多张组织学贴片图像上对cnn进行了训练和验证。当分子图谱可用时,使用逻辑算法
模型
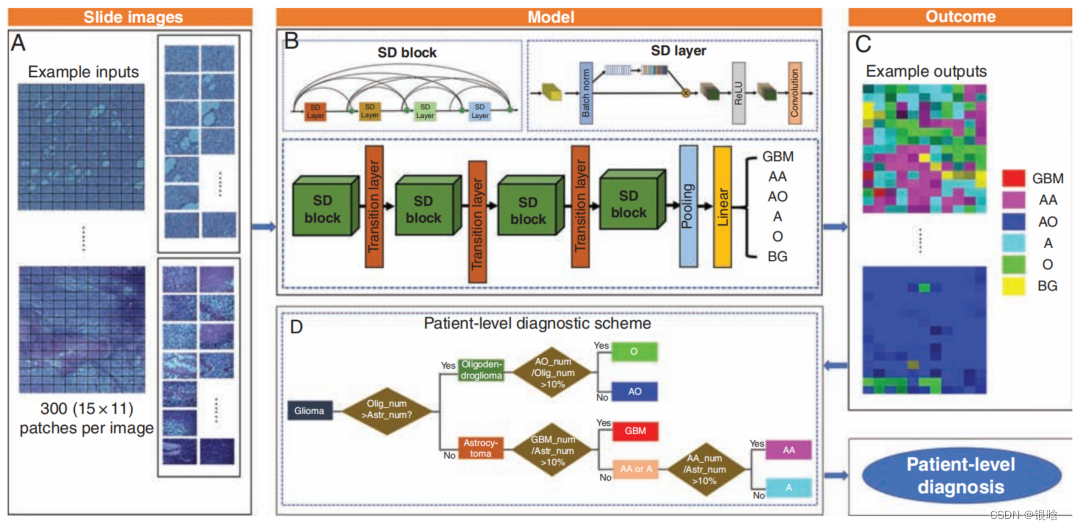
- 一个新的模型的挤压-激励块DenseNet加权交叉熵(称为SDNet_WCE)神经胶质瘤分类任务
思想策略:是开发一个基于图像贴片的诊断模型,然后将贴片预测结果与分子信息相结合,最终纳入患者的综合神经病理学诊断
流程:
- 所有的图像补丁首先进行预处理和重采样,大小为512×384,同时保持图像细节和高宽比,以节省计算GPU内存
- 在训练阶段,为了提高模型的生成能力,我们对每个图像进行了随机刚性变换(包括垂直/水平翻转和旋转)等图像增强过程。
- 为了避免由于光和染色过程的影响而导致的颜色和亮度不一致,每个训练图像的亮度、对比度和颜色都被随机改变后再输入网络
- DenseNet(SD)-Net_WCE的神经网络。简单地说,我们使用DenseNet结构作为主干,其中病理图像被发送到卷积层,这些特征图被用softmax操作符发送到完全卷积层,输出的是神经胶质瘤类别的最终预测。
- 在此基础上,我们在密集层的批量归一化层和整流线性单元层之间添加了挤压和激励块,称为SD-Net
- 由于不同的单元格具有不同的边界形状,由相应的卷积核生成的某些特征图通道对最终的分类任务的贡献更大。因此,添加SD块从理论上可以从理论上提高分类性能。(注意力机制的思想)
- 每个SD块之后都有一个过渡层,最终串联连接,形成所提出的SD-Net。
- 最后,采用修正的加权交叉熵损失作为损失函数,解决胶质瘤类别间数据不平衡的问题,代替DenseNet中原始的交叉熵损失函数。通过这种加权交叉熵损失函数优化的SD-Net被命名为SD-Net-WCE。
- 此外,为了加速收敛速度,还采用了迁移学习策略。也就是说,使用ImageNet数据集上的预训练参数来初始化我们的模型。采用Adam算法对模型进行优化;原来的学习率设置为10-4,权值衰减率设置为(0.9,0.999)。在测试阶段,训练后的模型被用于为测试图像补丁提供神经胶质瘤类别的预测。
数据获取
在本研究中,**通过在常规光学显微镜上添加数码相机和微定位平台,开发了一种低成本的扫描仪,而不是转换的载玻片扫描仪,而不是使用wsi的制造商扫描仪,**如图1所示。将染色的肿瘤切片放置在微定位平台上,每次运动后采集图像。每张切片在6分钟内为幻灯片扫描300个400倍像素的图像的patch,保存到我们的实验室服务器上,然后缝合在一起。
- 共收集了323例分类胶质瘤患者的组织病理学切片中的97 252张组织病理学图像。该数据集来自中枢神经系统疾病生物样本库
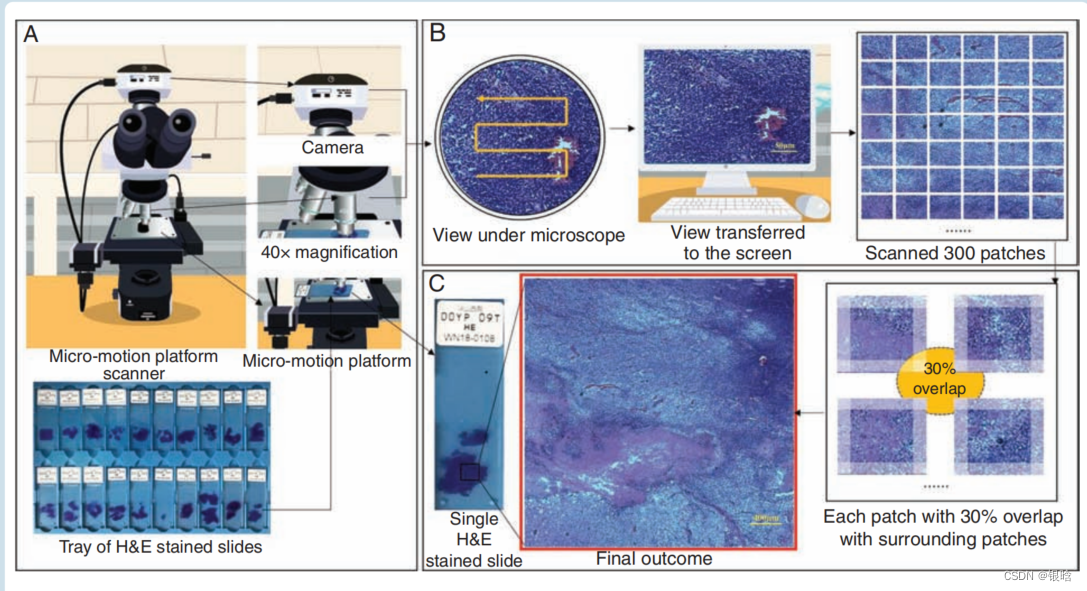
- 图像采集和拼接过程。
(A) 将h&e染色的载玻片样品放置在微运动平台扫描仪上。摄像机装在普通显微镜的顶部,舞台上装有一个烦躁平台以控制载玻片的运动。
(B) 扫描仪操作机制:当相机在显微镜下拍摄照片时,平台以s形移动。该视图被实时传输到计算机屏幕上。该平台允许载玻片每次水平移动0.2毫米或垂直移动0.15毫米。图像是在10个×目镜和40个×物镜的设置下拍摄的,大小为2048×1536。由于该平台水平捕获20张图像,垂直捕获15张图像,共拍摄并保存了300块图像。
(C ) 图像拼接:300个斑块中的每个贴片(图像)与周围斑块有30%的重叠,最终结果为完整的病理图像。