文章目录
最优化之遗传算法
一、 概述
1、 算法简介
计算智能(Computational Intelligence,CI)方法主要包括:
- 神经网络(Neural Network,NN);
- 模糊逻辑(Fuzzy Logic,FL);
- 遗传算法(Genetic Algorithm,GA);
- 蚁群优化算法(Ant Colony Optimization,ACO);
- 粒子群优化算法(Particle Swarm Op);
- 免疫算法(Immune Algorithm,IA);
- 分布估计算法(Estimation of Distribution Algorithm,EDA);
- Memetic算法(Memetic Algorithm,MA);
- 模拟退火(Simulated Annealing,SA);
- 禁忌搜索(Tabu Search,TS)。
后面会一一学习。
遗传算法(Genetic Algorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。遗传算法是从代表问题可能潜在的解集的一个种群(population)开始的,而一个种群则由经过基因(gene)编码的一定数目的个体(individual)组成。每个个体实际上是染色体(chromosome)带有特征的实体。染色体作为遗传物质的主要载体,即多个基因的集合,其内部表现(即基因型)是某种基因组合,它决定了个体的形状的外部表现,如黑头发的特征是由染色体中控制这一特征的某种基因组合决定的。因此,在一开始需要实现从表现型到基因型的映射即编码工作。由于仿照基因编码的工作很复杂,我们往往进行简化,如二进制编码,初代种群产生之后,按照适者生存和优胜劣汰的原理,逐代(generation)演化产生出越来越好的近似解,在每一代,根据问题域中个体的适应度(fitness)大小选择(selection)个体,并借助于自然遗传学的遗传算子(genetic operators)进行组合交叉(crossover)和变异(mutation),产生出代表新的解集的种群。这个过程将导致种群像自然进化一样的后生代种群比前代更加适应于环境,末代种群中的最优个体经过解码(decoding),可以作为问题近似最优解。
主要思想:
- 遗传算法是根据达尔文的“适者生存,优胜劣汰”的思想来找到最优解的额,其特点是所找到的解是全局最优解,相对于蚁群算法可能出现的局部最优解还是有优势的。
主要名词:
- 个体(染色体):一个染色体代表一个具体问题的一个解,一个染色体包含若干基因。
- 基因:一个基因代表具体问题解的一个决策变量。
- 种群:多个个体(染色体)构成一个种群。即一个问题的多组解构成了解的种群。我们的目的就是让种群中”优胜劣汰“,最终只剩下一个最优解。接下来介绍最基本遗传算法,只用了选择,交叉,变异三种遗传算子。
2、 简单实例
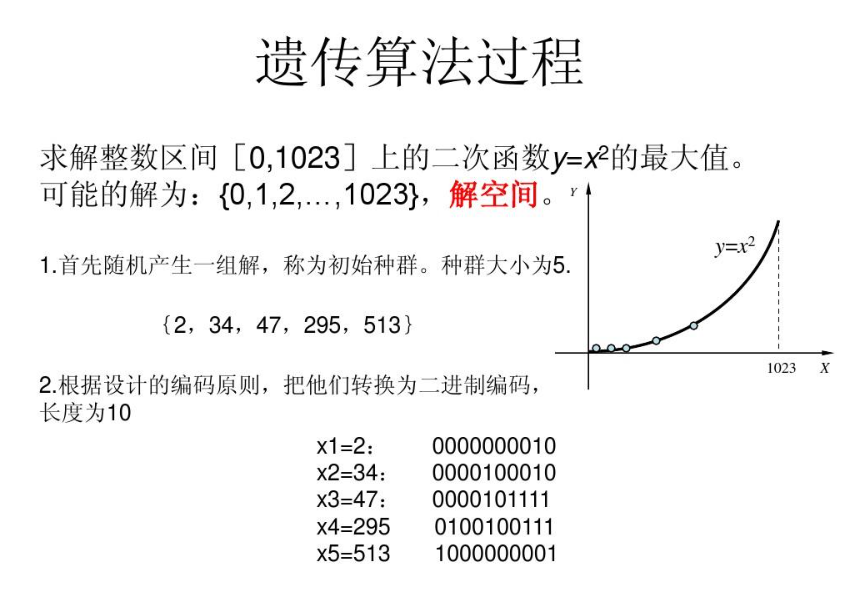
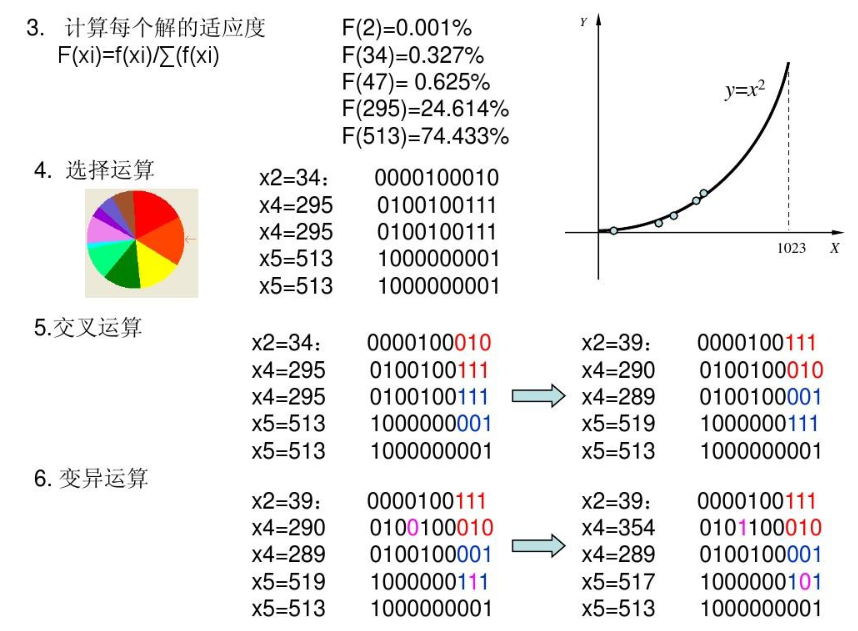
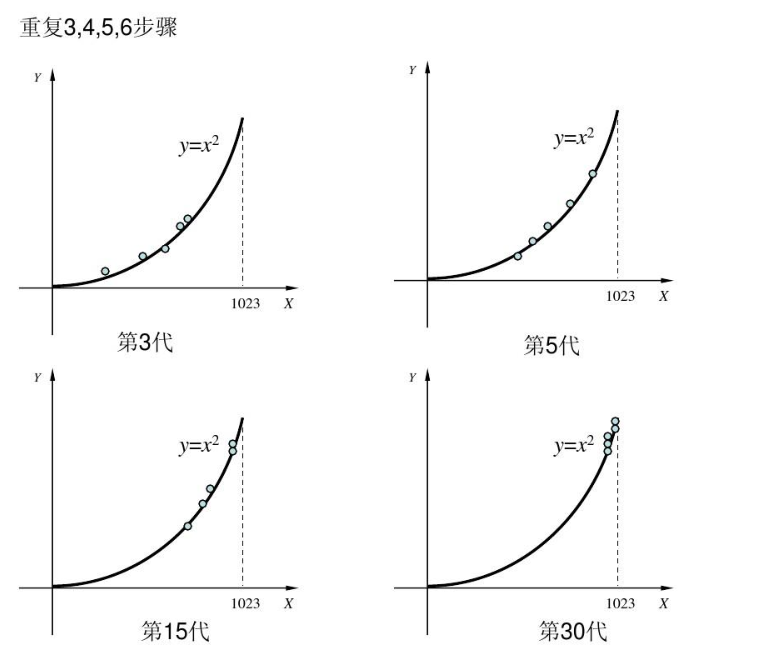
3、 主要步骤
相信大家看完那个简单实例之后,对我们遗传算法的实现原理有了一个初步的认识。现在,让我们来总结一些其实现的步骤。

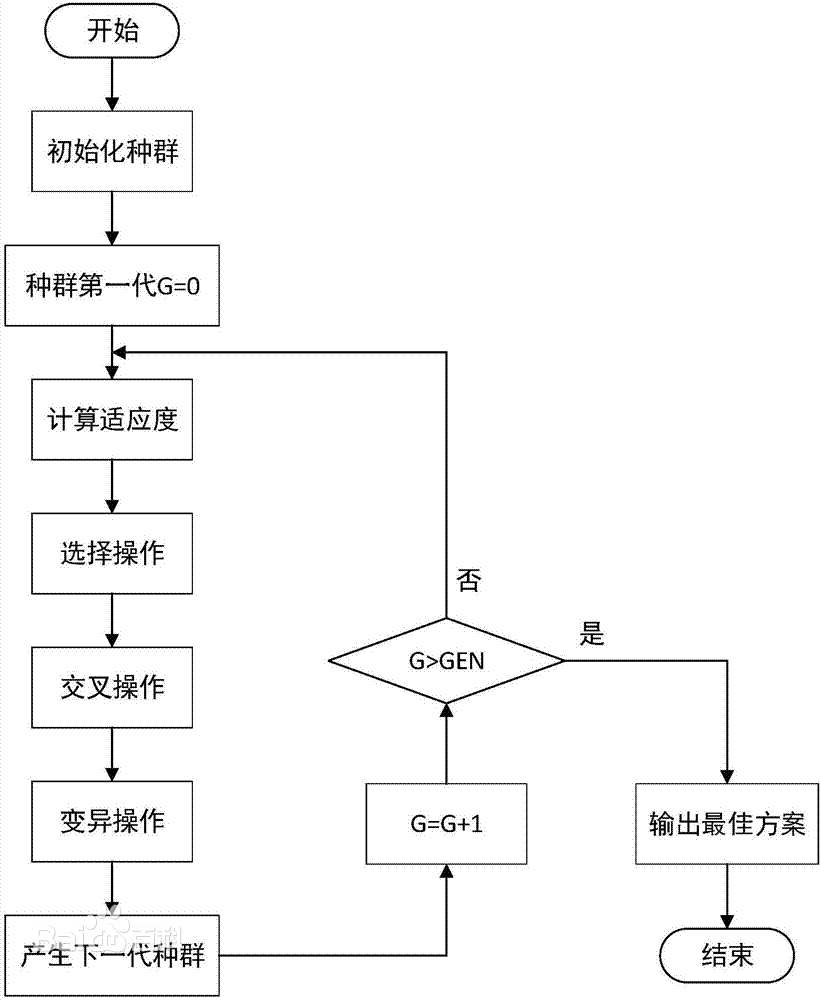
-
种群初始化。我们需要首先通过随机生成的方式来创造一个种群,一般该种群的数量为100~500,这里我们采用二进制将一个染色体(解)编码为基因型。随后用进制转化,将二进制的基因型转化成十进制的表现型。
-
适应度计算(种群评估)。这里我们直接将目标函数值作为个体的适应度。
-
选择(复制)操作。根据种群中个体的适应度大小,通过轮盘赌等方式将适应度高的个体从当前种群中选择出来。其中轮盘赌即是与适应度成正比的概率来确定各个个体遗传到下一代群体中的数量。
具体步骤如下:
- 首先,计算出所有个体的适应度总和 Σfi;
- 其次,计算出每个个体的相对适应度大小
fi/Σfi,类似于softmax; - 再产生一个 0 到 1 之间的随机数,依据随机数出现在上述哪个概率区域内来确定各个个体被选中的次数。
-
交叉(交配)运算。该步骤是遗传算法中产生新的个体的主要操作过程,它用一定的交配概率阈值(pc,一般是0.4到0.99)来控制是否采取单点交叉,多点交叉等方式生成新的交叉个体。
具体步骤如下:
- 先对群体随机配对;
- 再随机设定交叉点的位置;
- 再互换配对染色体间的部分基因。
-
变异运算。该步骤是产生新的个体的另一种操作。一般先随机产生变异点,再根据变异概率阈值(pm,一般是0.0001到0.1)将变异点的原有基因取反。
-
终止判断。如果满足条件(迭代次数,一般是200~500)则终止算法,否则返回 step2 。
二、 步骤详解
先来看一个简单的函数优化的实例,来体验下遗传算法是如何实际应用的。
已知  ,求函数y的最大值。要求解精确到小数点后4 位。
,求函数y的最大值。要求解精确到小数点后4 位。

1、 染色体编码
在遗传算法中,问题的每个有效解被称为一个“染色体 (chromosome)”,也称为“串”,对应于种群中的每个生物个体(individual)。
比如在这个问题中,每一个符合条件的有效解 ( x 1 , x 2 , x 3 , x 4 ) (x1, x2, x3, x4) (x1,x2,x3,x4),即表示一个染色体。
许多应用问题的结构很复杂,我们希望找到一种既简单又不影响算法性能的编码方式。
- 将问题结构变换为位串形式编码表示的过程叫做编码;
- 将位串形式编码表示变换为原问题结构的过程叫做解码或译码。
遗传算法中,一般的编码方法分为2种。
第1种是二进制编码方法。二进制编码方法产生的染色体是一个二进制符号序列,染色体的每一个基因只能取值 0 或 1 。
假定问题定义的有效解取值空间为 [ U m i n , U m a x ] [U_{min},U_{max}] [Umin,Umax] ,使用 L 位二进制符号串表示解的一维变量 ,则我们可以得到如下表所示的编码方式。

第2种是浮点数编码方法。浮点数编码方法中每个染色体用某一范围内的一个浮点数来表示,染色体的编码长度等于问题定义的解的变量个数,染色体的每一个基因等于解的每一维变量。其中使用D作为有效解的变量维数。
待求解问题的一个有效解为 $X_i=(x_i1,x_i2,⋯,x_i{D-1},x_i{D}) $。则该解对应的染色体编码为 ( x i 1 , x i 2 , ⋯ , x i D − 1 , x i D ) (x_i^1,x_i^2,⋯,x_i^{D-1},x_i^D) (xi1,xi2,⋯,xiD−1,xiD) 。
因为这种编码方法使用的是变量的真实值,所以浮点数编码方法也叫做真值编码方法。对于一些多维、高精度要求的连续函数优化问题,用浮点数编码来表示个体时将会有一些益处。
在我们的实例中,我们采用第二种浮点数编码方法构造染色体,即每个染色体以 ( x 1 , x 2 , x 3 , x 4 ) (x_1,x_2,x_3,x_4) (x1,x2,x3,x4) 的形式表示。
2、 种群初始化
采用生成随机数的方法,对染色体的每一维变量进行初始化赋值。初始化染色体时必须满足优化问题对有效解的定义。产生出规模为 N 的种群集合。
在我们的实例中,我们假设种群规模为5,随机初始化种群的染色体,得到:

3、 适应度评价
为了体现染色体的适应能力,引入了对每一个染色体都能进行度量的函数,叫做适应度函数 (fitness function)。通过计算适应度函数值,来决定染色体的优劣程度,它体现了自然进化中的优胜劣汰原则。
在遗传算法中,规定适应值越大的染色体越优。适应度函数要求能有效地反映每一个染色体的适应能力。若一个染色体与问题的最优解染色体之间的差距较小,则对应的适应度函数值就会较大。
评估函数常常根据问题的优化目标来确定,例如求解函数优化问题时,问题定义的目标函数可以作为评估函数的原型。
对于一些求解最大值的数值优化问题,我们可以直接套用问题定义的函数表达式。但是对于其他优化问题,问题定义的目标函数表达式必须经过一定的变换。
在我们的实例中,由于本身就是求解最大值的,故可以直接使用y作为适应值评价函数。即:
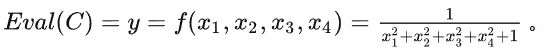
计算每个染色体的适应值如下:

因此,初始条件下的最优解为:
4、 选择算子
选择操作也叫做复制(reproduction)操作,根据个体的适应度函数值所度量的优劣程度决定它在下一代是被淘汰还是被遗传。
一般地,适应度较大(优良)的个体有较大的存在机会,而适应度较小(低劣)的个体继续存在的机会也较小。简单遗传算法采用赌轮选择机制。
根据每个染色体的适应值得到种群中所有染色体的适应值总和。计算每个染色体适应值与种群适应值总和的比 P i P_i Pi。
假设一个具有N个扇区的轮盘,每个扇区对应种群中的一个染色体,扇区的大小与对应染色体的 P i P_i Pi 值成正比。
轮盘赌模型的计算机实现可以参照下面实例中的操作方式。
在实例中,采用轮盘赌算法,计算种群适应度总和为0.0373603+0.0255988+0.0448529+0.0238214+0.0331319=0.164765。分别计算每个染色体适应值同种群适应值总和的比, [ C 1 , C 2 , C 3 , C 4 , C 5 ] [C_1, C_2, C_3, C_4, C_5] [C1,C2,C3,C4,C5] 依次为0.226749,0.155365,0.272223,0.144578,0.201085。累计值依次为0.226749,0.382114,0.654337,0.798915,1。
下面从[0,1]之间随机地产生5个随机数,落在哪个区间内,选择对应的染色体。
(1): 0.278756 对应选择的是 C 2 C_2 C2;
(2): 0.604389 对应选择的是 C 3 C_3 C3;
(3): 0.230964 对应选择的是 C 2 C_2 C2;
(4): 0.376263 对应选择的是 C 2 C_2 C2;
(5): 0.858791 对应选择的是 C 5 C_5 C5。
经过选择算子以后,得到的种群即为:

5、 交叉算子
交叉操作的简单方式是: 按交叉概率 P c P_c Pc 选择出两个父代个体 P 1 P_1 P1 和 P 2 P_2 P2 ,将两者的部分基因(码值)进行交换。(当然也可以采取其他方式,目的在于打乱顺序且不会错乱溢出。)
交叉概率的 P c P_c Pc 一般取值为:0.4 到 0.99 之间。随机数 Random(0, 1) 小于 P c P_c Pc ,则表示该染色体可进行交叉操作。随机产生一个有效的交配位置,父代染色体交换位于该交配位置后的所有基因。
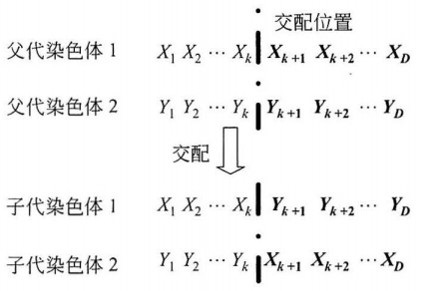
在我们的实例中,假设交配概率为 0.88。下面生成 5 个 0 到 1 之间的随机数,决定这 5 个染色体是否参加交配。
(1): 0.341044<0.88,故 C 1 ′ C_1^{'} C1′ 参加交配;
(2): 0.613782<0.88,故 C 2 ′ C_2^{'} C2′ 参加交配;
(3): 0.972173>0.88,故 C 3 ′ C_3^{'} C3′ 不参加交配;
(4): 0.310424<0.88 ,故 C 4 ′ C_4^{'} C4′ 参加交配;
(5): 0.672331<0.88,故 C 5 ′ C_5^{'} C5′ 参加交配。
即 C 1 ′ C_1^{'} C1′ 和 C 2 ′ C_2^{'} C2′ , C 4 ′ C_4^{'} C4′ 和 C 5 ′ C_5^{'} C5′ 进行交配,每对染色体交配时随机生成1到4之间的自然数作为交配位

这样交叉操作之后得到的新种群为:

6、 变异算子
变异操作的简单方式就是改变码串中某个位置上的数码,变异概率的 P m P_m Pm 一般取值为:0.001 到 0.2 之间。随机数 Random(0, 1) 小于 P m P_m Pm ,则表示该染色体可进行变异操作随机产生一个有效的变异位置,染色体上位于该位置的基因发生改变。
二进制编码表示的每个位置的数码只有0和1两种可能,二进制编码的简单变异操作是将0 与1互换:0变异为1,1变异为0。浮点数编码形式的染色体若某基因发生变异,则可采用随机数方法产生一个满足问题定义的数值 取代该基因现有的值。
在实例当中,假设变异概率为0.1。对于5个染色体的20个基因随机生成0 到1 之间的随机数,若该随机数小于0.1,则改变该基因的值为另一符合条件的值作为变异,否则不改变基因的值。
以下是发生变异的两个染色体和基因改变的过程:

7、 适者生存
重新计算新种群中各个染色体的适应值。倘若新种群的最大适应值大于现在的最佳适应值, 则以该最大适应值对应的染色体更新为最优染色体。
在我们的例子中,计算新种群中每个染色体的适应值如下:

8、 终止条件
决定算法何时停止运行,输出找到的最优解。采用何种终止条件,跟具体问题的应用有关。
可以使算法在达到最大进化代数时停止,最大进化代数一般可设置为 100 代到 1000 代,根据具体问题可对该建议值作相应的修改。也可以通过考察找到的当前最优解的情况来控制算法的停止。
例如,当目前进化过程算法找到的最优解达到一定的误差要求,则算法可以停止。 误差范围的设置同样跟具体的优化问题相关。或者是算法在持续很长的一段进化时间内所找到的最优解没有得到改善时,算法可以停止。
三、 python实现
1、 种群初始化
这里我们需要初始化一个种群:
def init(N_species):
"""
生成 -1 ~ 1 之间的随机数
Parameters
----------
N : int
需要初始化的种群数量.
Returns
-------
species : list
返回生成的初始化种群
"""
species = []
for i in range(N_species):
species.append([
random() * 2 - 1, random() * 2 - 1, random() * 2 - 1, random() * 2 - 1
])
return species
我们使用的是浮点数编码,则这里可以跳过编码的环节。
2、 计算适应度
首先,我们需要确定我们的目标是什么,由此来确定适应度的表达方式
def f(x_1, x_2, x_3, x_4):
return 1 / (pow(x_1, 2) + pow(x_2, 2) + pow(x_3, 2) + pow(x_4, 2) + 1)
def fitness(f, species):
"""
计算种群适应度
Parameters
----------
f : function
传入需要求最大值的函数.
species : list
传入种群所有的种群里面的值.
Returns
-------
fitness : list
返回适应度占比组成的列表.
best : double
返回当前的最优解
"""
_fitness = []
for i in species:
val = f(*tuple(i)) # 计算出函数值
_fitness.append(val)
return _fitness
def bests(population, _fit):
"""
获取种群中,最优的个体,返回二进制数据以及适应度
Parameters
----------
population : list
传入种群.
_fit : list
传入种群的适应度.
Returns
-------
best_individual : double
返回最优个体的具体数值.
best_fit : double
返回最优的适应度.
"""
best_individual_lis, best_fit = [], _fit[0]
# 查找种群中最优的个体
for i in range(len(population)):
if best_fit < _fit[i]:
best_fit = _fit[i] # 获得适应度最高的个体
best_individual_lis = population[i] # 传入染色体
return [best_fit, best_individual_lis]
由上面的的选择操作可知,适应度是选择操作的主要参考依据,适应度函数(Fitness Function)的选取直接影响到遗传算法的收敛速度以及能否找到最优解。因而适应度函数的选择问题在遗传算法中是一项很值得研究的课题。一般情况下,关于适应度与目标函数的选择有以下这两种关系:
3、 选择算子
def cumsum(fitness):
"""
传入计算的适应度,给适应度划分区间;
计算适应度斐波纳挈列表,这里是为了求出累积的适应度
Parameters
----------
fitness : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
for i in range(len(fitness) - 1, -1, -1):
total = 0
j = 0
while(j <= i):
total += fitness[j]
j += 1
# 这里是为了将适应度划分成区间
fitness[i] = total
def selection(population, _fit):
"""
使用轮盘赌的方法来筛选优势种群
Parameters
----------
population : list
传入种群.
_fit : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
relative_fitness = []
# 将所有个体的适应度概率化,类似于softmax
total_fit = sum(_fit)
for i in _fit:
relative_fitness.append(i / total_fit)
# 将适应度转换为斐波那契列表
cumsum(relative_fitness)
# 使用随机数的方法,来确定哪些种群可以存活
# 产生种群个数的随机值
exist_population = []
for i in range(len(population)):
exist_population.append(random())
exist_population.sort() # 升序排列,会从低到高不断匹配种群
new_population = []
fit_i, new_i = 0, 0 # 分别存放适应度指针,和随机数指针
while new_i < len(population):
if (exist_population[new_i] < relative_fitness[fit_i]):
new_population.append(population[fit_i])
new_i += 1
else:
fit_i += 1
population = new_population
4、 交叉算子
def crossover(population, chromosome_length, Cross_rate):
"""
进行交叉操作,这里是利用列表,直接对原数据进行修改,不设置返回值
这里选择的是一个双点交叉,可以进行多点交叉
Parameters
----------
population : list
传入种群.
chromosome_length : int
染色体长度.
Cross_rate : double
交叉概率,[0.4, 0.99].
Returns
-------
None.
"""
for i in range(len(population) - 1):
if random() > Cross_rate: # 如果大于交叉概率,则不进行交叉
continue
# 这里进行双点交叉
num1, num2 = randint(0, round(chromosome_length / 2) ), randint(round(chromosome_length / 2), chromosome_length - 1)
population[i][num1:num2], population[i + 1][num1:num2] = population[i + 1][num1:num2], population[i][num1:num2]
# 把第 i + 1 个染色体中的后 num1 到第 i + 1 个染色体中的 num2 与 i 的互换
5、 变异算子
def mutation(Muta_rate, species):
"""
进行变异操作,这里是利用列表,直接对原数据进行修改,不设置返回值
这里选择的是单点变异
Parameters
----------
Muta_rate : double
变异概率,[0.001, 0.2].
species : list
种群.
Returns
-------
None.
"""
for i in range(len(species)):
if random() < Muta_rate:
muta_num = randint(0, 3)
species[i][muta_num] = random() * 2 - 1
6、 主程序
def main(N_species, Cross_rate, Muta_rate, max_iteration, results):
"""
主函数
Parameters
----------
N_species : int
种群的数量.
Cross_rate : double
交叉概率.
Muta_rate : double
变异概率.
max_iteration : int
最大繁衍的代数.
results : list
用来存放最优适应度.
Returns
-------
status_code.
"""
species = init(N_species)
for i in range(max_iteration):
_fitness = fitness(f, species)
results.append(bests(species, _fitness))
selection(species, _fitness) # 进行选择操作
crossover(species, 4, Cross_rate) # 进行交叉操作
mutation(Muta_rate, species) # 进行变异操作
results.sort()
X, Y = [], []
for i in range(max_iteration):
X.append(i)
Y.append(results[i][0])
plt.plot(X, Y)
return 0
7、 总代码
# -*- coding: utf-8 -*-
"""
Created on Wed Mar 29 19:09:23 2023
@author: steve
"""
from random import (
random, randint
)
import matplotlib.pyplot as plt
def init(N_species):
"""
生成 -1 ~ 1 之间的随机数
Parameters
----------
N : int
需要初始化的种群数量.
Returns
-------
species : list
返回生成的初始化种群
"""
species = []
for i in range(N_species):
species.append([
random() * 2 - 1, random() * 2 - 1, random() * 2 - 1, random() * 2 - 1
])
return species
def f(x_1, x_2, x_3, x_4):
return 1 / (pow(x_1, 2) + pow(x_2, 2) + pow(x_3, 2) + pow(x_4, 2) + 1)
def fitness(f, species):
"""
计算种群适应度
Parameters
----------
f : function
传入需要求最大值的函数.
species : list
传入种群所有的种群里面的值.
Returns
-------
fitness : list
返回适应度占比组成的列表.
best : double
返回当前的最优解
"""
_fitness = []
for i in species:
val = f(*tuple(i)) # 计算出函数值
_fitness.append(val)
return _fitness
def cumsum(fitness):
"""
传入计算的适应度,给适应度划分区间;
计算适应度斐波纳挈列表,这里是为了求出累积的适应度
Parameters
----------
fitness : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
for i in range(len(fitness) - 1, -1, -1):
total = 0
j = 0
while(j <= i):
total += fitness[j]
j += 1
# 这里是为了将适应度划分成区间
fitness[i] = total
def selection(population, _fit):
"""
使用轮盘赌的方法来筛选优势种群
Parameters
----------
population : list
传入种群.
_fit : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
relative_fitness = []
# 将所有个体的适应度概率化,类似于softmax
total_fit = sum(_fit)
for i in _fit:
relative_fitness.append(i / total_fit)
# 将适应度转换为斐波那契列表
cumsum(relative_fitness)
# 使用随机数的方法,来确定哪些种群可以存活
# 产生种群个数的随机值
exist_population = []
for i in range(len(population)):
exist_population.append(random())
exist_population.sort() # 升序排列,会从低到高不断匹配种群
new_population = []
fit_i, new_i = 0, 0 # 分别存放适应度指针,和随机数指针
while new_i < len(population):
if (exist_population[new_i] < relative_fitness[fit_i]):
new_population.append(population[fit_i])
new_i += 1
else:
fit_i += 1
population = new_population
def crossover(population, chromosome_length, Cross_rate):
"""
进行交叉操作,这里是利用列表,直接对原数据进行修改,不设置返回值
这里选择的是一个双点交叉,可以进行多点交叉
Parameters
----------
population : list
传入种群.
chromosome_length : int
染色体长度.
Cross_rate : double
交叉概率,[0.4, 0.99].
Returns
-------
None.
"""
for i in range(len(population) - 1):
if random() > Cross_rate: # 如果大于交叉概率,则不进行交叉
continue
# 这里进行双点交叉
num1, num2 = randint(0, round(chromosome_length / 2) ), randint(round(chromosome_length / 2), chromosome_length - 1)
population[i][num1:num2], population[i + 1][num1:num2] = population[i + 1][num1:num2], population[i][num1:num2]
# 把第 i + 1 个染色体中的后 num1 到第 i + 1 个染色体中的 num2 与 i 的互换
def mutation(Muta_rate, species):
"""
进行变异操作,这里是利用列表,直接对原数据进行修改,不设置返回值
这里选择的是单点变异
Parameters
----------
Muta_rate : double
变异概率,[0.001, 0.2].
species : list
种群.
Returns
-------
None.
"""
for i in range(len(species)):
if random() < Muta_rate:
muta_num = randint(0, 3)
species[i][muta_num] = random() * 2 - 1
def bests(population, _fit):
"""
获取种群中,最优的个体,返回二进制数据以及适应度
Parameters
----------
population : list
传入种群.
_fit : list
传入种群的适应度.
Returns
-------
best_individual : double
返回最优个体的具体数值.
best_fit : double
返回最优的适应度.
"""
best_individual_lis, best_fit = [], _fit[0]
# 查找种群中最优的个体
for i in range(len(population)):
if best_fit < _fit[i]:
best_fit = _fit[i] # 获得适应度最高的个体
best_individual_lis = population[i] # 传入染色体
return [best_fit, best_individual_lis]
def main(N_species, Cross_rate, Muta_rate, max_iteration, results):
"""
主函数
Parameters
----------
N_species : int
种群的数量.
Cross_rate : double
交叉概率.
Muta_rate : double
变异概率.
max_iteration : int
最大繁衍的代数.
results : list
用来存放最优适应度.
Returns
-------
status_code.
"""
species = init(N_species)
for i in range(max_iteration):
_fitness = fitness(f, species)
results.append(bests(species, _fitness))
selection(species, _fitness) # 进行选择操作
crossover(species, 4, Cross_rate) # 进行交叉操作
mutation(Muta_rate, species) # 进行变异操作
results.sort()
X, Y = [], []
for i in range(max_iteration):
X.append(i)
Y.append(results[i][0])
plt.plot(X, Y)
return 0
if __name__ == "__main__":
N_species = 60 # 种群的数量,最好选择偶数,便于进行交叉
Cross_rate = 0.5 # 交叉概率,[0.4, 0.99]
Muta_rate = 0.01 # 变异概率,[0.001, 0.2]
max_iteration = 1000 # 最大繁殖的代数,[100, 1000]
results = [] # 用来存放每一次的最优适应度
main(N_species, Cross_rate, Muta_rate, max_iteration, results)
四、 案例
1、 需求分析
求 y = 2 ∗ s i n ( x ) + c o s ( x ) y = 2*sin(x)+cos(x) y=2∗sin(x)+cos(x)的最大值:
分析步骤:
2、 实现代码
# -*- coding: utf-8 -*-
"""
Created on Thu Mar 30 10:47:02 2023
需求: 目标求解2*sin(x)+cos(x)最大值
@author: steve
"""
from random import (
randint, random
)
from math import (
sin, cos
)
from matplotlib import pyplot as plt
f = lambda x : 2*sin(x)+cos(x) # 我们的目标函数
def species_origin(population_size, chromosome_length):
"""
进行种群的初始化操作
Parameters
----------
population_size : int
种群的规模.
chromosome_length : int
个体中染色体的数量.
Returns
-------
list
返回生成的种群,其是一个二维数组.
"""
populations = [] # 用来存放初始化完成的种群
for i in range(population_size):
temp_chromoseome = [] # 用来存放生成的染色体
for j in range(chromosome_length):
temp_chromoseome.append(randint(0, 1)) # 随机产生一个染色体,存入准备好的容器中
populations.append(temp_chromoseome) # 将生成的个体存入准备好的容器中
return populations # 将种群返回,种群是个二维数组,个体和染色体两维
def translation(populations, chromosome_length):
"""
进行二进制到十进制数据的转换
Parameters
----------
populations : list
传入一个种群.
chromosome_length : int
传入每个个体染色体的数量.
Returns
-------
list
返回转换后的十进制数据.
"""
temp_data = []
for population in populations:
total = 0
for (index, value) in enumerate(population):
num = chromosome_length - index - 1 # 二进制数据转换是从右到左的
total += value * pow(2, num) # 二进制转换为十进制的方法是按权相加
temp_data.append(total) # 将十进制的值存入容器中
return temp_data # 将十进制数据返回
def fitness(populations, chromosome_length, max_value):
"""
目标函数相当于环境 对染色体进行筛选,这里是2*sin(x)+cos(x)
这里是计算函数的值
这里求的是最大值:
小于零的函数值,其几乎没什么作用,但是又会对整体的适应度之和照成影响,把其取为 0
Parameters
----------
populations : list
种群.
chromosome_length : int
染色体数量.
max_value : int
x 的最大值.
Returns
-------
list
返回函数值构成的列表.
"""
data = translation(populations, chromosome_length) # 进行数据的反编码
temp_fit = []
for i in data:
# 首先,我们需要对数据进行
x = i * max_value / (pow(2,chromosome_length)-1) # 计算出基因的值
temp_fit.append(f(x) if f(x) > 0 else 0) # 计算出函数的 y 值,并添加到容器中,小于0的y值,等于 0
return temp_fit
def cumsum(fitness):
"""
传入计算的适应度,给适应度划分区间;
计算适应度斐波纳挈列表,这里是为了求出累积的适应度
Parameters
----------
fitness : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
for i in range(len(fitness) - 1, -1, -1):
total = 0
j = 0
while(j <= i):
total += fitness[j]
j += 1
# 这里是为了将适应度划分成区间
fitness[i] = total
def selection(population, _fit):
"""
使用轮盘赌的方法来筛选优势种群
Parameters
----------
population : list
传入种群.
_fit : list
传入适应度.
Returns
-------
None.
使用指针,直接修改原列表.
"""
relative_fitness = []
# 将所有个体的适应度概率化,类似于softmax
total_fit = sum(_fit)
for i in _fit:
relative_fitness.append(i / total_fit)
# 将适应度转换为斐波那契列表
cumsum(relative_fitness)
# 使用随机数的方法,来确定哪些种群可以存活
# 产生种群个数的随机值
exist_population = []
for i in range(len(population)):
exist_population.append(random())
exist_population.sort() # 升序排列,会从低到高不断匹配种群
new_population = []
fit_i, new_i = 0, 0 # 分别存放适应度指针,和随机数指针
while new_i < len(population):
if (exist_population[new_i] < relative_fitness[fit_i]):
new_population.append(population[fit_i])
new_i += 1
else:
fit_i += 1
population = new_population
def crossover(population, chromosome_length, Cross_rate):
"""
进行交叉操作,这里是利用列表,直接对原数据进行修改,不设置返回值
这里选择的是一个双点交叉,可以进行多点交叉
Parameters
----------
population : list
传入种群.
chromosome_length : int
染色体长度.
Cross_rate : double
交叉概率,[0.4, 0.99].
Returns
-------
None.
"""
for i in range(len(population) - 1):
if random() > Cross_rate: # 如果大于交叉概率,则不进行交叉
continue
# 这里进行双点交叉
num1, num2 = randint(0, round(chromosome_length / 2) ), randint(round(chromosome_length / 2), chromosome_length - 1)
population[i][num1:num2], population[i + 1][num1:num2] = population[i + 1][num1:num2], population[i][num1:num2]
# 把第 i + 1 个染色体中的后 num1 到第 i + 1 个染色体中的 num2 与 i 的互换
def mutation(population, chromosome_length, Muta_rate):
"""
进行变异操作
Parameters
----------
population : list
传入种群.
chromosome_length : TYPE
传入基因的长度.
Muta_rate : TYPE
传入变异率,[0.001, 0.2].
Returns
-------
None.
"""
for i in range(len(population) - 1):
if random() > Muta_rate: # 如果大于变异率,则不进行变异
continue
num = randint(0, chromosome_length - 1) # 寻找变异位点
population[i][num] = 0 if population[i][num] == 1 else 1 # 进行位点的变化
def bests(population, chromosome_length, max_value, _fit):
"""
获取种群中,最优的个体,返回二进制数据以及适应度
Parameters
----------
population : list
传入种群.
chromosome_length : int
个体中染色体的数量.
max_value : int
个体的最大值.
_fit : list
传入种群的适应度.
Returns
-------
best_individual : double
返回最优个体的具体数值.
best_fit : double
返回最优的适应度.
"""
best_individual_lis, best_fit = [], _fit[0]
# 查找种群中最优的个体
for i in range(len(population)):
if best_fit < _fit[i]:
best_fit = _fit[i] # 获得适应度最高的个体
best_individual_lis = population[i] # 传入染色体
best_individual = translation([best_individual_lis], chromosome_length)[0] * max_value / (pow(2,chromosome_length)-1) # 求出个体的具体数值
return [best_fit, best_individual]
def main(chromosome_length, population_size, max_value, Cross_rate, Muta_rate, max_iter, results):
populations = species_origin(population_size, chromosome_length) # 初始化种群
for i in range(max_iter):
_fit = fitness(populations, chromosome_length, max_value) # 计算适应度
results.append(bests(populations, chromosome_length, max_value, _fit)) # 计算最优的个体
selection(populations, _fit) # 进行选择
crossover(populations, chromosome_length, Cross_rate) # 进行交叉运算
mutation(populations, chromosome_length, Muta_rate) # 进行变异运算
results.sort()
X=[]
Y=[]
for i in range(max_iter):#500轮的结果
X.append(i)
Y.append(results[i][0])
plt.plot(X,Y)
if __name__ == "__main__":
chromosome_length = 20 # 染色体数量
population_size = 300 # 种群的规模
max_value = 10 # 限制基因中允许出现的最大值
Cross_rate = 0.8 # 进行双点交叉的概率
Muta_rate = 0.1 # 传入变异的概率
max_iter = 100 # 传入最大繁衍次数
results = [] # 传入每次繁衍的最优个体
main(chromosome_length, population_size, max_value, Cross_rate, Muta_rate, max_iter, results) # 运行主要的程序
