摘要
单细胞 RNA 测序能够以单细胞分辨率捕获基因活动,从而能够重建细胞类型特异性基因调控网络 (GRN)。用于重建 GRN 的可用算法通常是针对批量 RNA-seq 数据设计的,很少有算法适用于通过处理丢失事件和细胞异质性来分析 scRNA-seq 数据。在本文中,作者将基因对的联合基因表达分布表示为图像,并提出了一种称为 DeepDRIM 的新型监督深度神经网络,它利用目标 TF 基因对的图像和潜在邻居的图像来重建 GRN scRNA-seq 数据。由于考虑了 TF-基因对的邻域上下文,DeepDRIM 可以有效消除由传递性基因-基因相互作用引起的误报。作者将 DeepDRIM 与专为批量或单细胞 RNA-seq 数据设计的九种 GRN 重建算法进行了比较。它对从八个细胞系收集的 scRNA-seq 数据取得了明显更好的性能。模拟数据表明,DeepDRIM 对 dropout 率、细胞数量和训练数据大小具有鲁棒性。作者关注细胞类型特异性的 GRN 改变,并观察到差异化的 TF 靶点。
介绍
基因调控网络(GRN)的重建对于理解协同基因效应和特定背景转录动力学的机制至关重要。但是之前忽略了细胞异质性。单细胞 RNA 测序 (scRNA-seq) 提供了捕获细胞特异性基因表达的机会,从而可以更深入地了解细胞异质性和细胞类型特异性基因活动。
但是大多数用于 GRN 重建的可用算法都是为批量基因表达而设计的,并通过解决两个计算挑战来发挥作用。在这种情况下,如果采用 scRNA-seq 数据,则会出现独特的困难。首先,假定的 TF 基因相互作用是通过检查它们的共表达而得出的。大量基因表达数据通常归一化为标准高斯分布,以便可以通过互信息 (MI) 、皮尔逊相关系数 (PCC) 等方法量化 TF 基因相关性。由于转录本采样不平衡,scRNA-seq 基因表达数据为零。尽管可以在计算 TFgene 共表达之前插补零条目,但这可能会引入不可预测的噪声和偏差 ,因为大多数插补算法都利用基因-基因共表达。其次,由于传递相互作用(例如由一个或多个中间基因桥接的那些)而具有强共表达的 TF 基因对应该被消除。已经设计了几种策略来通过调节其他混杂基因来消除这些传递性相互作用;例子包括高斯图形模型、条件MI、基于上下文的归一化和边缘去除及基于树的集成方法。这些算法最初是为了分析大量基因表达数据而开发的,并不适合对 scRNA-seq 数据进行建模 。有人提出了一些算法重构。
SCODE , PIDC, SINCERITIES ,GENIE3。尽管这些专用策略旨在解决 scRNA-seq 数据中的固有问题,但它们都没有产生以细胞类型特异性 ChIP-seq 数据为基准的可接受的结果,有些甚至接近随机猜测 。
CNNC 是一个有监督的深度神经网络,它将基因对的联合表达表示为图像,并使用卷积神经网络(CNN)从 scRNA-seq 数据预测基因-基因共表达。我们注意到,CNNC 获得的大量误报集中在具有强 Pearson 相关性的基因对中(如图 )。
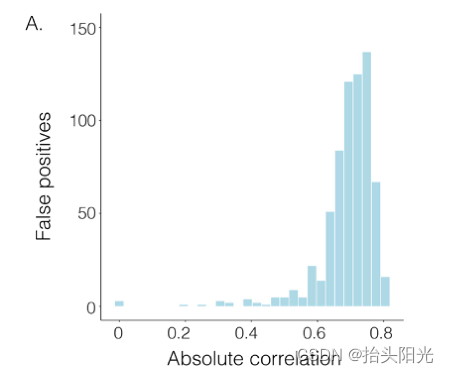
作者提出了 DeepDRIM(基于深度学习的直接调节交互模型),这是一种有监督的深度神经网络,可以通过考虑主图像和邻近图像,从 scRNA-seq 数据重建高度准确的细胞类型特异性 GRN。 DeepDRIM 的基本原理和工作流程如图2 所示。
结果
相邻图像在消除传递相互作用方面的有效性
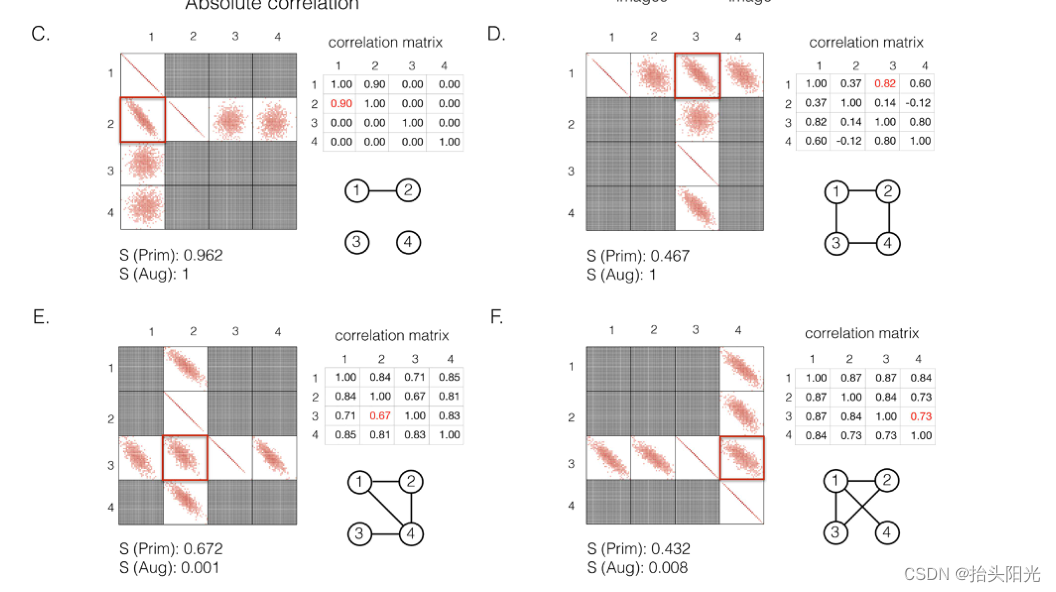
先生成模拟数据并尝试使用两种类型的输入来训练 CNNC,一种仅包含主图像,另一种包含增强图像。可以发现,当考虑模型中的邻近图像时,误报和由于传递交互造成的误报的总体比例显着下降了 40.4% 和 55.4%(图 1B)。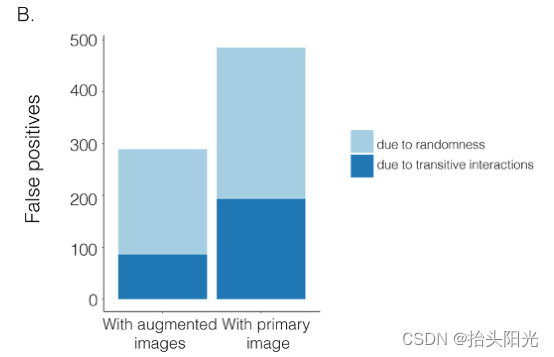
这一观察背后的基本原理可以被视为对主图像对其邻域进行“归一化”,以减轻对交互强度的高估。此外,图1C和D清楚地表明,考虑邻近图像不会削弱预测直接相互作用的能力(例如图1C中的基因1⇒基因2,图1D中的基因1⇒基因3)。在图1E中,基因2通过间接边基因2⇒基因4⇒基因3连接到基因3。此外,我们注意到{基因2,基因4}(|PCC| = 0.81)和{基因4的相关性, 基因 3} (|PCC| = 0.83) 强于目标 {基因 2, 基因 3} (|PCC| = 0.67),这提供了有明确证据表明{基因 2,基因 3} 应标记为误报。通过考虑邻近图像,模型将{基因 2,基因 3} 的预测置信度得分从 0.672 降低到 0.001,与图 1F 中观察到的类似情况相同。这些发现巩固了在 GRN 构建中考虑局部邻域的重要性,以消除由于传递相互作用而导致的误报。
DeepDRIM 概述
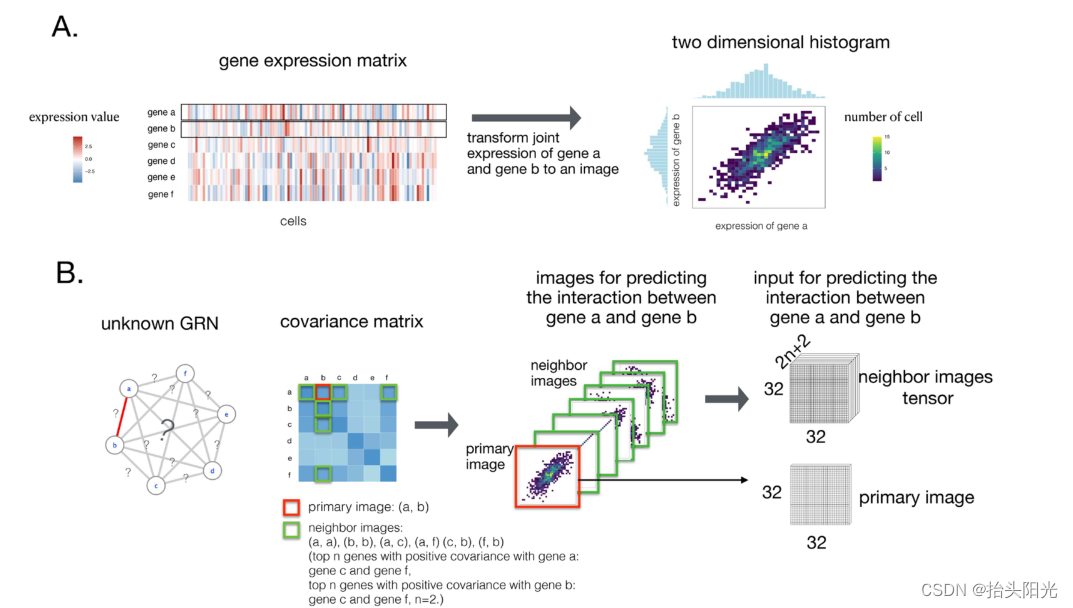
DeepDRIM 被提出从 scRNA-seq 数据重建细胞类型特异性 GRN,具有高精度和低假阳性率。图 2 说明了如何使用 DeepDRIM 来预测基因 a 和基因 b 之间的相互作用。首先,DeepDRIM将基因a和基因b的联合基因表达转化为a,具有 32 x 32 个箱的二维直方图(主图像,图 2A),其中每个箱的强度是指落入其中的细胞数量。其次,DeepDRIM 构建 2n + 2 个邻近图像,其中涉及 n 个基因的 2n 个图像与基因 a (a, i) 或基因 b (b, j) 具有顶部正协方差,并且这两个图像代表自身图像 ( a、a)和(b、b)。
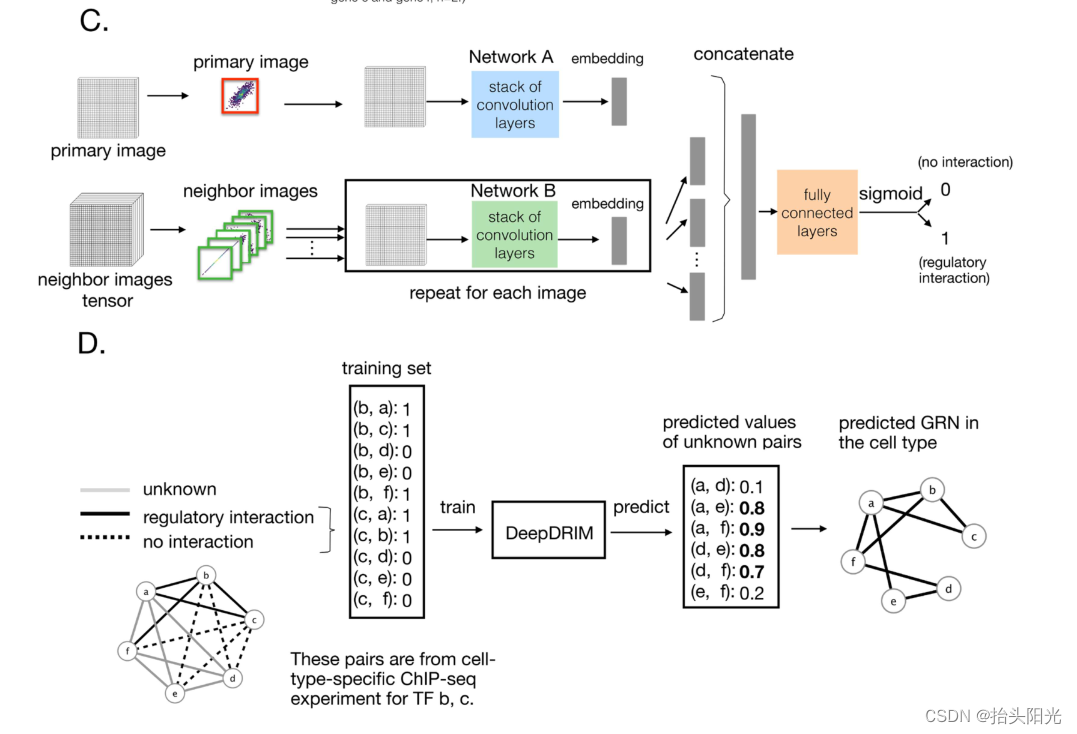
这些邻近图像被提供给模型以捕获主图像的邻近上下文(图 2B),这提供了区分直接交互和传递交互所需的关键信息。我们将相邻图像组织为张量而不是增强图像,以在实际数据上获得更好的性能(补充图 S2)。第三,两个 CNN 分别用于处理主图像(网络 A)和邻近图像张量(32 x 32 x 2n+2)(网络 B)(图 2C,方法和补充图 S3)。网络A遵循VGGnet[32],与CNNC类似。网络 B 是一个类似暹罗的神经网络,设计用于处理多个相邻图像。神经网络通过已知的 TF 基因相互作用进行训练,该相互作用取自公开可用的细胞类型特异性 ChIP-seq 数据。最后,通过具有置信度分数(0 到 1 之间,图 2D)的有向边来预测未知的交互作用。
DeepDRIM 优于重建细胞类型特定 GRN 的现有算法
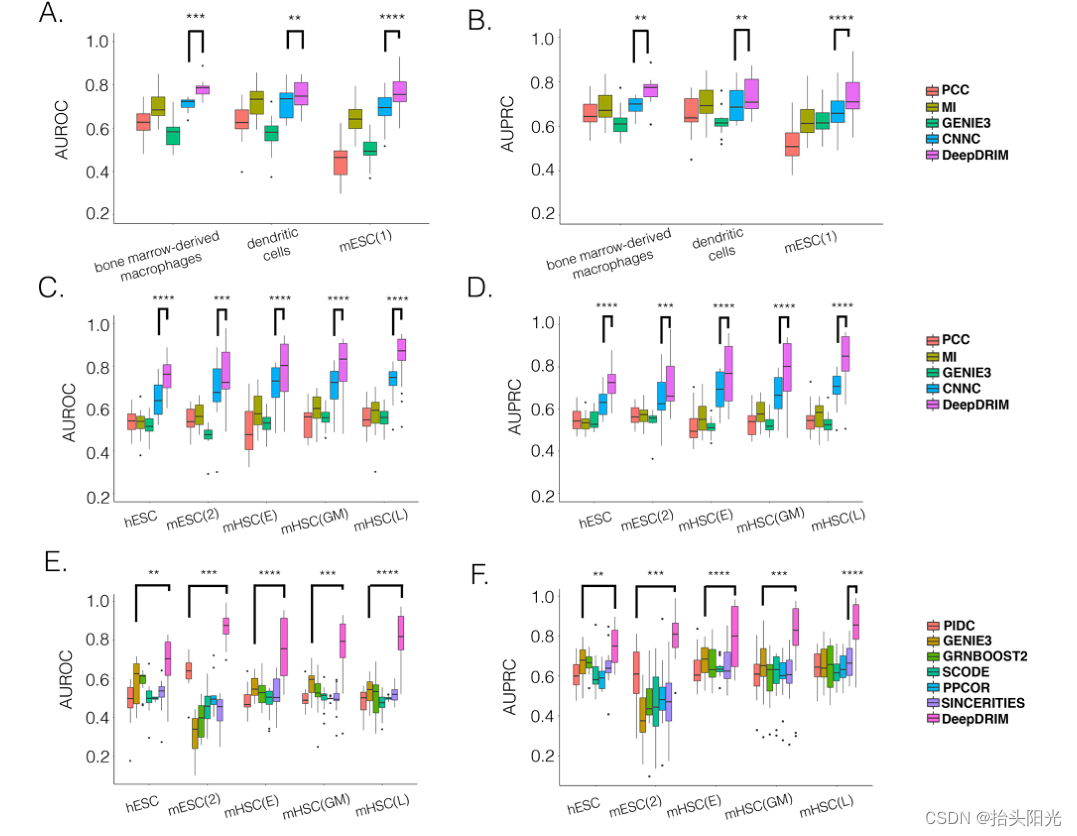
本文从两个来源收集了来自八个细胞系的 scRNA-seq 数据集及其相应的 ChIP-seq 数据 ,以使用 TF- 将 DeepDRIM 与现有方法(表 1)进行比较意识到三倍交叉验证(方法)。我们首先使用 PCC、MI、CNNC 和 GENIE3 评估 DeepDRIM; GENIE3 是在 scRNA-seq 和大量基因表达数据 上重建 GRN 的最佳算法之一。
DeepDRIM 对 scRNA-seq 数据的质量和训练集的大小具有鲁棒性
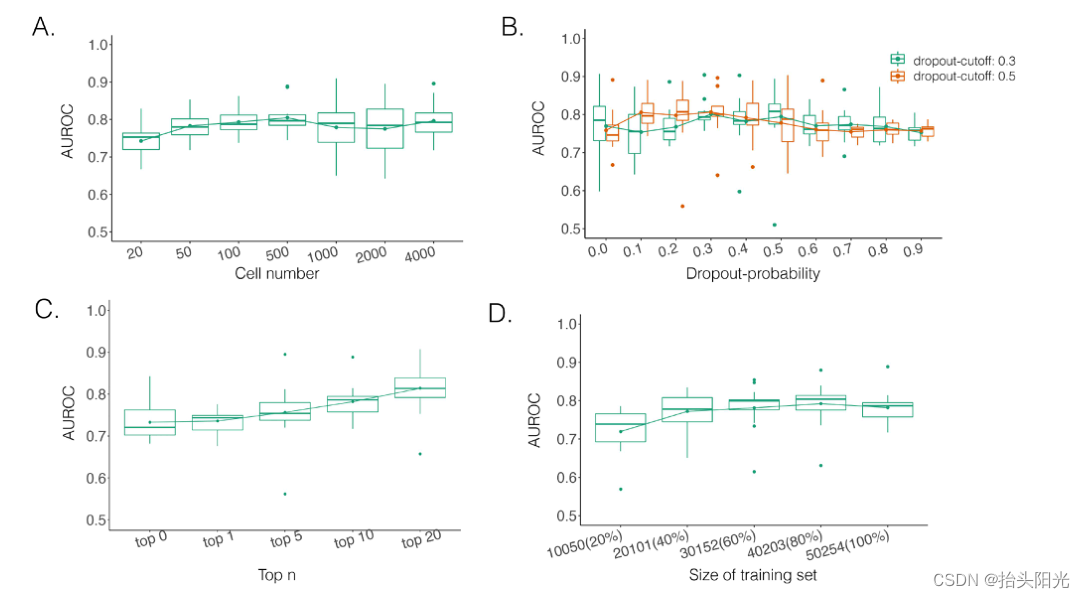
DeepDRIM 的性能可能受到 scRNA-seq 数据质量(丢失率和细胞数量)、涉及的邻近图像数量和训练集大小的影响。为了评估 DeepDRIM 对这些因素的鲁棒性,本文首先选择来自骨髓源性巨噬细胞的 scRNA-seq 数据作为模板,并使用一系列参数模拟一系列 scRNAseq 数据。通过对所涉及的细胞数量(从 20 到 4000 个细胞)进行二次采样,生成了 7 个 scRNAseq 基因表达数据集,这反过来又改变了主图像和邻近图像的分辨率。作者发现当细胞数量大于 100 时,DeepDRIM 对低分辨率图像具有鲁棒性(图 4A)。接下来,作者使用 MAGIC 估算模板中的 dropout,然后将条目随机屏蔽为具有一系列 dropout 率的 dropout(方法)。DeepDRIM在不同的压差配置中表现出稳定的能。第三,作者通过改变输入到模型中的相邻图像的数量来比较 DeepDRIM 的性能。结果,作者发现涉及的相邻图像越多,DeepDRIM 的性能就越好(图 4C)。实际上,涉及更多图像的计算成本会更高。在本文的研究中,作者选择了与目标 TF 或基因正协方差最强的前 10 个基因,因此总共涉及 22 个相邻图像(如果未指定)来平衡这两个因素。此外,为了评估训练集大小的影响,本文对基准 TF 基因对的 20%、40%、60%、80% 和 100% 进行了二次采样以进行训练。本文的结果表明,训练集的大小并没有显着影响 DeepDRIM 的性能(图 4D),并且当应用 40% 的训练集(包括 20 101 个 TF 基因对)时几乎达到稳定水平。
结论
DeepDRIM,一种有监督的深度神经网络模型,用于根据 scRNA-seq 数据预测 GRN。 DeepDRIM 将 TF-基因对的联合表达转换为主图像,并将相邻图像视为主图像的邻域上下文,以消除由于传递交互而导致的误报。 DeepDRIM 还利用训练集来捕获 CNN 嵌入中的关键区域,这些区域可以识别 TF 基因的相互作用和因果关系。我们的研究结果表明,DeepDRIM 在测试的八种细胞类型上优于九种现有算法,并且对 scRNA-seq 数据的质量具有稳健性。
方法
1 基因对联合表达的表示
2 DeepDRIM的网络结构
3 模拟 scRNA-seq 数据以检查邻近图像的影响
4 来自八个细胞系的 scRNA-seq 数据
5 DeepDRIM 与现有 GRN 重建算法的比较
6 模拟 scRNA-seq 数据以评估稳健性
DeepDRIM is available at https://github.com/jiaxchen2-c/DeepDRIM.