来自Manolis Kellis教授(MIT计算生物学主任)的课《人工智能与机器学习》
主要内容就是调控基因组学和深度卷积网络的结合
由于这部分在我学习的课程中内容很少,下面贴出油管链接(这个每节课一个半小时):
教授详细的课1:Deep Learning for Regulatory Genomics - Regulator binding, Transcription Factors TFs
教授详细的课2:Regulatory Genomics - Deep Learning in Life Sciences - Lecture 07 (Spring 2021)
Regulatory Genomics
Biological motivation of Deep CNN
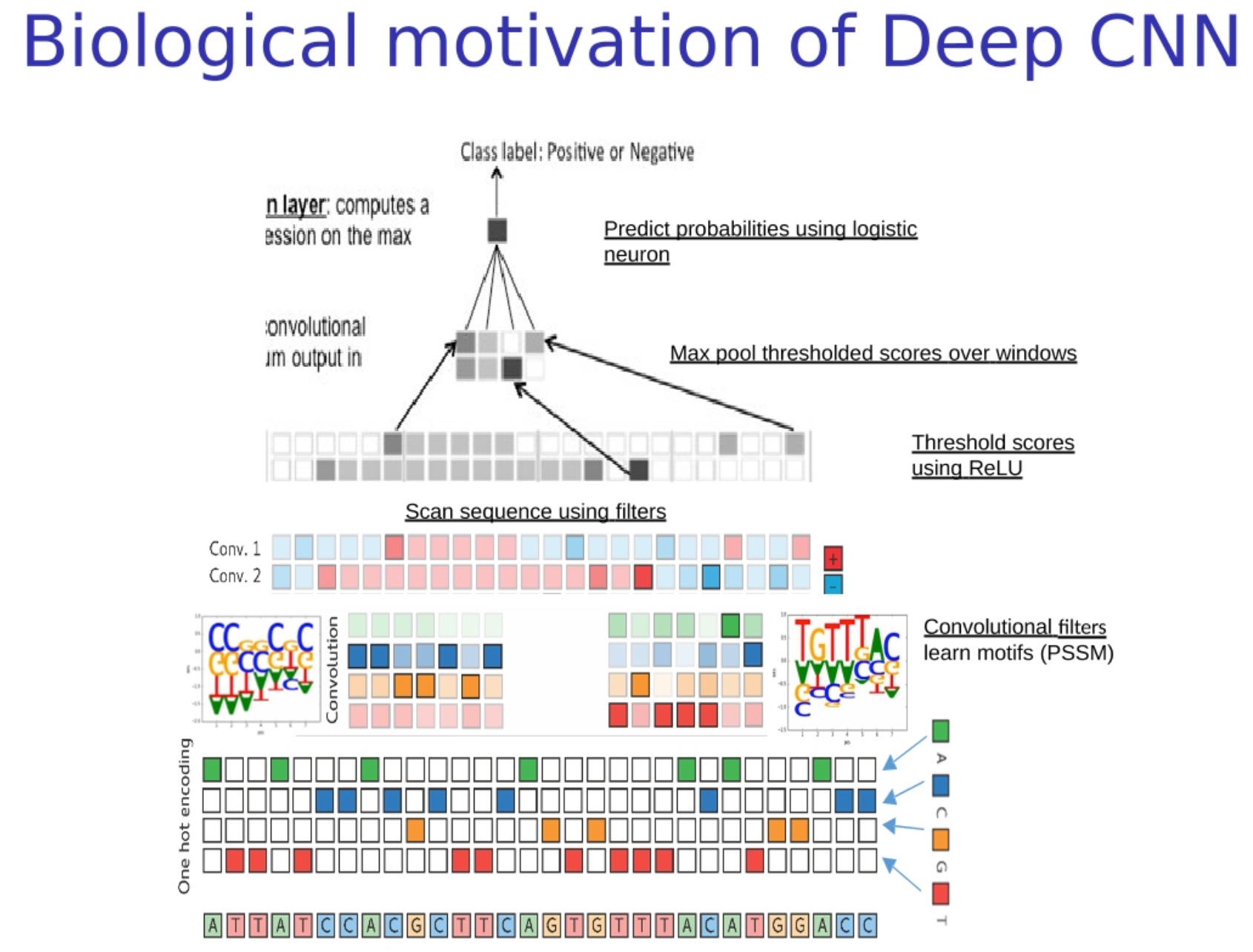
深度序列模型"或"深度位点特异性模型"的特殊类型的深度卷积神经网络(Deep Convolutional Neural Networks,简称 Deep CNNs),它在处理生物序列(如DNA,RNA或蛋白质序列)时,将滤波器初始化为位点特异性评分矩阵(Position-Specific Scoring Matrix,简称PSSM)或其他有生物学意义的模式(motifs)。这种模型的优点是可以利用已有的生物学知识来指导模型的学习,从而加速训练过程,提高模型的准确性。
如果你不会CNN的话,可以看这个快速入门一下:CNN(卷积神经网络)、RNN(循环神经网络)、DNN(深度神经网络)的内部网络结构有什么区别
以下是这种模型的工作流程:
- 将生物序列转化为数值型的表示:和之前一样,我们需要将生物序列(如DNA)转化为数值型的表示,常用的方式是独热编码(one-hot encoding)。
- 使用生物学意义的滤波器扫描序列:在这个步骤,我们首先将卷积层的滤波器初始化为PSSM或其他有生物学意义的模式(motifs)。这些滤波器会在序列上滑动,根据每个滤波器对应的模式,计算序列在各个位置的匹配程度。(与边缘检测不同的是,这里使用的卷积核(滤波器)是有生物意义的motifs)
- 通过ReLU进行阈值化:ReLU(Rectified Linear Unit)是一种常见的激活函数,它对负数输出0,对正数保持不变。这个操作可以增加模型的非线性,使得模型可以学习更复杂的模式。
- 最大池化(Max Pooling):这是一种减小序列长度,同时保留关键信息的操作。在卷积神经网络中,池化层通常跟在卷积层后面,用于降低特征的维度和控制过拟合。
- 使用逻辑回归预测概率:在所有处理步骤之后,我们可以用一个逻辑回归层(通常是一个全连接层,加上一个sigmoid激活函数)来预测类别。
值得注意的是,虽然我们在开始时将滤波器初始化为PSSM或其他有生物学意义的模式,但在训练过程中,滤波器的参数会被进一步调整,以更好地适应训练数据。这就使得我们既可以利用已有的生物学知识,又可以从数据中学习到新的知识。
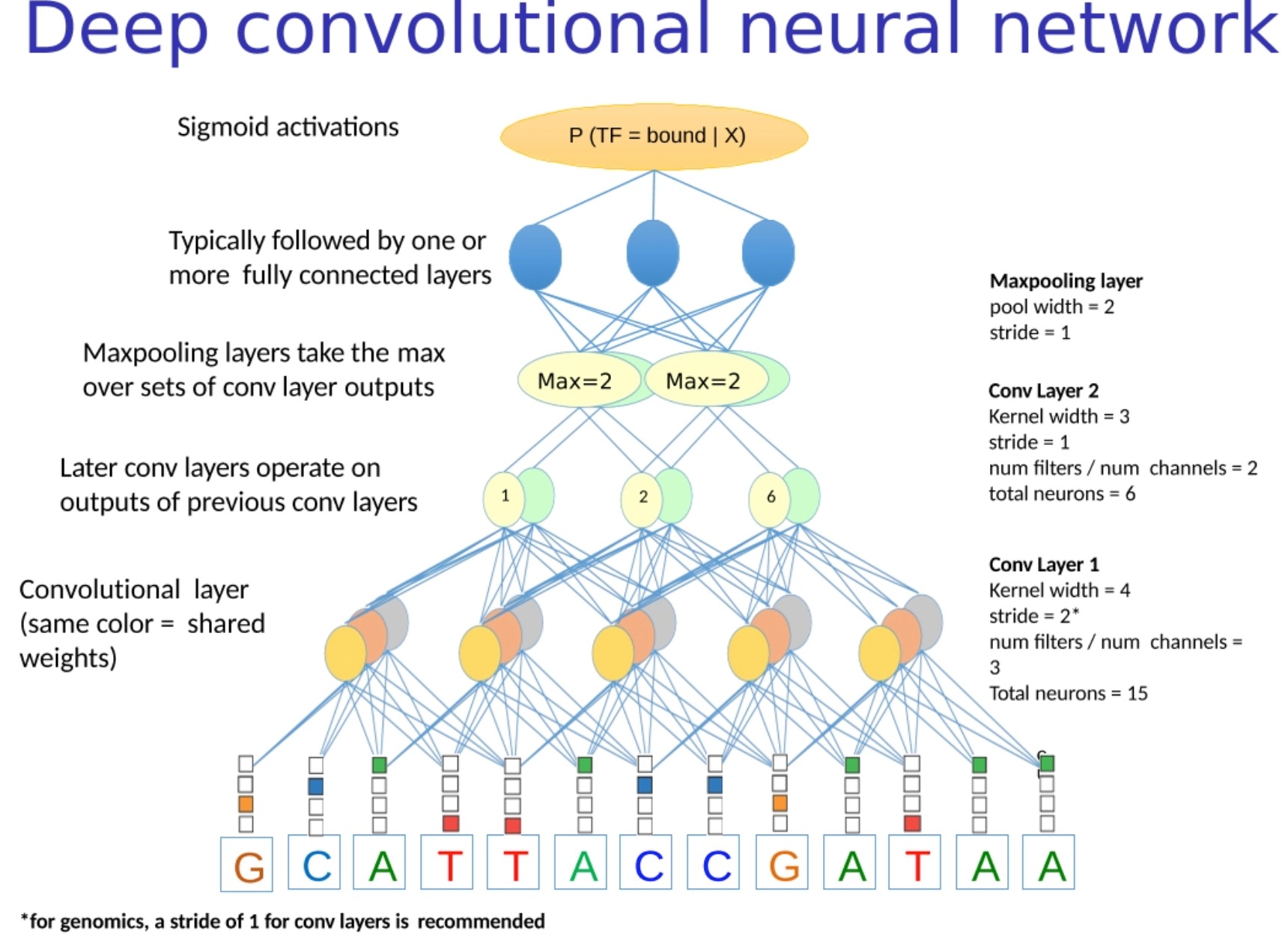
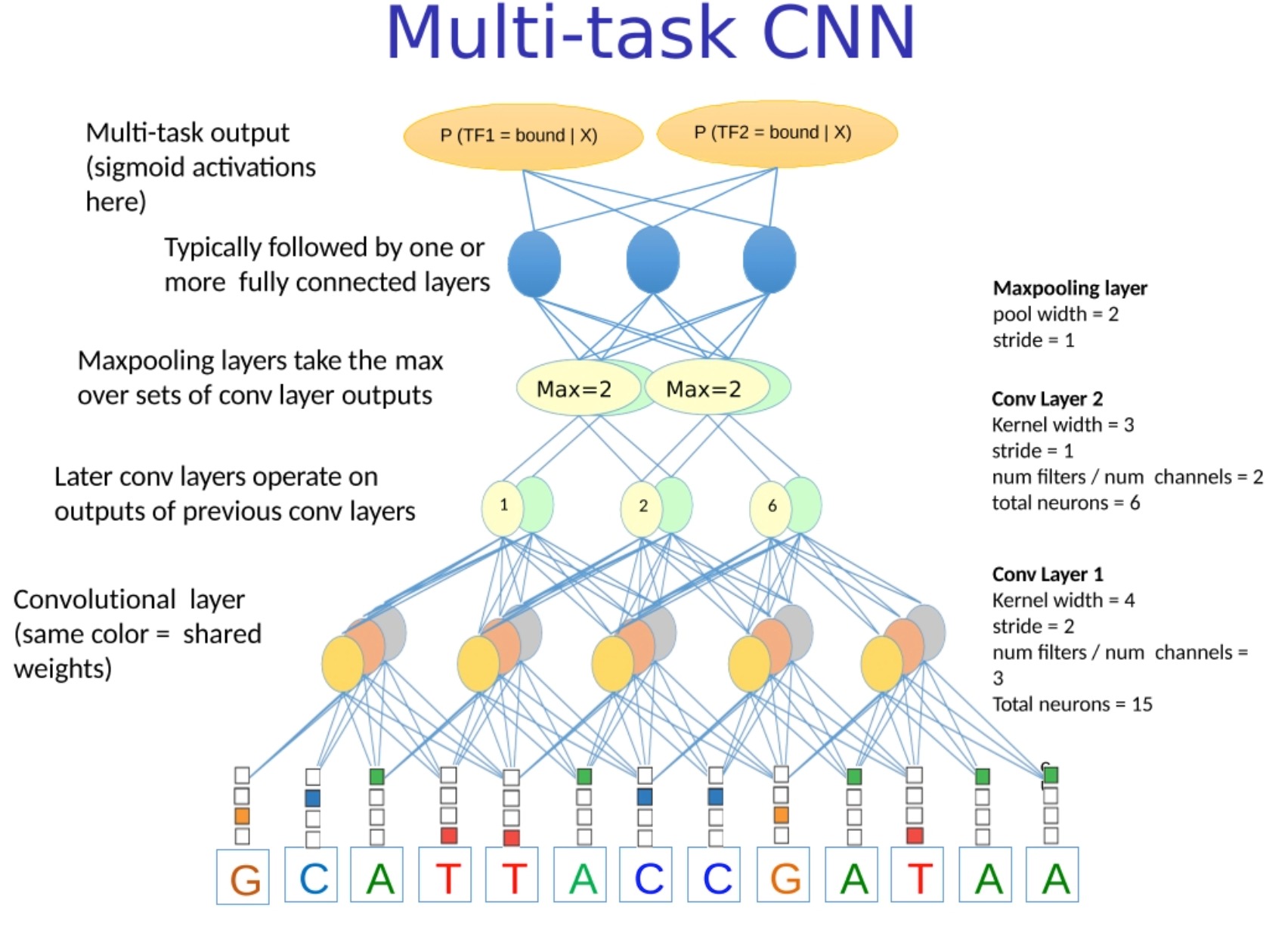
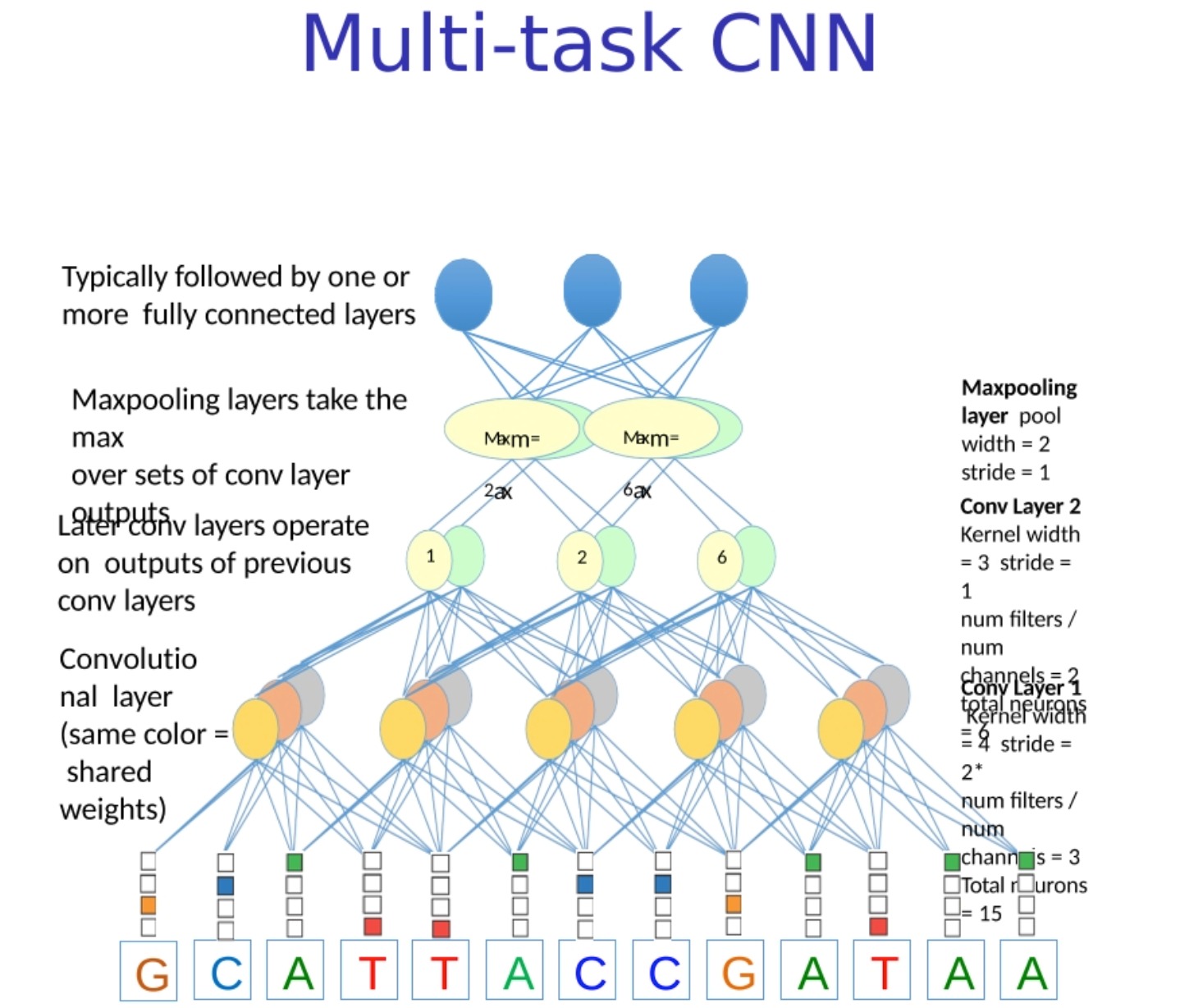
Multi-task CNN