高通量测序时代,微生物组研究中常用扩增子测序及宏基因组测序技术研究微生物群落的多样性、结构和功能。大量的数据信息处理及可视化成为微生物组研究的迫切需求,而用于分析的R语言包软件纷繁复杂,功能类似,难以抉择,给许多研究人员探索微生物组数据带来了重大挑战。
南京农业大学袁军组&中国农业科学院刘永鑫组共同汇总介绍了基于R语言的324个常用R包的数据挖掘过程,根据微生物组研究的6大功能类别对这些R包进行了功能分类,不仅总结了微生物组数据分析中的常见内容,还对常用的集成R包的优点及局限性进行了细致介绍,提出了最适合微生物组数据挖掘的分析流程。
相关代码可获取自:
https://github.com/taowenmicro/EasyMicrobiomeR 大家可随时取用哦!
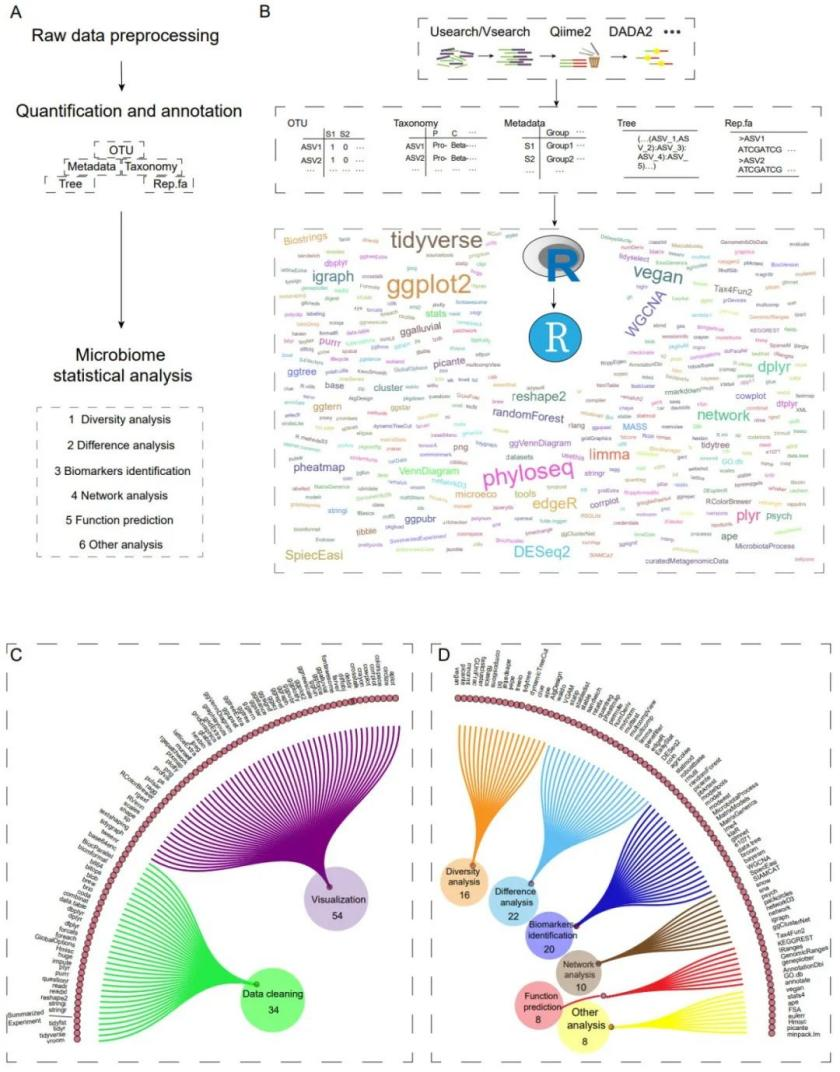
在该综述中,作者首先介绍了基于扩增子测序的微生物群落数据分析工作流程(A图)。其核心文件是OTU聚类注释信息,包括OTU表、分类表、样品元数据(Metadata)、系统发育树(Tree)和代表序列(Rep.fa)。首先,原始数据可以通过使用USEARCH/VSEARCH、QIIME2、DADA2软件包进行处理。然后,将重要文件保存,用于RStudio软件下的R语言环境开展下游分析。许多微生物分析方法都依赖于R语言开发的R包。
然而,下游分析R包的数量已经达到了令人眼花缭乱的水平。下图词云图中的字体大小代表R包的引用次数(B图)。
文章梳理了常用于数据预处理和可视化的常用的R包共计88项(C图),以及微生物群落分析的六大类分析的R包(D图 )(包括多样性分析、差异性分析、标志物识别、相关性及网络分析、功能预测及其他相关分析等),并针对不同的研究内容对应软件进行了详尽的优势介绍。

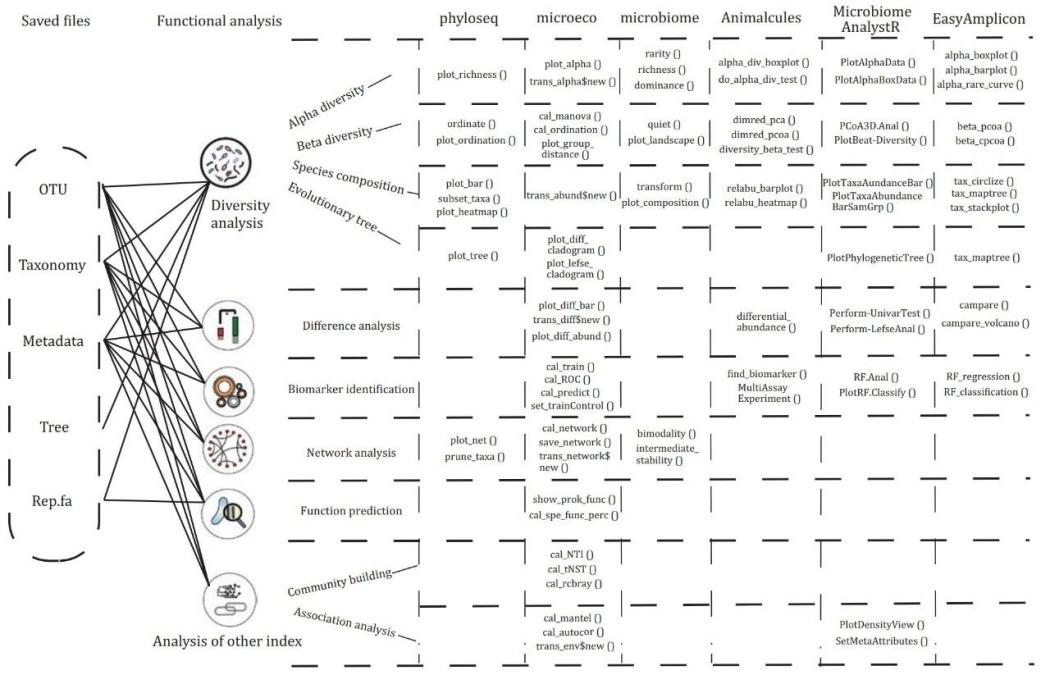
此外,专用于微生物数据处理的R包也被纳入介绍体系,作者将常用的六大种微生物组分析集成R包进行了详尽介绍,系统梳理了这六个常用分析集成包的功能。包括:
-
Phyloseq包
-
Microbiome包
-
MicrobiomeAnalystR包
-
Microeco包(强烈推荐!)
-
amplicon包
由于过多的R包可能会阻碍微生物组研究人员进行有效地选择。因此,作者在六种分析中选择了高效、常用、用户友好的函数包,涵盖了微生物研究中的多项分析内容:1) 多样性分析,2) 差异分析,3) 生物标志物识别,4) 相关性和网络分析,5) 功能预测,6) 其他微生物组分析。新的R函数整合总结了微生物组中大部分常见的分析内容,形成了最适合微生物组分析的路径,实践成果实例如下图:

使用适当的数据结构可以加速微生物组数据分析。对R语言包的开发和集成包的构建不断推进了微生物组研究的发展和数据挖掘的深入。这篇综述系统树立了R语言包的功能和优势,针对冗余的功能软件进行了系统性评价,避免重复使用相同部分或类似内容,突出了R包的优势,更有利于数据挖掘、机器建模分析,为今后开发更好的微生物组工具提供了重要的理论依据和实践参考。