数据降维
概念:在尽量减少信息量的前提下,采用某种映射方法(函数)
把原来的高维数据(变量多)---映射--->低维数据(变量少)
避免维数灾难 :增加样本量
常用的降维方法:
线性方法 非线性方法
有监督方法 --> LDA(线性判别分析) 无
无监督方法 --> PCA(主成分分析) 局部线性嵌入(LLE)拉普拉斯特征映射
一、主成分分析(PCA)原理(无监督)
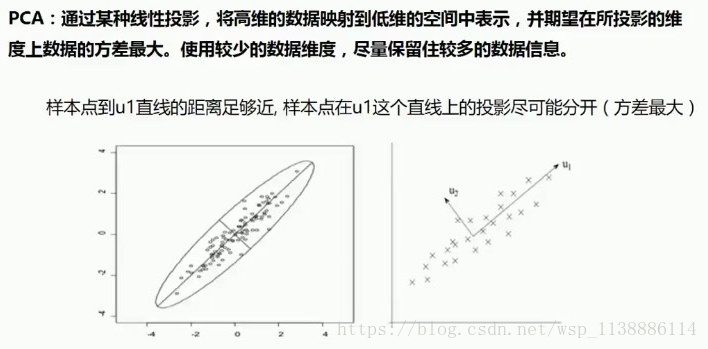
理解 PCA 的关键,一是坐标变换,二是新坐标(也就是投影)
1.1 通过线性投影
1.2 主成分分析(PCA)操作流程
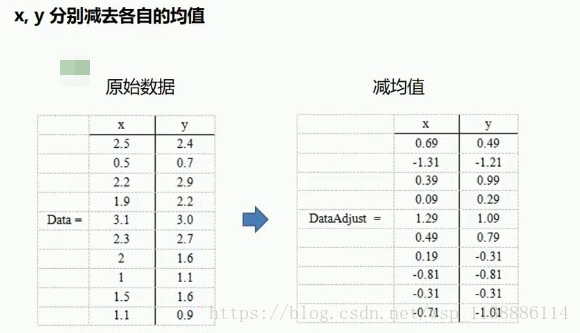
A 原始数据—减均值
B 求特征变量的协方差矩阵

C 求协方差的 特征值 和 特征向量
D 将特征值大->小排序,选择最大的k(1)个,然后对k(1)个特征向量分别作为 列向量组成特征向量矩阵(最大的特征根对应的特征向量)
E 将样本点投影到选取的向量上,得到最终降维后的新维度(1个)代码: from IPython.core.interactiveshell import InteractiveShell InteractiveShell.ast_node_interactivity = 'all' from sklearn import datesets iris= datasets.load_iris() #导入iris数据 X = iris.data y = iris.target X[:10] y[:10] #PCA 降维 from sklearn.decomposition import PCA # sklearn.decomposition.PCA(n_components=None,copy = True whiten = False) # n_components:主成分个数 # copy:是否在运行算法时,将原始数据复制一份,缺省时默认 Ture # whiten:白化,使得每个特征具有相同的方差,缺省时默认 False PCA = PCA(n_components = 3) #定义一个PCA模型 pca.fit(X) #fit X_new = pca.transform(X) #transform X_new[:5] X_new = pca.fit_transform(X) #fit_transform --可以替代fit 和 transform(X) X_new[:5] #主成分 解释方差占比 print pca.explained_variance_ratio_ print pca.explained_variance_ #PCA 降维后可视化 pca = PCA(n_components = 2) pca.fit(X) X_new = pca.transform(X) import matplotlib.pyplot as plt %matplotlib inline plt.scatter(X_new[:,0],X_new[:,1],mark = 'o',c = y) plt.show()
二、线性判别分析(LDA)原理(有监督)
LDA 是一种 监督学习 的线性降维技术,与PCA最大的区别,它需要一个目标类别变量
LDA 思想:投影后类内方差最小,类间方差最大。--能最好的把目标变量的类别区分开 LDA降维后得到的新维度可以继续作用目标变量分类预测的特征
PCA 与 LDA对比
PCA 投影后的目的:整体方差最大(不关心目标变量各类别的区隔,强调整体方差最大化 即显示所有数据) LDA ----------:类内方差最小,类间方差最大(目标变量各类别区隔明显,强调局部) PCA 与 LDA总结 如果研究问题有目标变量(类别型) 优先使用LDA 降维 可以先使用PCA做小幅度的降维,消去噪声,然后再使用LDA降维 如果研究的问题没有目标变量 优先使用PCA降维代码:iris数据集 iris = datasets.load_iris() #导入iris数据 X = iris.data y = iris.target X[:10] y[:10] LDA 降维 from sklearn.lda import LDA lda = LDA(n_components=2) #定义一个LDA模型 X_new = lda.fit_transform(X,y) #fit_transform --可以替代fit 和 transform(X) X_new[:5] lda.predict(X) #predict(X) lda.score(X,y) #score 对比 PCA 与LDA: from sklearn.decomposition import PCA #PCA降维后作图 pca = PCA(n_components = 2) pca.fit(X) X_new = pca.transform(X) import matplotlib.pyplot as plt %matplotlib inline plt.scatter(X_new[:,0],X_new[:,1],mark = 'o',c = y) plt.show() from sklearn.decomposition import LDA LDA降维后作图 lda = PCA(n_components = 2) lda.fit(X,y) X_new = lda.transform(X) import matplotlib.pyplot as plt %matplotlib inline plt.scatter(X_new[:,0],X_new[:,1],mark = 'o',c = y) plt.show()