
题目:Latent generative landscapes as maps of functional diversity in protein sequence space
文献来源:Nature Communications | (2023) 14:2222
代码:https://github.com/morcoslab/LGLVAE/
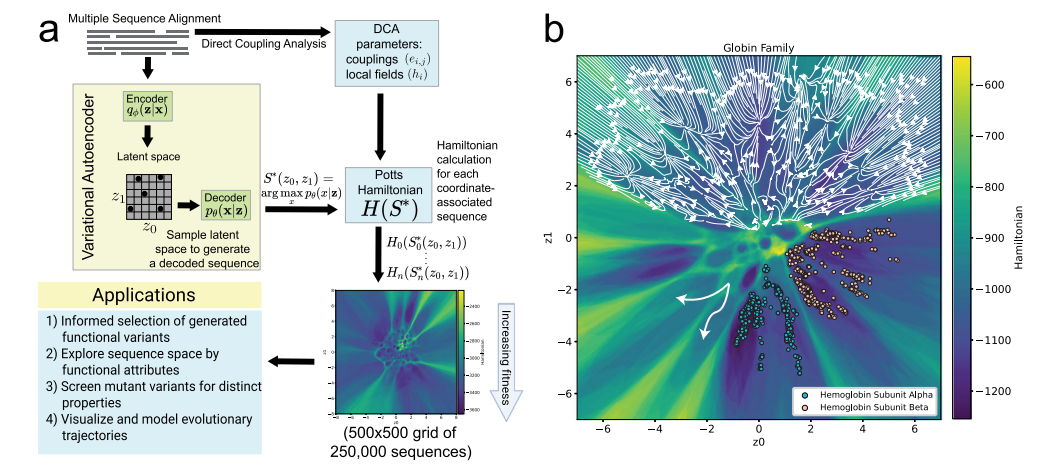
简介:变分自编码器是一种具有生成能力的无监督学习模型,当应用于蛋白质数据时,它们通过系统发育学对序列进行分类,并生成保留蛋白质组成统计特性的从头序列。虽然以前的研究集中在聚类和生成特征上,但在这里,作者评估了嵌入序列信息的潜在流形。为了研究潜在流形的性质,他们利用直接耦合分析和波茨哈密顿模型来构建一个潜在的生成景观。本文展示了这一景观如何捕获多个系统的系统发育分组、功能和适应度特性,包括球蛋白、β-内酰胺酶、离子通道和转录因子。作者为景观如何帮助理解在实验数据中观察到的序列变异性的影响提供了支持,并为定向的和自然的蛋白质进化提供了见解。我们提出,结合变分自编码器的生成特性和功能预测能力,以及协同进化分析,可能有利于蛋白质工程和设计的应用。
主要内容:
-------------------------------------------
欢迎点赞收藏转发!
下次见!
扫描二维码关注公众号,回复:
15024749 查看本文章

