介绍MNE-Python系列文章,持续更新中…
0. 什么是MNE ?
开门见山地说,MNE是我用过的最强生理信号分析神器(可能是我见识浅薄,大佬勿喷),处理范围涵盖EEG、MEG等各种类型。
MNE本质上就是一个开源的Python第三方库/模块,我认为 MNE官网 首页的介绍就是对MNE最准确的概述,如图:

1. 文章思路
因为MNE的功能实在是很多,基本包括了读取数据集、预处理、分类、可视化等完整流程,所以我不可能全部介绍到。本着授之以鱼不如授之以渔的原则,我决定该系列的第一篇文章思路是:详细介绍一个简单样例demo,展示使用MNE的大致流程。
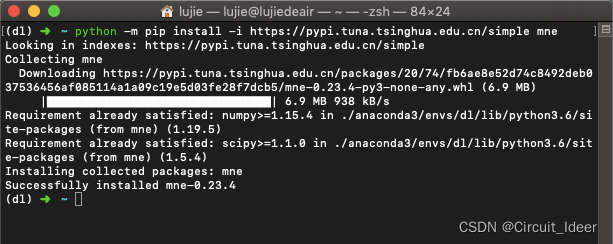
2. MNE安装
与其他所有的Python第三方库/模块一样,使用 pip install mne 安装即可,不再过多赘述(这里我使用了清华镜像源):
3. MNE 代码实战
先贴一段MNE的代码,如果你之前不了解MNE的话此刻应该比较懵圈,问题不大,接下来我带你逐行分析一下全部代码,Let’s Go ! :
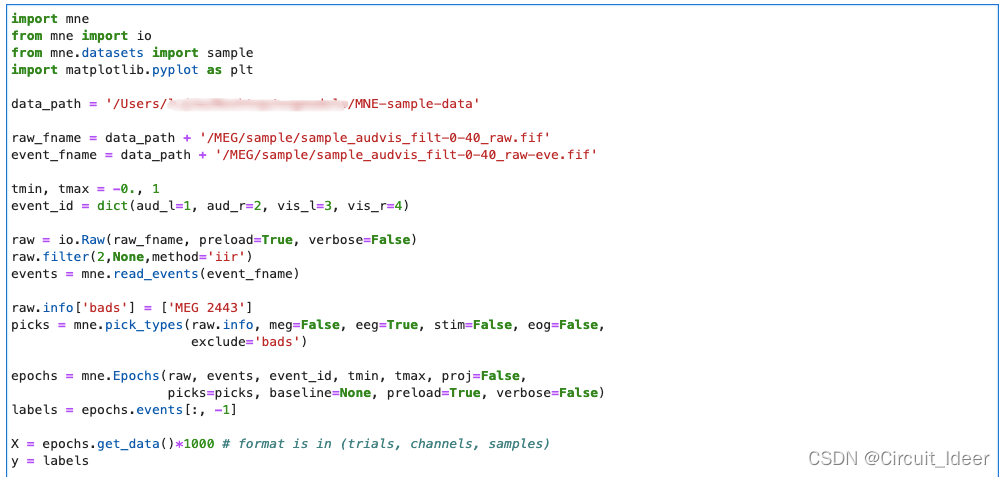
导入MNE相关包
import mne
from mne import io
from mne.datasets import sample
# 数据可视化包
import matplotlib.pyplot as plt
导入数据集
导入数据集有两种方式:
1) 远程下载数据集
在上一步中,我们已经使用 from mne.datasets import sample 导入了sample数据集,注意:这里 sample 是一个数据集的名字!(关于此数据集的介绍放在文末)
然后我们就可以使用data_path() 获取这个数据集里的全部数据:
data_path = sample.data_path()
2) 本地已下载好数据集
# 自定义数据集存放位置
data_path = '/Users/xxx/xxx/MNE-sample-data'
创建数据对象
MNE的数据结构其中之一为raw数据结构,通过以下方式创建raw数据对象:
# 设置相关参数
raw_fname = data_path + '/MEG/sample/sample_audvis_filt-0-40_raw.fif'
event_fname = data_path + '/MEG/sample/sample_audvis_filt-0-40_raw-eve.fif'
tmin, tmax = -0., 1
event_id = dict(aud_l=1, aud_r=2, vis_l=3, vis_r=4)
# 使用1):preload=False,使用2):preload=True
raw = io.Raw(raw_fname, preload=True, verbose=False)
我们通过 print(raw) ,可以发现其核心数据为 n_channels(数据维度)和 times(数据时长):

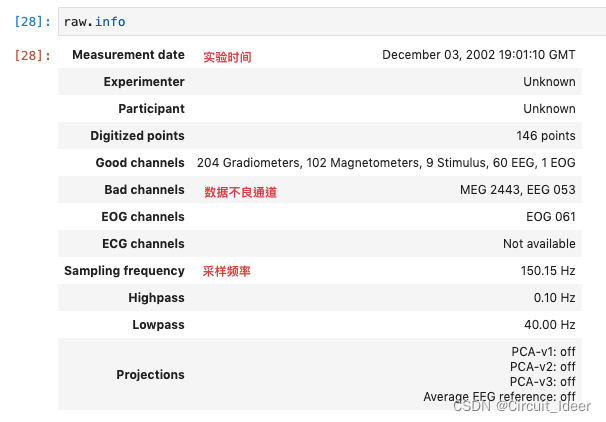
还可以通过 raw.info 查看raw数据的具体信息:
raw.filter(2, None, method='iir') # 使用iir滤波器进行滤波
events = mne.read_events(event_fname)
raw.info['bads'] = ['MEG 2443'] # 设定不良通道
# 通过种类和名字挑选通道
picks = mne.pick_types(raw.info, meg=False, eeg=True, stim=False, eog=False,
exclude='bads')
创建 epochs
将数据切分为 epoch 可以方便神经网络进行处理,在MNE中有一个Epochs类,我们能直接通过该类的方法直接从raw数据对象中提取Epochs:
# 创建Epochs类的对象,传入之前设置的参数,(tmin,tmax)为epochs中每个trial的时间长度
epochs = mne.Epochs(raw, events, event_id, tmin, tmax, proj=False,
picks=picks, baseline=None, preload=True, verbose=False)
labels = epochs.events[:, -1]
提取数据
X = epochs.get_data()*1000
Y = labels
get_data() 返回数据的格式为 (trials, channels, samples),通过 print(X.shape) 输出为(278,61,150):
分别与277.7s(数据时长)/1s(每个trial的时间长度)、通道数(60EEG+1EOG)、采样频率(150Hz)对应。
数据可视化
MNE集成了许多现成的功能函数,只需要简单的一行代码就可以实现许多复杂的功能,来点图片感受一下:
# 分析0-1s内的数据
import matplotlib.pyplot as plt
raw.info['bads'] = ['MEG 2443']
picks = mne.pick_types(raw.info, meg=False, eeg=True, stim=False, eog=False,
exclude='bads')
t_idx = raw.time_as_index([-0., 1.])
data, times = raw[picks, t_idx[0]:t_idx[1]]
plt.plot(times,data.T)
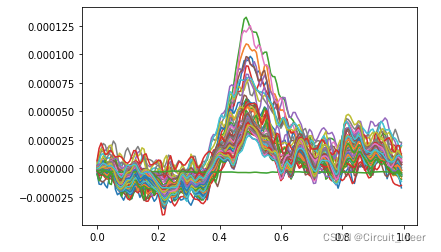
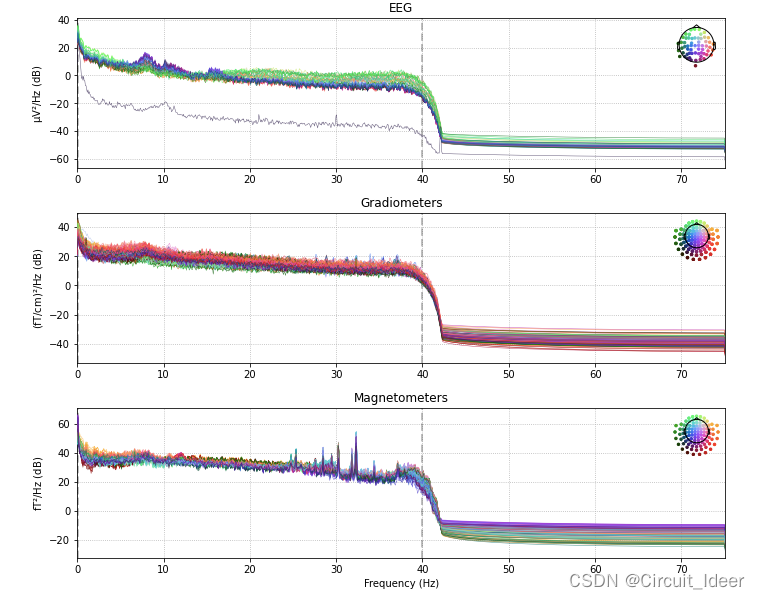
# 绘制各通道的功率谱密度
raw.plot_psd()
plt.show()
# 绘制SSP矢量图
raw.plot_projs_topomap()
plt.show()

4. Sample数据集介绍
数据采集
该数据集是通过位于MGH/HMS/MIT(麻省总医院)的Athinoula A. Martino生物医学成像中心的Neuromag Vectorview系统获得的。同时采集60通道电极帽的MEG(脑磁图)数据。原始MRI(核磁共振)数据集是通过使用MPRAGE序列的西门子1.5 T Sonata扫描仪获取的。
实验设置
在实验中,受试者的左右视野中会出现棋盘图案,同时会伴随出现在左右耳的音调,刺激间隔为750 ms。此外,在受试者的视野中心会随机出现笑脸图案,受试者被要求在笑脸出现后尽快用右手食指按下按键。实验中刺激和响应的对应关系如下:
数据集内容
Sample数据集主要包含两个部分:MEG/sample (MEG/EEG数据)和来自另一位受试者的MRI重建数据 subjects/sample ,我们使用的主要前者,其目录如下:
结束语
Ok,到这里一个MNE的小demo就已经解读完了,或许你已经感受到了MNE的强大和简洁,我会在之后的文章中陆续整理MNE中的各种类和函数,感兴趣的话欢迎点个关注再走呀 ^ _ ^ !!



