导读
岁岁年年花相似,细细推敲,实则年年岁岁花不同。人类进化历程中,万事万物都在悄然的变化着,这积沙成塔的量到质的跳跃,正是无数科研人员孜孜以求的方向–群体进化。
群体进化研究是指通过获得某物种自然群体各亚群的SNP、InDel等变异信息。然后基于群体变异信息,解析群体的遗传多样性、遗传结构、基因交流情况、物种形成机制以及群体进化动态等生物学问题,从分子层面深入研究该物种的进化历程。
而其中比较重要的一趴就是进化树,今天我们就先从群体进化开局–进化树开聊。
一、何为进化树?
系统树(Phylogenetic Tree)或者进化树(Evolutionary Tree)是描述基因、个体、种群、物种之间系统发生关系假说的树状结构,其核心要素是拓扑结构和分支长度。
进化树的形式
根据拓扑结构展现形式的不同,进化树的常见形式有“有根树”和“无根树”。
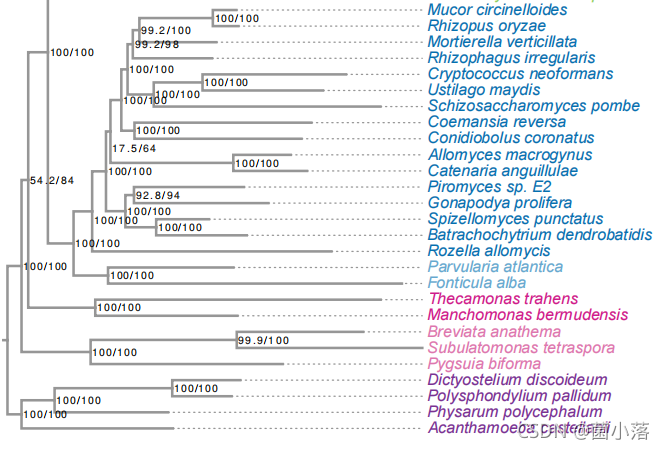
经典有根树
有根树:有一个特殊的根节点,表示所有进化枝的共同祖先(一般是假设原始祖先),从根节点只有唯一路径经进化到达其他任何节点,即有方向性。
辐射状进化树
无根树圈状进化树(含外群)
无根树:只表明了节点之间的关系,没有进化方向,其中线段的两个演化方向都有可能,通过引入外群(Outgroup)可在无根树中指派根节点。
2、具体解读进化树
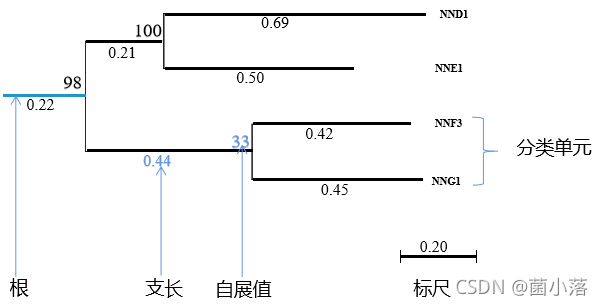
再来顺顺几个关键名词:
外群:即与分析序列相关的生物序列,但是具有较远的亲缘关系;
距离标尺:生物或序列见差异数值的单位长度,相当于进化树的比例尺。
进化分支长度:代表进化分支变化的程度,越短代表差异越小,进化距离越近。如上图NNE1与NND1的遗传变异度为0.69+0.50=1.19。变异度的计算方法很简单,即变异度=变异碱基数/总碱基数(%),这样看是不是很容易理解?
Bootstrap检验,也叫自展值,是一种放回式抽样统计方法,具体是对数据集有放回的多次重复抽样,构建多个进化树,用来检查给定树的分支可信度,一般抽样次数>1000。虽然根据严格的统计学概念,自展值需要大于95%才较为可信。而在实际应用中,一般任务节点的Bootstrap value>70,这个分组就是可靠的。
上图中大家有没有好奇,为什么根节点处的自展值是98%,而NNF3与NNG1节点的自展值为33%,会不会太低不可信?这里说明下:如果低Bootstrap value靠近支末端,则可能是因为两支相似度太高,而很难分开;如果Bootstrap value更靠近根节点,则表示相似度较低。
二、构树三种方法综述
构建进化树可以是蛋白序列也可以是核酸序列,用类似树状分支的图表示各种(类)物种之间亲缘关系,并推测物种的进化历史。
构树三种方法:
1.邻接(邻位)法(NJ, Neighbor-joining):适用于大样本量,快速构建进化树。代表软件MEGA。
2.极大似然法(ML, Maximum likelihood):代表软件RAxml,RAxml可间接利用测序数据得到的vcf文件多线程、长时间分析来构建进化树。速度慢于NJ法。
3.贝叶斯法(Bayes):考虑构树参数且模型(默认JC模型)较多,一般要对核酸或蛋白序列做模型预测和各碱基先验频率。蛋白序列可选Prottest软件预测,核酸序列可用Modeltest软件预测。速度更慢于ML法。代表软件MrBayes。
三、回归文章
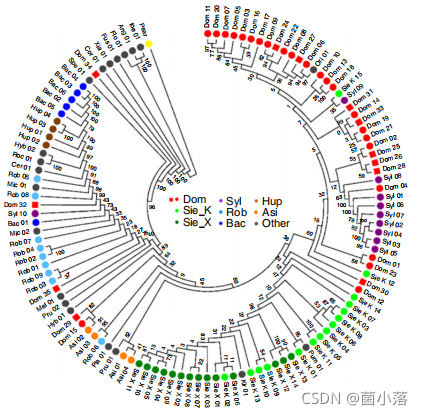
1、如果你的进化树看似样本混乱交叉,实则有实际生物学意义,不妨参考本文的描述,会有提神醒脑之效哟。
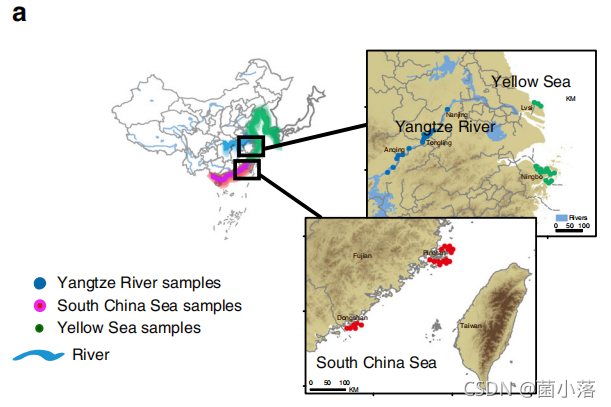
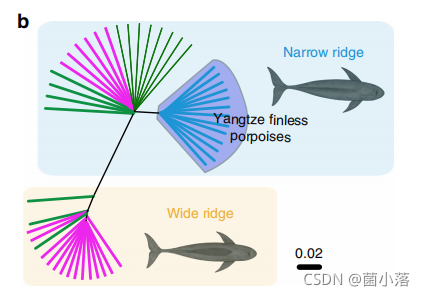
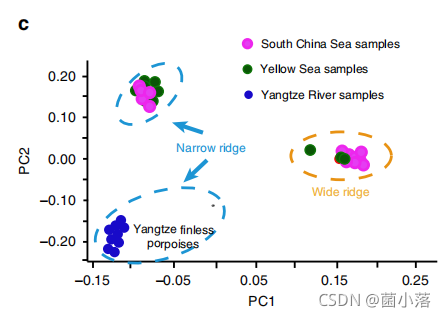
江豚分为窄脊和宽脊,上图进化树图可以看出:宽脊江豚包含了南海和黄海的个体;窄脊江豚则主要由黄海、南海和长江流域个体组成,而且只有来自长江的所有个体单独聚到一个亚群内,说明长江江豚的特异性,为文章长江江豚为一个独立物种的结论提供了有力支撑。
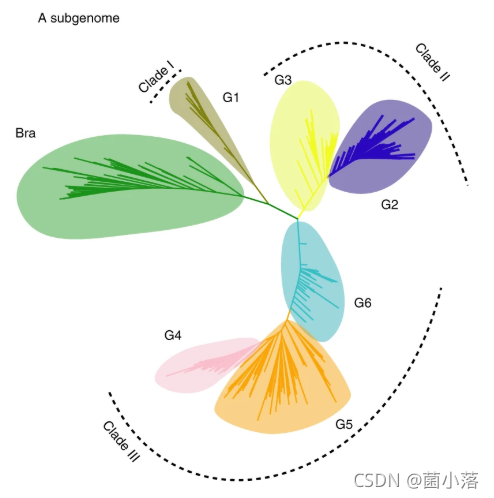
2、进化树与Strucutre以及表型数据结合,构建抓人眼球的酷炫图
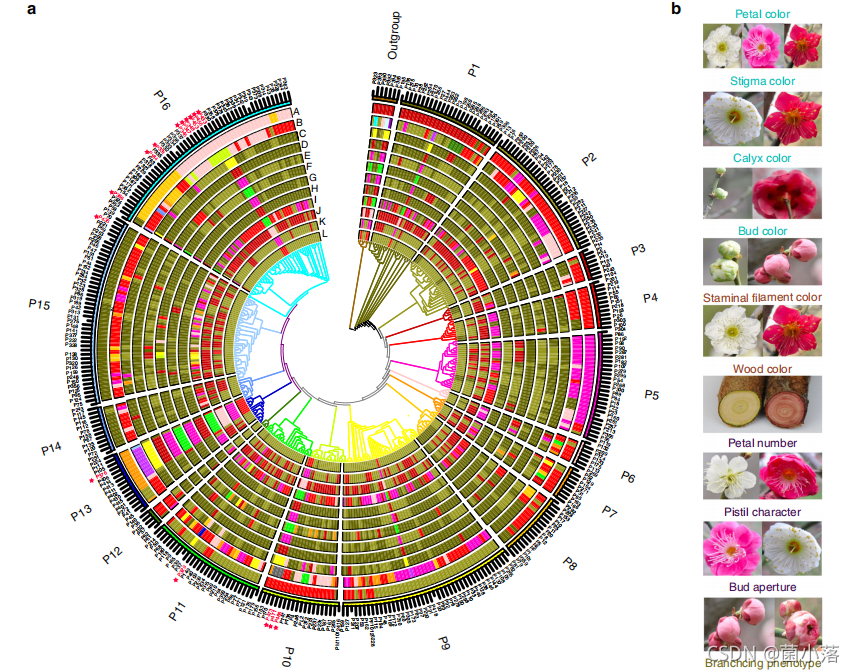
最内圈的分支状为进化树,包含16个亚群和一个外类群,分支颜色与最外圈样本ID颜色对应。其余圈层从A~L为群体结构,分别代表种群结构、品种群和各种性状表型(花瓣色、柱头色、花萼色、花蕾色、雄蕊花丝色、木色等,每个圆圈中的颜色代表性状的表型)。
四、总结
达尔文曾大胆猜想“所有生物来自于一个共同祖先,现存的和灭绝了的所有物种组成了一个宏伟的‘生命之树’,或称为系统发育树”。随着测序技术的发展,越来越多物种从原来的依据外部特征、SSR等分子标记,走向了基因组DNA水平分类,各种酷炫的进化树应用而生,是不是渐欲迷人眼?