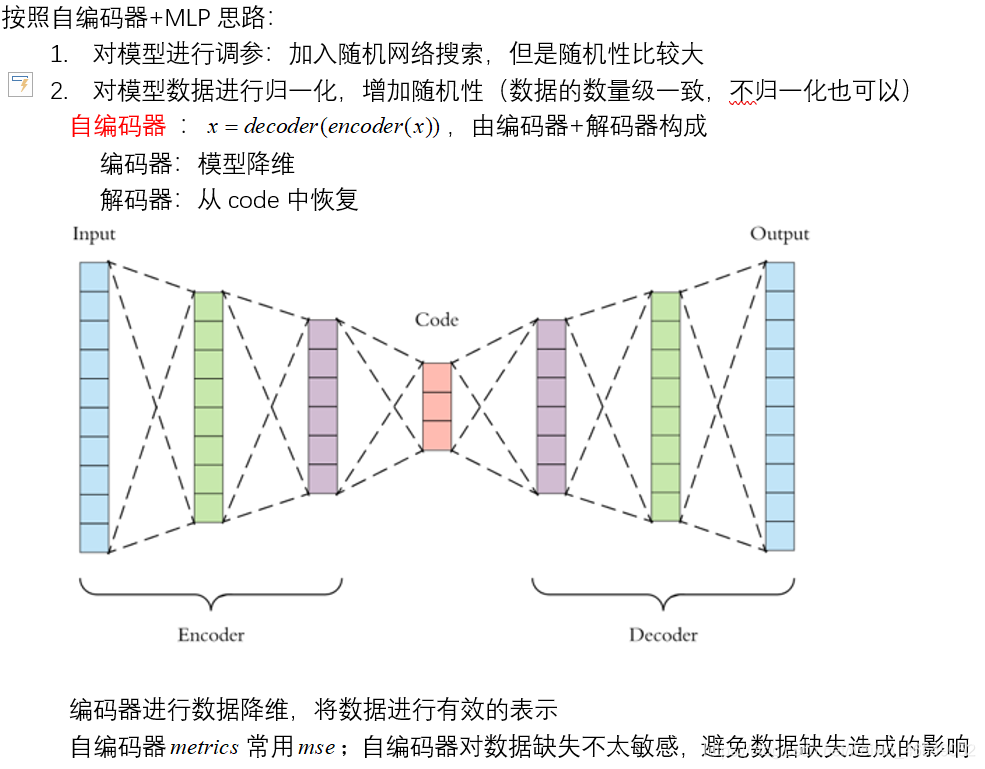
文章目录
一、结构化赛题知识点

二、赛题数据分析
1.resp字段分析

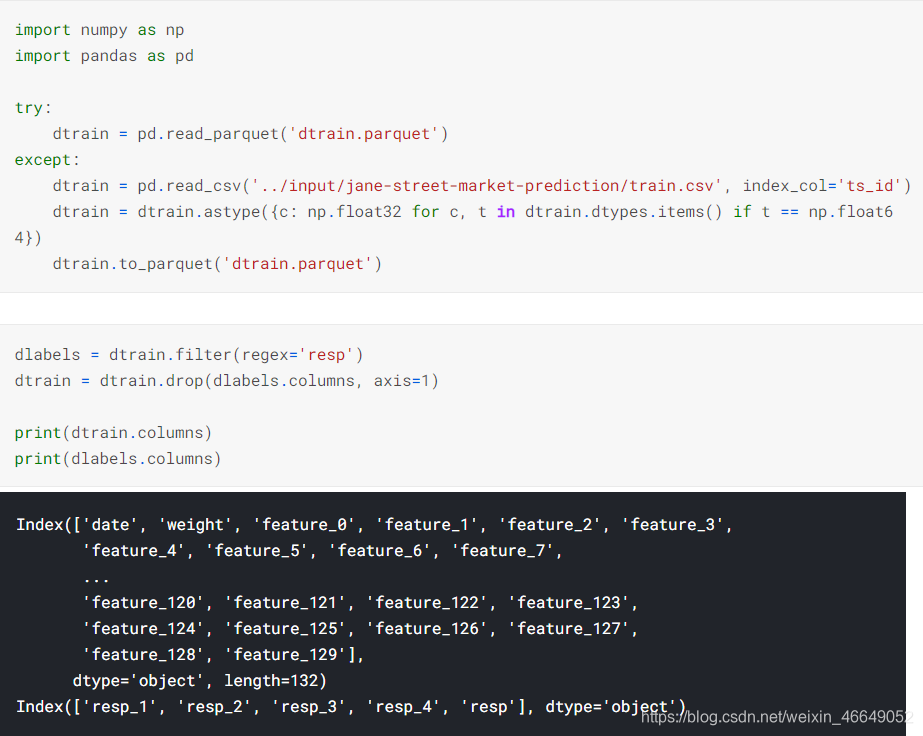
2. feature.csv数据
原网址

feature.csv的目的是显示匿名特征之间的关系,tag0〜tag28是特征推导中使用的匿名共享组件/概念。例如,如果(feature_i,tag_j)的值为True,则意味着tag_j用于派生feature_i。
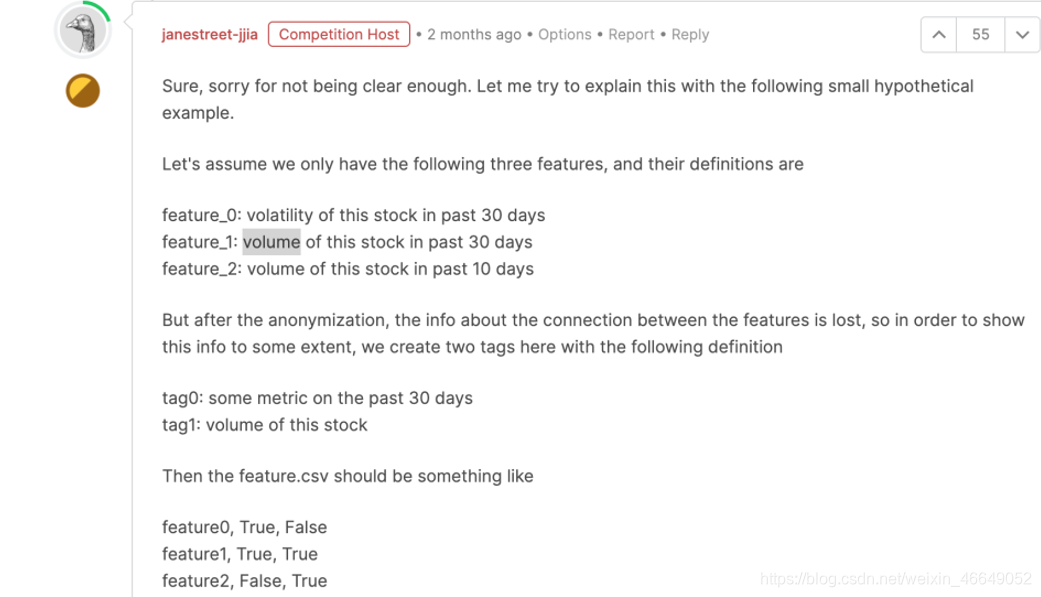

实际上tag是如何分布的?
参考网址
-
导入库
-
加载数据

-
对Example_sample_submission进行分析

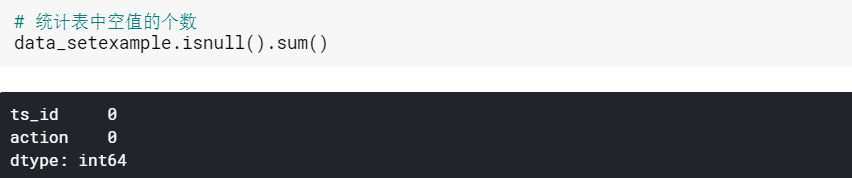
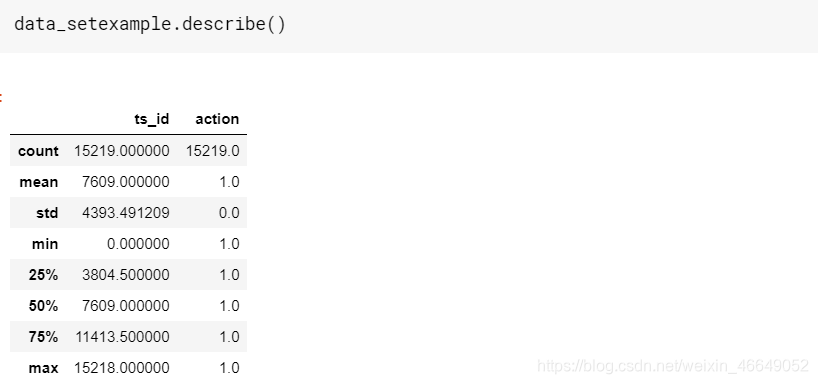
-
对feature.csv进行分析
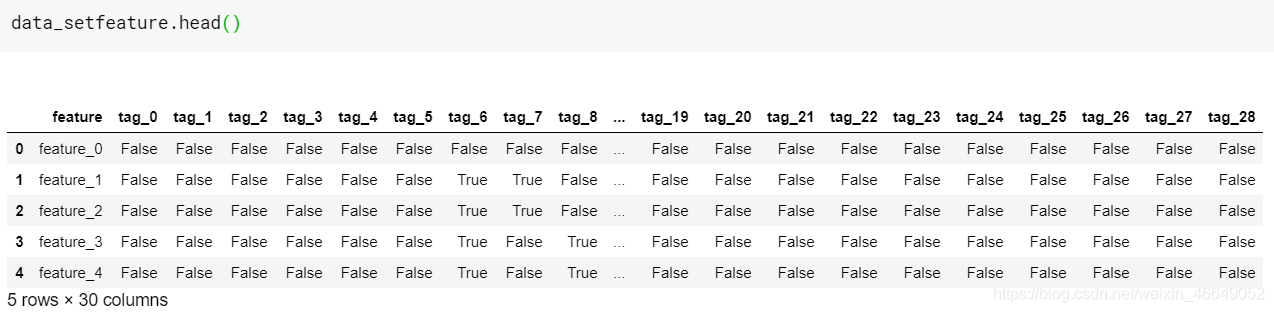
把表中每个feature的True+False进行统计



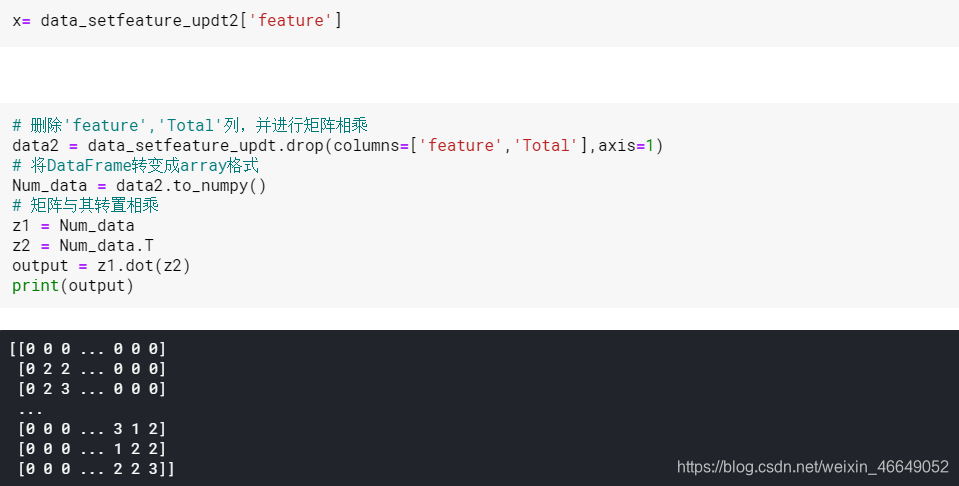

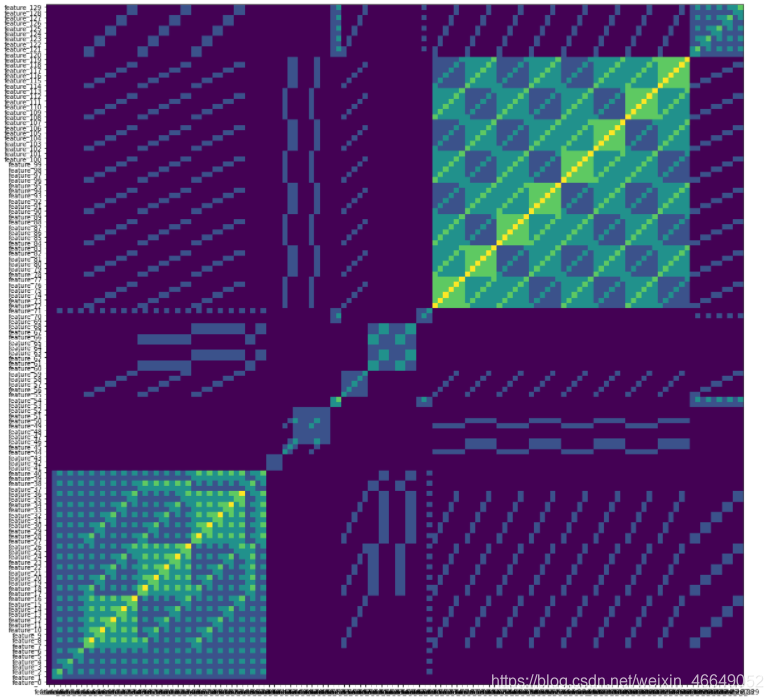
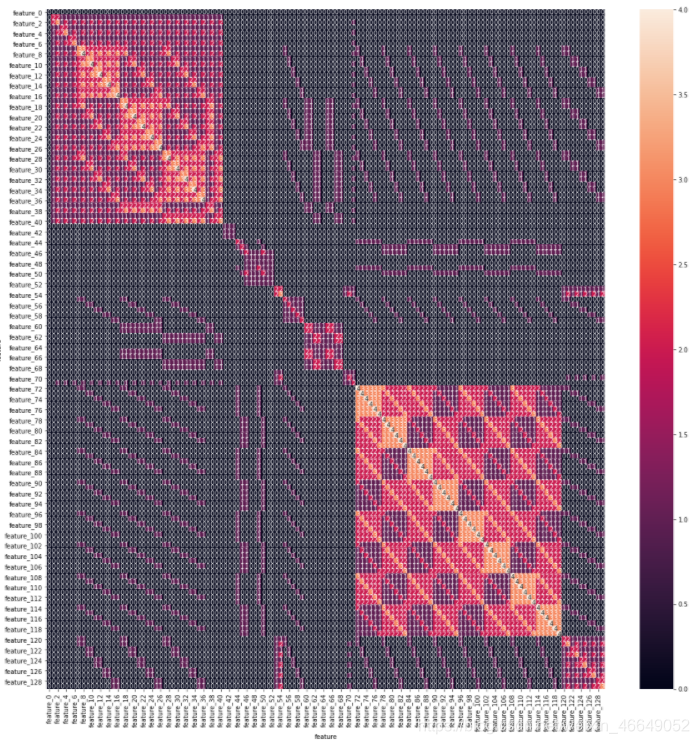
可以看出,一些特征与其他特征相比具有相似性。
Features 1 to 40 form set 1
Features 61 to 70 form set 2
Features 71 to 120 form set 3
Features 121 to 129 form set 4

- 对example_test数据进行分析
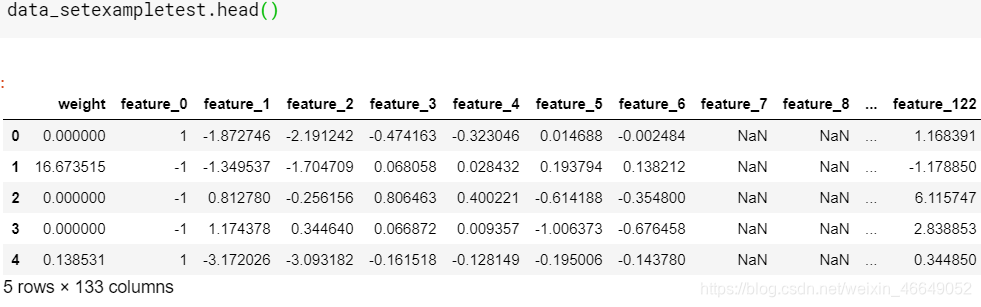
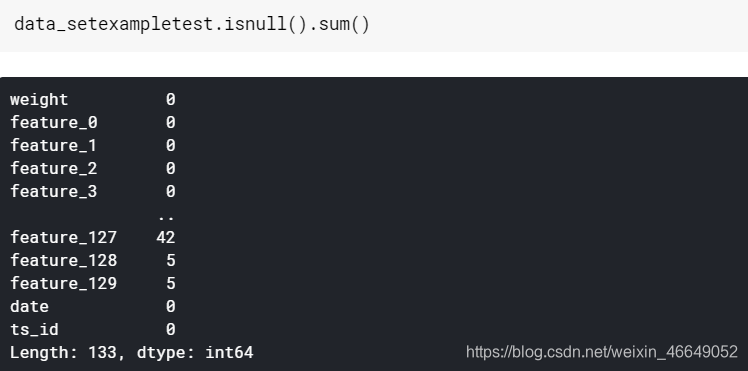
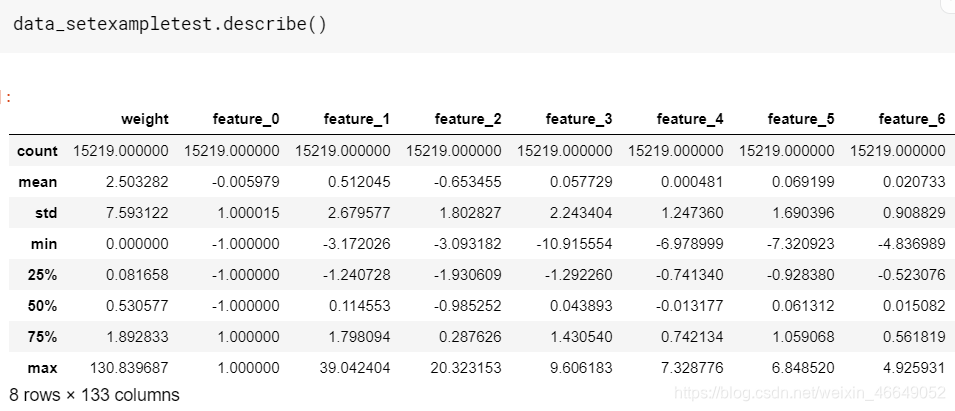
Data grouped by ID and Date

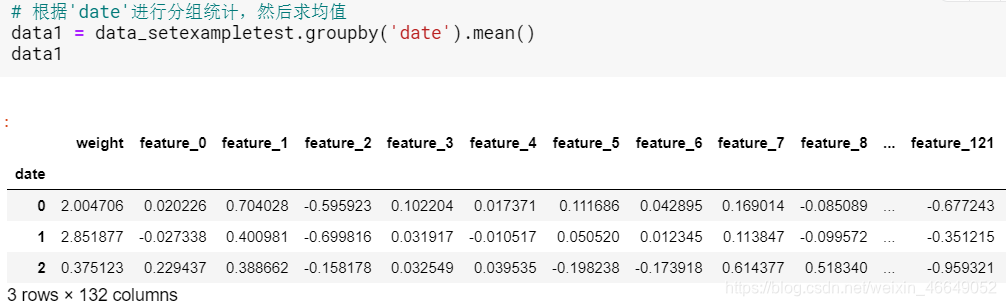
3.原始baseline中提出date>85(筛选数据),如何选择的?
原网址
这个网址中通过数据分析—从累计日收益的图表中找到趋势随时间的变化,发现许多特征似乎在第85天左右发生了变化。
网上有如下几种见解:
1.前85天是一个时间窗口,滚动计算时,由于数据不全导致第一个窗口不能用
我自己的看法是:在date比较小时,特征中数据缺失的比较多,因此是有可能是这个原因的。至于应该如何补充数据?还存在疑惑。常见的诸如填充均值、中值等不可行,利用随机森林算法等来填充的话,基于数据分布,会出现用未来的数据来预测过去的数据以进行填充,这个也是有问题的。所以可以考虑将缺失值前面缺失值较多的天数进行删除。
2.有人认为前85天与后面date可能是不同的交易模型。如果前85天数据趋势是由于市场波动导致与后几天数据趋势不同的话,那么应该保留几天的时间,以便更好地对这种情况进行建模。因为你不能保证测试数据所对应的时间内没有发生该市场波动。如果,发生了该市场波动,但是训练模型时对数据进行了删除,那么模型的泛化能力会很差!
4.分析feature_0
featue_0的数据格式为1,-1,尝试分析feature_0是否与每行数据的类别是对应的。下面用所有特征的肺形UMAP,表示两个主要的“肺” /簇,来展示具有的一些内部结构
- TriMap of features 1-130
对除feature_0以外的所有feature进行降维,以便从肺UMAP中找到两个主要聚类,分析这两个聚类是否对应于要素0的值?使用TriMAP代替UMAP进行分析,但是我希望我们会在TriMAP空间中看到类似的群集
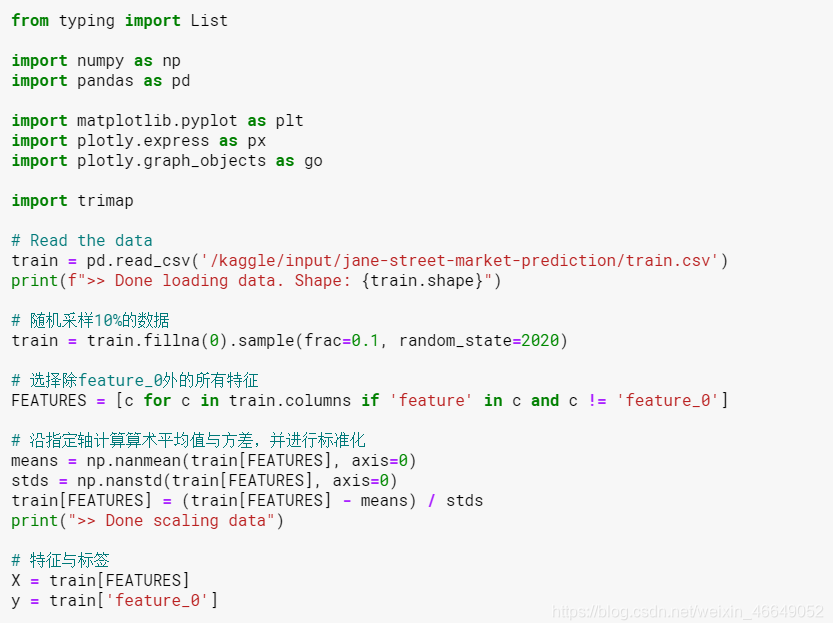
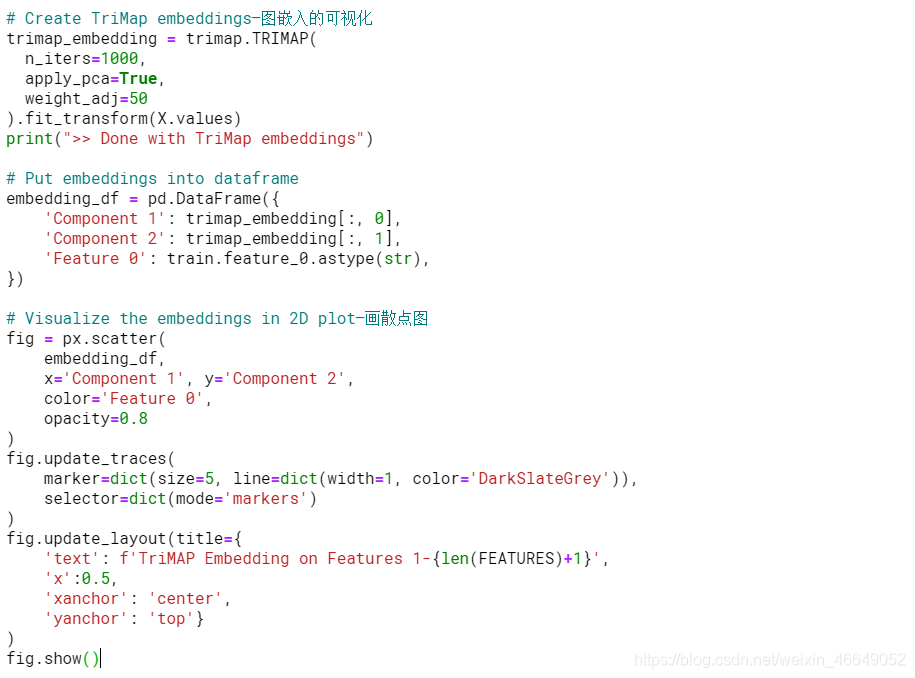
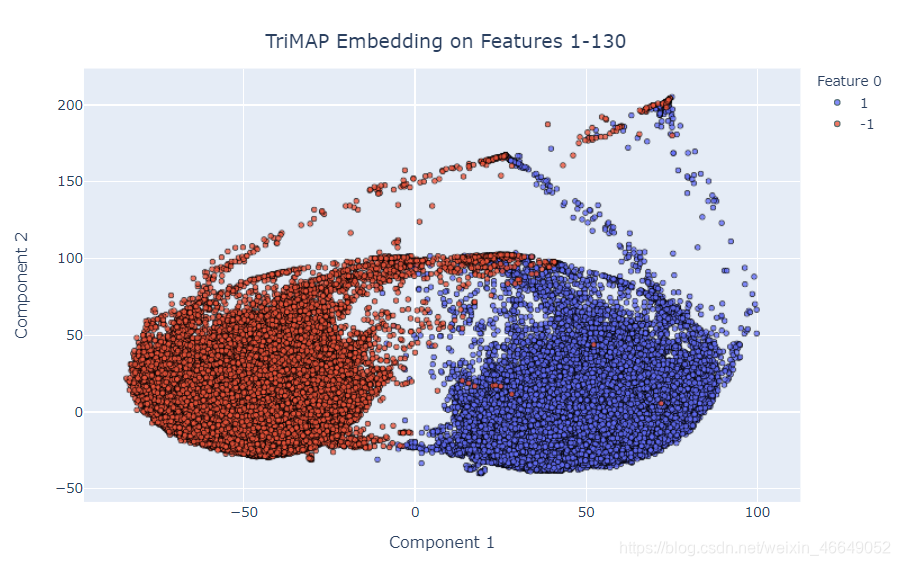
正如预期的那样,feature1-130分为两个不同的分布,实际上,这些分布与`feature_0’完全匹配。 这是否表示买入/卖出,买入/卖出或其他我不了解的金融知识,但是很显然,“ feature_0”的值会影响数据集中某些(如果不是全部)其他特征的值
- 找到与feature_0相关的特性
很明显,feature_0将其余的特性划分为两个数据分布,但是图只说明了这么多。为了进一步研究feature_0对其他特性的影响,我在这里尝试解决基于feature_0 1-130预测feature_0的反向问题。然后,我将删除一个最重要的特性(根据特性的重要性),并再次检查其余的特性对feature_0的预测能力。通过迭代地删除最重要的特性,我们可以了解预测feature_0需要多少特性,从而有多少特性与feature_0“相关
采用这种方法的原因不仅仅是查看“ feature_0”和“ feature_x”之间的相关性,还在于我们还想考虑可能的非线性效应,即“ feature_i”到“ feature_j”之间可能相互结合在一起预测“ feature_0”,但不能自行预测
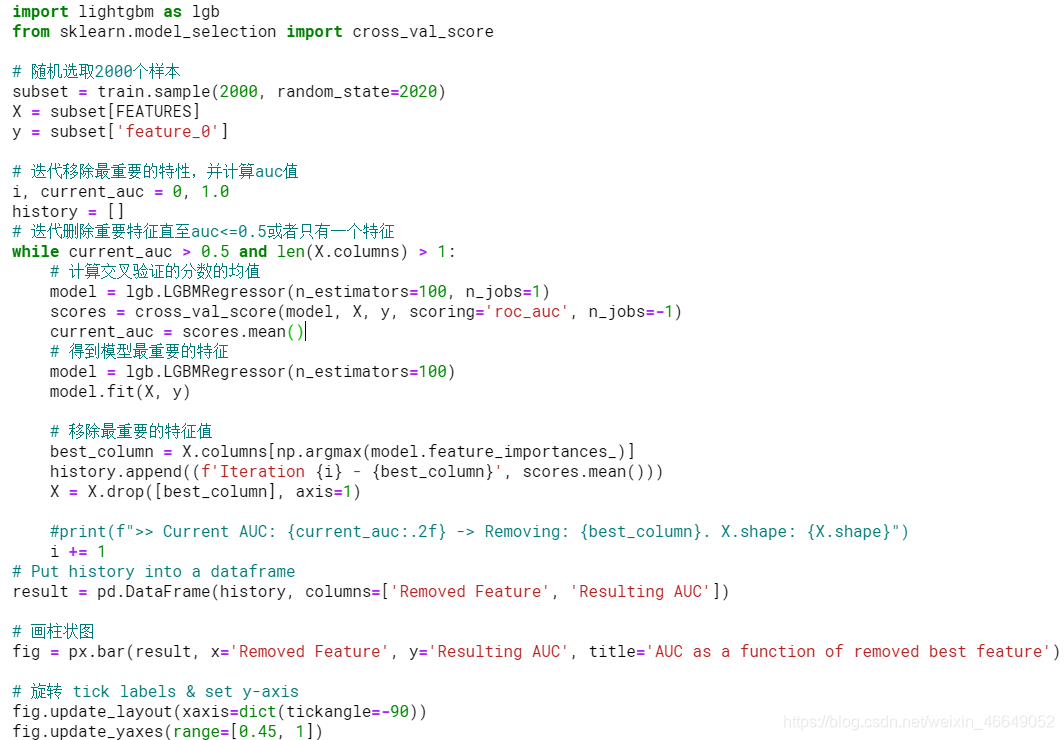
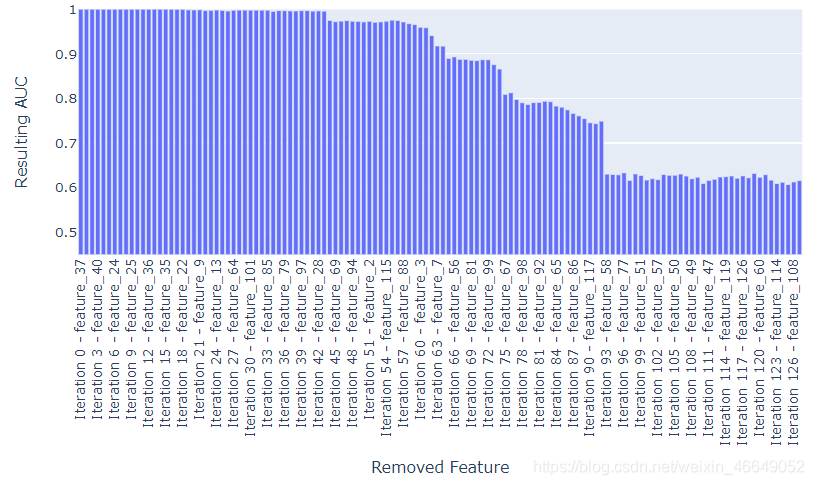
我们可以删除许多最重要的特性,但仍然能够完美地对feature_0进行分类—它不仅仅是一个依赖于feature_0值的单一特性的分布;相反,它是大量与feature_0值相关联的特性的分布。
- 特征间的相互关系
上面,反复地从其他特性中移除对feature_0最有预测性的特征,以了解与feature_0相关的最小特征集。在这个实验中,我将采用另一种方法,看看哪些特征本身足以预测feature_0的值

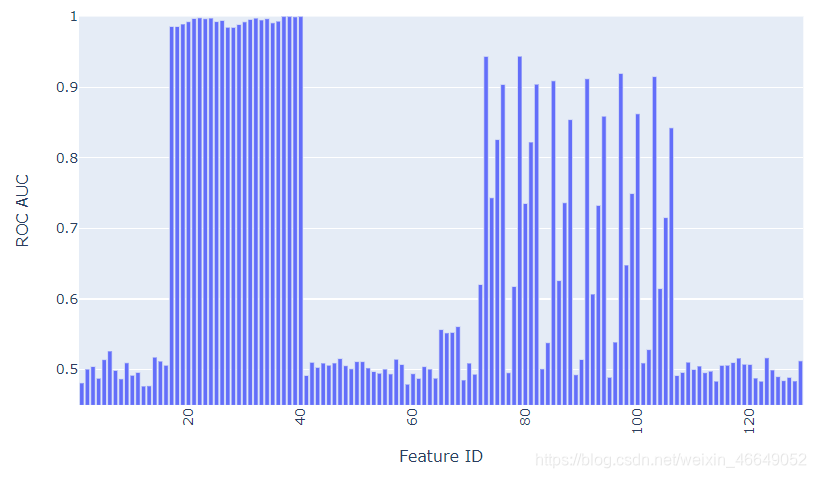
为了更好地衡量,让我们看看我们使用所有评分< 0.6的模型的AUC是有多少
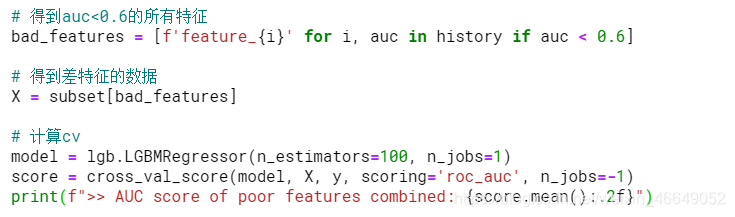
>> AUC score of poor features combined: 0.97
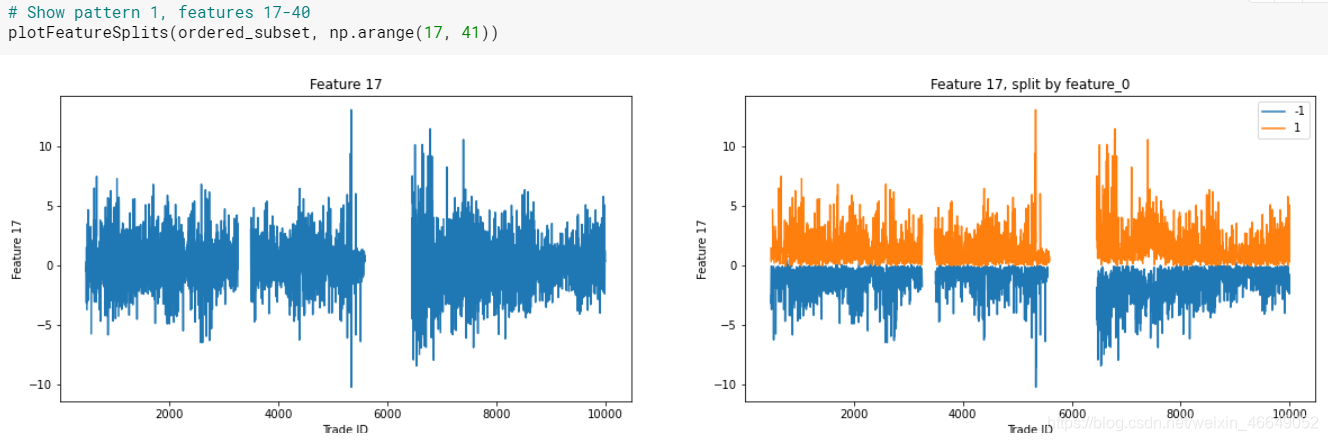
模式1:功能17到40非常擅长分隔Feature_0,查看tags文件,这似乎与给定的tags不是特别匹配
模式2:功能65-68似乎与feature_0有关,这些feature都没有任何标签
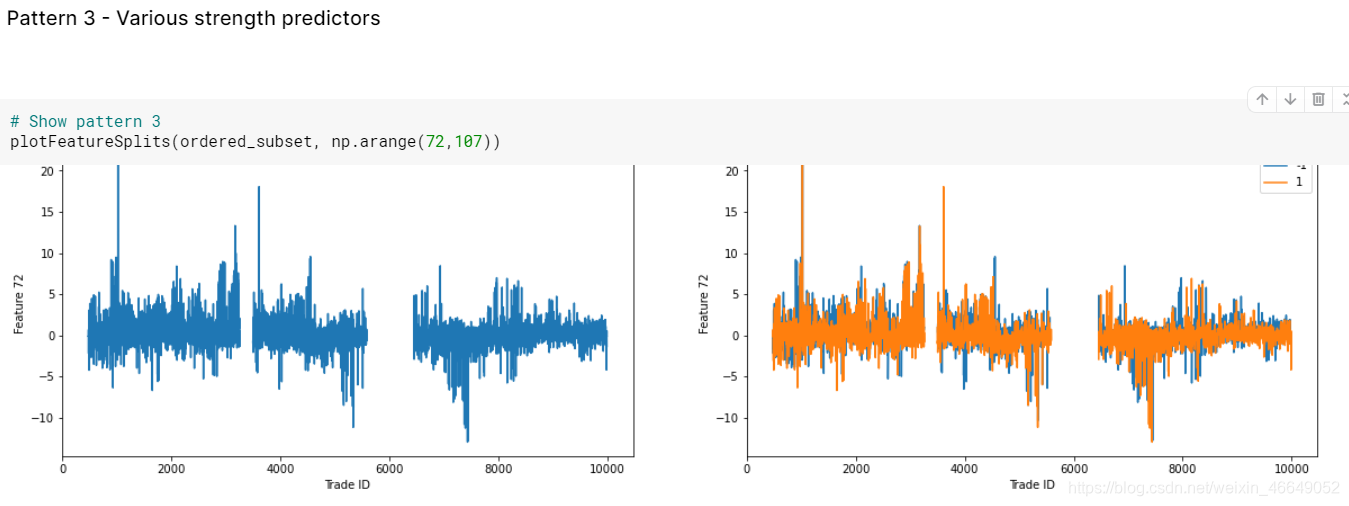
模式3:特征72-106似乎具有与feature_0相关的重复模式
模式4 :即使基于单个特征的模型评分数字表明某些特征本身与Feature_0不相关,但是当所有这些“较差”特征组合在一起时,它们就能够很好地对Feature_0进行分类,这表明Feature_0的值确实会产生影响许多其他特征的分布
- Inspecting feature patterns
使用上面所标识的不同模式,让我们看一下按feature_0划分的特征的实际值,看看我们是否可以实际观察到分布上的差异
图形部分未展示全,感兴趣的查看原网址!!!

5.做可视化有什么用?

三、模型训练与验证
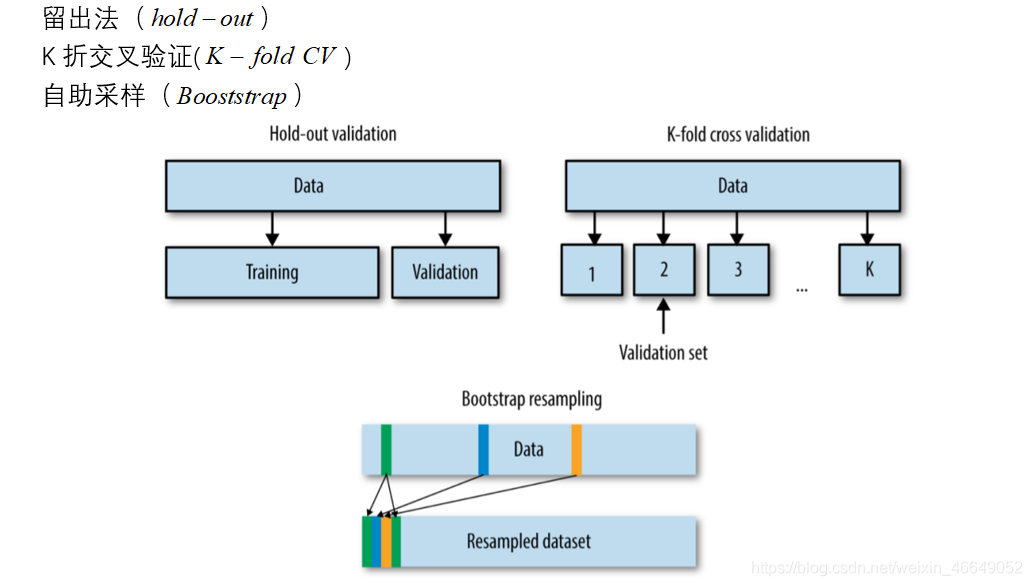
1.数据划分方法
2.时间序列数据划分方法
在这个网址里面,别人不仅做了细致分析,还提出了一种新的针对时序数据的划分方法

- train_test_split—留出法
使用shuffle=False;否则我们就会失去所有的时间顺序
这种方法存在问题,因为通过不断地验证相同的数据,我们也慢慢开始对测试集过度拟合(“泄漏”)。再次将测试集拆分为验证集可以解决此问题:在验证集上对模型的超参数进行调整和验证,一旦完全完成,我们将在测试集上对其进行测试(仅一次!)

2. Cross-validation—交叉验证
在交叉验证(CV)中,从训练集派生出多个验证集。
每折都会将训练集的新部分用作验证集,以前用于验证的数据现在又会成为训练集的一部分

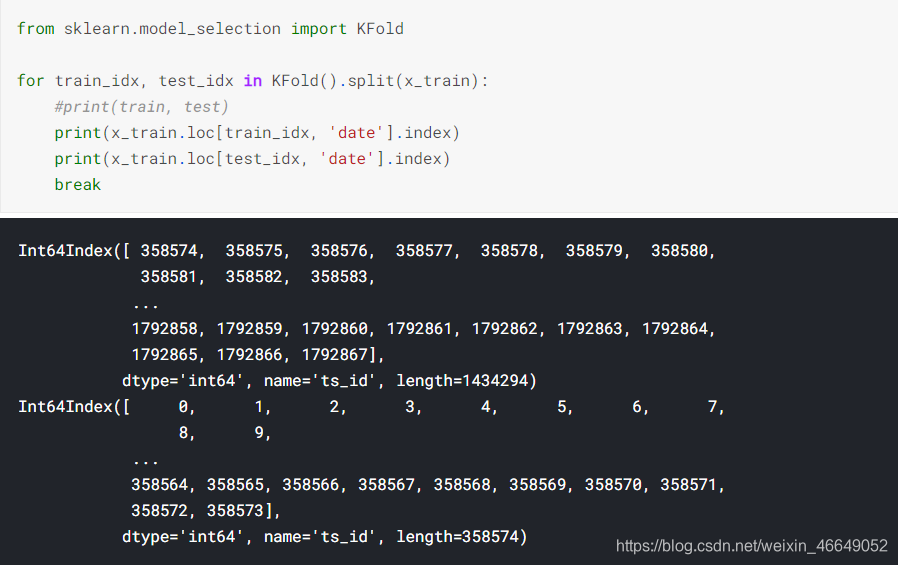
由结果可以看出,k折交叉验证分割的数据,测试集的索引部分小于训练集的索引,在时间序列预测问题中,用未来预测过去毫无意义!
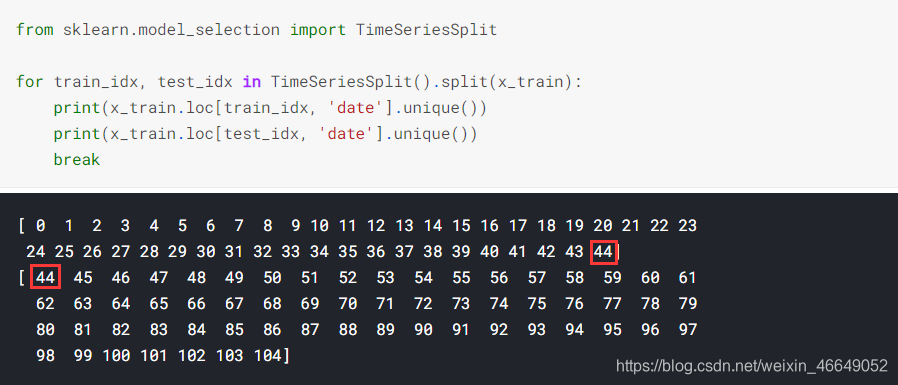
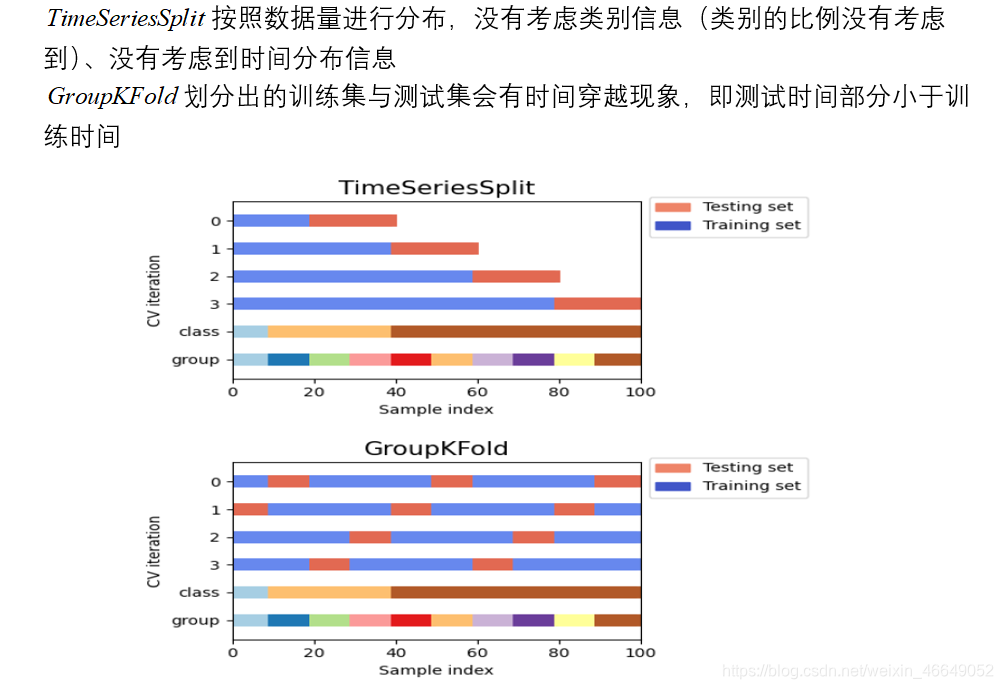
- TimeSeriesSplit—时间序列分割
TimeSeriesSplit与Cross-validation相比,解决了会出现测试集索引小于训练集索引的问题
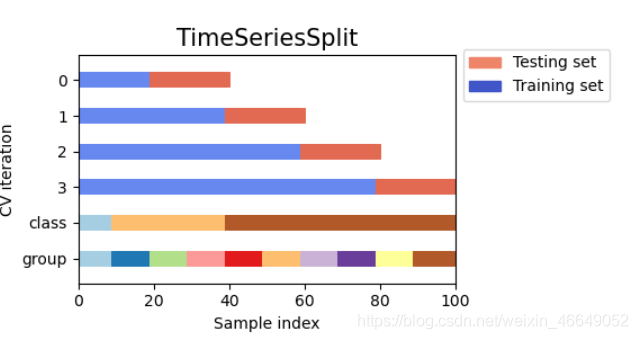
但是,TimeSeriesSplit不考虑数据中可用的组。虽然在这个图中不是很明显,但是我们可以想象,一个组可以部分地落在训练集中,部分地落在测试集中
从运行结果我们可以看出:
训练和测试集中都有来自第44天的数据,这意味着我们正在训练某一天一半的交易,只是为了验证它们在当天另一半交易中的表现。我们当然想要对特定一天的所有交易进行培训,并在随后的一天进行验证!否则会再次泄漏。
- GroupKFold—按组交叉验证
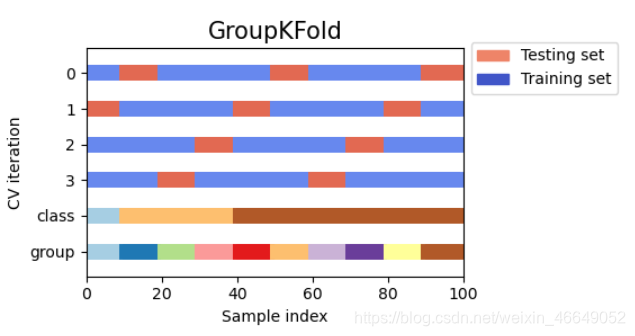
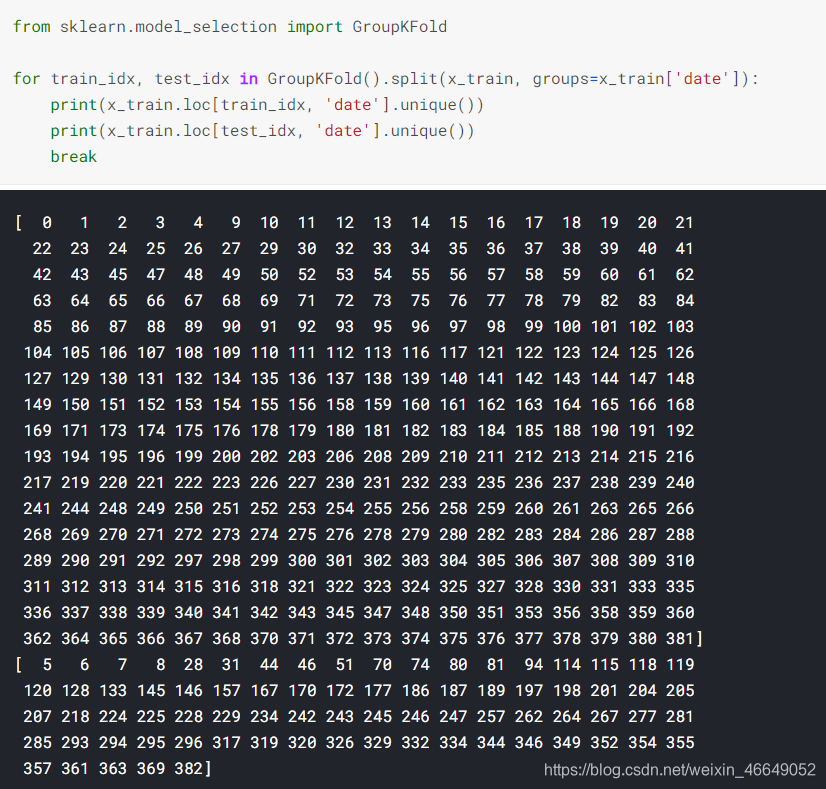
GroupKFold迭代器确实尊重了分组,一天的数据不会同时出现在训练集与测试集中,但是混淆了时间顺序,造成了时间穿越现象。我们需要对GroupKFold和TimeSeriesSplit进行结合:GroupTimesSeriesSplit
- GroupTimesSeriesSplit—时序数据分组切割
from sklearn.model_selection._split import _BaseKFold, indexable, _num_samples
from sklearn.utils.validation import _deprecate_positional_args
# https://github.com/getgaurav2/scikit-learn/blob/d4a3af5cc9da3a76f0266932644b884c99724c57/sklearn/model_selection/_split.py#L2243
class GroupTimeSeriesSplit(_BaseKFold):
"""Time Series cross-validator variant with non-overlapping groups.
Provides train/test indices to split time series data samples
that are observed at fixed time intervals according to a
third-party provided group.
In each split, test indices must be higher than before, and thus shuffling
in cross validator is inappropriate.
This cross-validation object is a variation of :class:`KFold`.
In the kth split, it returns first k folds as train set and the
(k+1)th fold as test set.
The same group will not appear in two different folds (the number of
distinct groups has to be at least equal to the number of folds).
Note that unlike standard cross-validation methods, successive
training sets are supersets of those that come before them.
Read more in the :ref:`User Guide <cross_validation>`.
Parameters
----------
n_splits : int, default=5
Number of splits. Must be at least 2.
max_train_size : int, default=None
Maximum size for a single training set.
Examples
--------
>>> import numpy as np
>>> from sklearn.model_selection import GroupTimeSeriesSplit
>>> groups = np.array(['a', 'a', 'a', 'a', 'a', 'a',\
'b', 'b', 'b', 'b', 'b',\
'c', 'c', 'c', 'c',\
'd', 'd', 'd'])
>>> gtss = GroupTimeSeriesSplit(n_splits=3)
>>> for train_idx, test_idx in gtss.split(groups, groups=groups):
... print("TRAIN:", train_idx, "TEST:", test_idx)
... print("TRAIN GROUP:", groups[train_idx],\
"TEST GROUP:", groups[test_idx])
TRAIN: [0, 1, 2, 3, 4, 5] TEST: [6, 7, 8, 9, 10]
TRAIN GROUP: ['a' 'a' 'a' 'a' 'a' 'a']\
TEST GROUP: ['b' 'b' 'b' 'b' 'b']
TRAIN: [0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10] TEST: [11, 12, 13, 14]
TRAIN GROUP: ['a' 'a' 'a' 'a' 'a' 'a' 'b' 'b' 'b' 'b' 'b']\
TEST GROUP: ['c' 'c' 'c' 'c']
TRAIN: [0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14]\
TEST: [15, 16, 17]
TRAIN GROUP: ['a' 'a' 'a' 'a' 'a' 'a' 'b' 'b' 'b' 'b' 'b' 'c' 'c' 'c' 'c']\
TEST GROUP: ['d' 'd' 'd']
"""
@_deprecate_positional_args
def __init__(self,
n_splits=5,
*,
max_train_size=None
):
super().__init__(n_splits, shuffle=False, random_state=None)
self.max_train_size = max_train_size
def split(self, X, y=None, groups=None):
"""Generate indices to split data into training and test set.
Parameters
----------
X : array-like of shape (n_samples, n_features)
Training data, where n_samples is the number of samples
and n_features is the number of features.
y : array-like of shape (n_samples,)
Always ignored, exists for compatibility.
groups : array-like of shape (n_samples,)
Group labels for the samples used while splitting the dataset into
train/test set.
Yields
------
train : ndarray
The training set indices for that split.
test : ndarray
The testing set indices for that split.
"""
if groups is None:
raise ValueError(
"The 'groups' parameter should not be None")
X, y, groups = indexable(X, y, groups)
n_samples = _num_samples(X)
n_splits = self.n_splits
n_folds = n_splits + 1
group_dict = {
}
u, ind = np.unique(groups, return_index=True)
unique_groups = u[np.argsort(ind)]
n_samples = _num_samples(X)
n_groups = _num_samples(unique_groups)
for idx in np.arange(n_samples):
if (groups[idx] in group_dict):
group_dict[groups[idx]].append(idx)
else:
group_dict[groups[idx]] = [idx]
if n_folds > n_groups:
raise ValueError(
("Cannot have number of folds={0} greater than"
" the number of groups={1}").format(n_folds,
n_groups))
group_test_size = n_groups // n_folds
group_test_starts = range(n_groups - n_splits * group_test_size,
n_groups, group_test_size)
for group_test_start in group_test_starts:
train_array = []
test_array = []
for train_group_idx in unique_groups[:group_test_start]:
train_array_tmp = group_dict[train_group_idx]
train_array = np.sort(np.unique(
np.concatenate((train_array,
train_array_tmp)),
axis=None), axis=None)
train_end = train_array.size
if self.max_train_size and self.max_train_size < train_end:
train_array = train_array[train_end -
self.max_train_size:train_end]
for test_group_idx in unique_groups[group_test_start:
group_test_start +
group_test_size]:
test_array_tmp = group_dict[test_group_idx]
test_array = np.sort(np.unique(
np.concatenate((test_array,
test_array_tmp)),
axis=None), axis=None)
yield [int(i) for i in train_array], [int(i) for i in test_array]
时序数据分割结果为

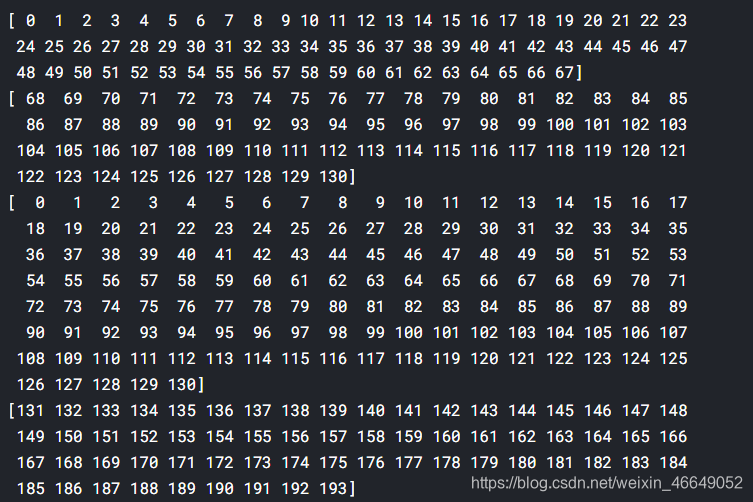
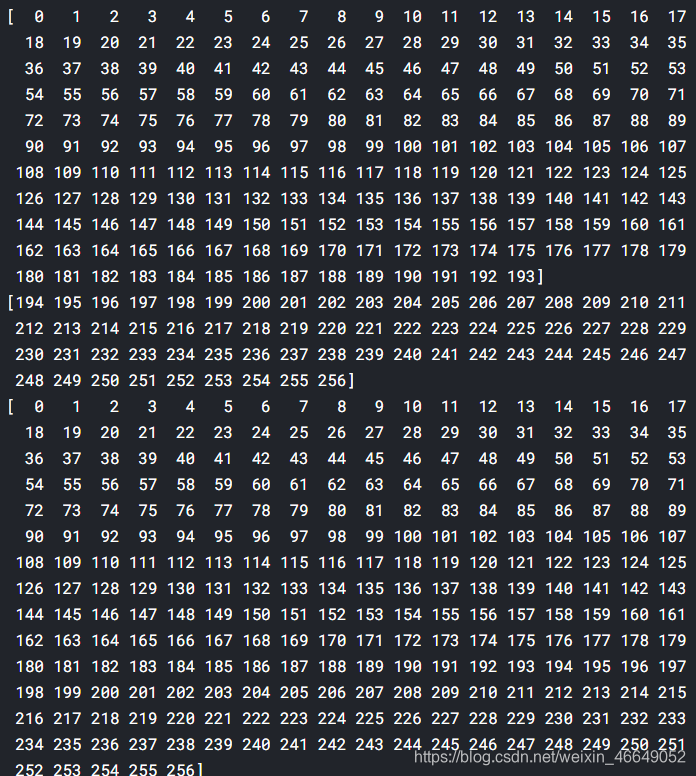
四、baseline强化路线