该代码是本人根据B站up主侯昶曦的代码所修改的。
原代码github地址:https://github.com/Houchangxi/heuristic-algorithm/blob/master/TSP问题遗传算法/Genetic Algorithm.py
遗传算法步骤不用讲了,将再多还是不会写代码,倒不如花一上午读懂下面的代码。不仅明白了具体步骤还能写出代码。
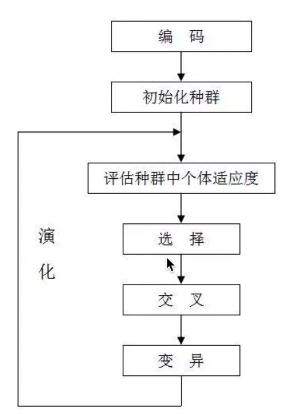
#!/usr/bin/env python
# coding: utf-8
# Author:侯昶曦 & 孟庆国
# Date:2020年5月19日 21点16分
# * 本代码中使用的城市坐标需要保存在一个`csv`类型的表中。
# * 下面的代码可以生成随机的指定数量的城市坐标,保存到当前目录的`cities.csv`文件中。
# * 如果需要本地数据,请在`main()`中修改文件路径。
# * 相关参数在`main()`中可以修改。
from matplotlib import pyplot as plt
import numpy as np
import pandas as pd
import time
# 生成城市坐标
city_num = 10 # 城市数量
name = ["city's name"] * city_num # 这个并没什么用,但是不要省,省了的话还要修改代码
x = [np.random.randint(0, 100) for i in range(city_num)]
y = [np.random.randint(0, 100) for i in range(city_num)]
with open("cities.csv", "w") as f:
for i in range(city_num):
f.write(name[i]+","+str(x[i])+","+str(y[i])+"\n")
f.write(name[0]+","+str(x[0])+","+str(y[0])+"\n") # 最后一个节点即为起点
# 打印城市的坐标
position = pd.read_csv("cities.csv", names=['ind','lat','lon'])
plt.scatter(x=position['lon'], y=position['lat'])
plt.show()
position.head()
def create_init_list(filename):
data = pd.read_csv(filename,names=['index','lat','lon']) # lat->纬度 lon->经度
data_list = []
for i in range(len(data)):
data_list.append([float(data.iloc[i]['lon']),float(data.iloc[i]['lat'])])
return data_list
def distance_matrix(coordinate_list, size): # 生成距离矩阵,邻接矩阵
d = np.zeros((size + 2, size + 2))
for i in range(size + 1):
x1 = coordinate_list[i][0]
y1 = coordinate_list[i][1]
for j in range(size + 1):
if (i == j) or (d[i][j] != 0):
continue
x2 = coordinate_list[j][0]
y2 = coordinate_list[j][1]
distance = np.sqrt((x1 - x2) ** 2 + (y1 - y2) ** 2)
if (i == 0): # 起点与终点是同一城市
d[i][j] = d[j][i] = d[size + 1][j] = d[j][size + 1] = distance
else:
d[i][j] = d[j][i] = distance
return d
def path_length(d_matrix, path_list, size): # 计算路径长度
length = 0
for i in range(size + 1):
length += d_matrix[path_list[i]][path_list[i + 1]]
return length
def shuffle(my_list):#起点和终点不能打乱
temp_list=my_list[1:-1]
np.random.shuffle(temp_list)
shuffle_list=my_list[:1]+temp_list+my_list[-1:]
return shuffle_list
def product_len_probability(my_list,d_matrix,size,p_num): # population, d, size,p_num
len_list=[] # 种群中每个个体(路径)的路径长度
pro_list=[]
path_len_pro=[]
for path in my_list:
len_list.append(path_length(d_matrix,path,size))
max_len=max(len_list)+1e-10
gen_best_length=min(len_list) # 种群中最优路径的长度
gen_best_length_index=len_list.index(gen_best_length) # 最优个体在种群中的索引
# 使用最长路径减去每个路径的长度,得到每条路径与最长路径的差值,该值越大说明路径越小
mask_list=np.ones(p_num)*max_len-np.array(len_list)
sum_len=np.sum(mask_list) # mask_list列表元素的和
for i in range(p_num):
if(i==0):
pro_list.append(mask_list[i]/sum_len)
elif(i==p_num-1):
pro_list.append(1)
else:
pro_list.append(pro_list[i-1]+mask_list[i]/sum_len)
for i in range(p_num):
# 路径列表 路径长度 概率
path_len_pro.append([my_list[i],len_list[i],pro_list[i]])
# 返回 最优路径 最优路径的长度 每条路径的概率
return my_list[gen_best_length_index],gen_best_length,path_len_pro
def choose_cross(population,p_num): # 随机产生交配者的索引,越优的染色体被选择几率越大
jump=np.random.random() # 随机生成0-1之间的小数
if jump<population[0][2]:
return 0
low=1
high=p_num
mid=int((low+high)/2)
# 二分搜索
# 如果jump在population[mid][2]和population[mid-1][2]之间,那么返回mid
while(low<high):
if jump>population[mid][2]:
low=mid
mid=(low+high) // 2
elif jump<population[mid-1][2]: # 注意这里一定是mid-1
high=mid
mid=(low+high) // 2
else:
return mid
def product_offspring(size, parent_1, parent_2, pm): # 产生后代
son = parent_1.copy()
product_set = np.random.randint(1, size+1)
parent_cross_set=set(parent_2[1:product_set]) # 交叉序列集合
cross_complete=1
for j in range(1,size+1):
if son[j] in parent_cross_set:
son[j]=parent_2[cross_complete]
cross_complete+=1
if cross_complete>product_set:
break
if np.random.random() < pm: #变异
son=veriation(son,size,pm)
return son
def veriation(my_list,size,pm):#变异,随机调换两城市位置
ver_1=np.random.randint(1,size+1)
ver_2=np.random.randint(1,size+1)
while ver_2==ver_1:#直到ver_2与ver_1不同
ver_2 = np.random.randint(1, size+1)
my_list[ver_1],my_list[ver_2]=my_list[ver_2],my_list[ver_1]
return my_list
def main(filepath, p_num, gen, pm):
start = time.time()
coordinate_list=create_init_list(filepath)
size=len(coordinate_list)-2 # 除去了起点和终点
d=distance_matrix(coordinate_list,size) # 各城市之间的邻接矩阵
path_list=list(range(size+2)) # 初始路径
# 随机打乱初始路径以建立初始种群路径
population = [shuffle(path_list) for i in range(p_num)]
# 初始种群population以及它的最优路径和最短长度
gen_best,gen_best_length,population=product_len_probability(population,d,size,p_num)
# 现在的population中每一元素有三项,第一项是路径,第二项是长度,第三项是使用时转盘的概率
son_list = [0] * p_num # 后代列表
best_path=gen_best # 最好路径初始化
best_path_length=gen_best_length # 最好路径长度初始化
every_gen_best=[gen_best_length] # 每一代的最优值
for i in range(gen): #迭代gen代
son_num=0
while son_num < p_num: # 循环产生后代,一组父母亲产生两个后代
father_index = choose_cross(population, p_num) # 获得父母索引
mother_index = choose_cross(population, p_num)
father = population[father_index][0] # 获得父母的染色体
mother = population[mother_index][0]
son_list[son_num] = product_offspring(size, father, mother, pm) # 产生后代加入到后代列表中
son_num += 1
if son_num == p_num:
break
son_list[son_num] = product_offspring(size, mother, father, pm) # 产生后代加入到后代列表中
son_num += 1
# 在新一代个体中找到最优路径和最优值
gen_best, gen_best_length,population = product_len_probability(son_list,d,size,p_num)
if(gen_best_length < best_path_length): # 这一代的最优值比有史以来的最优值更优
best_path=gen_best
best_path_length=gen_best_length
every_gen_best.append(gen_best_length)
end = time.time()
print(f"迭代用时:{(end-start)}s")
print("史上最优路径:", best_path, sep=" ")#史上最优路径
print("史上最短路径长度:", best_path_length, sep=" ")#史上最优路径长度
# 打印各代最优值和最优路径
x = [coordinate_list[point][0] for point in best_path] # 最优路径各节点经度
y = [coordinate_list[point][1] for point in best_path] # 最优路径各节点纬度
plt.figure(figsize=(8, 10))
plt.subplot(211)
plt.plot(every_gen_best) # 画每一代中最优路径的路径长度
plt.subplot(212)
plt.scatter(x,y) # 画点
plt.plot(x,y) # 画点之间的连线
plt.grid() # 给画布添加网格
plt.show()
if __name__ == '__main__':
filepath = r'cities.csv' # 城市坐标数据文件
p_num = 200 #种群个体数量
gen = 1000 #进化代数
pm = 0.1 #变异率
main(filepath, p_num, gen, pm)