逻辑回归:
逻辑回归是最常用的分类算法之一。
from sklearn.linear_model import LogisticRegression
logreg=LogisticRegression().fit(x_train,y_train)
print("Training set score:{:.3f}".format(logreg.score(x_train,y_train)))
print("Test set score:{:.3f}".format(logreg.score(x_test,y_test)))
Training set score:0.781
Test set score:0.771
正则化参数C=1(默认值)的模型在训练集上准确度为78%,在测试集上准确度为77%。
logreg100=LogisticRegression(C=100).fit(x_train,y_train)
print("Training set score:{:.3f}".format(logreg100.score(x_train,y_train)))
print("Test set score:{:.3f}".format(logreg100.score(x_test,y_test)))
Training set score:0.785
Test set score:0.766
而将正则化参数C设置为100时,模型在训练集上准确度稍有提高但测试集上准确度略降,说明较少正则化和更复杂的模型并不一定会比默认参数模型的预测效果更好。
因此,我们选择默认值C=1。
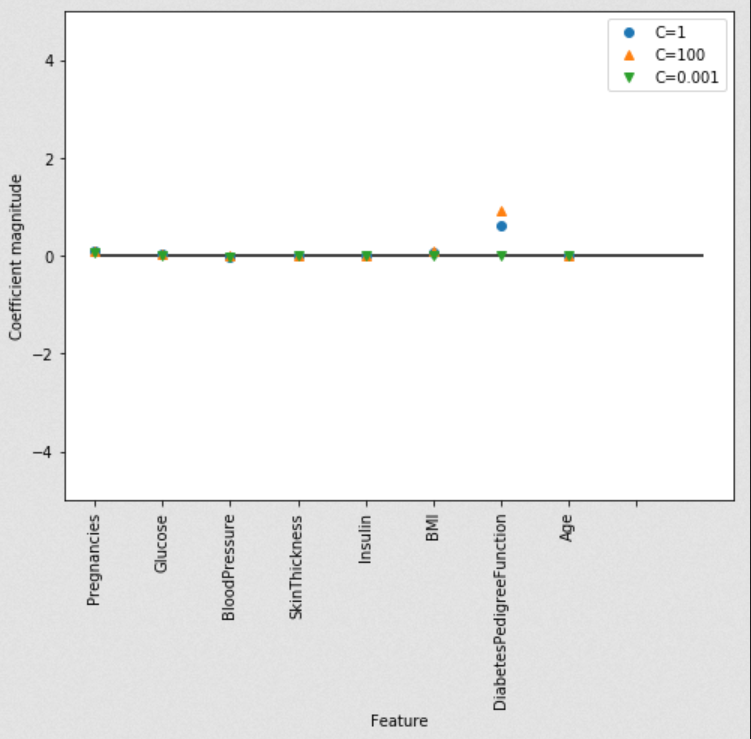
让我们用可视化的方式来看一下用三种不同正则化参数C所得模型的系数。
更强的正则化(C = 0.001)会使系数越来越接近于零。仔细地看图,我们还能发现特征“DiabetesPedigreeFunction”(糖尿病遗传函数)在 C=100, C=1 和C=0.001的情况下, 系数都为正。这表明无论是哪个模型,DiabetesPedigreeFunction(糖尿病遗传函数)这个特征值都与样本为糖尿病是正相关的。
diabetes_features=[x for i,x in enumerate(diabetes.columns) if i!=8]
plt.figure(figsize=(8,6))
plt.plot(logreg.coef_.T,'o',label="C=1")
plt.plot(logreg100.coef_.T,'^',label="C=100")
plt.plot(logreg001.coef_.T,'v',label="C=0.001")
plt.xticks(range(diabetes.shape[1]),diabetes_features,rotation=90)
plt.hlines(0,0,diabetes.shape[1])
plt.ylim(-5,5)
plt.xlabel("Feature")
plt.ylabel("Coefficient magnitude")
plt.legend()
决策树:
from sklearn.tree import DecisionTreeClassifier
tree=DecisionTreeClassifier(random_state=0)
tree.fit(x_train,y_train)
print("Accuracy on training set:{:.3f}".format(tree.score(x_train,y_train)))
print("Accuracy on test set:{:.3f}".format(tree.score(x_test,y_test)))
Accuracy on training set:1.000
Accuracy on test set:0.714
训练集的准确度可以高达100%,而测试集的准确度相对就差了很多。这表明决策树是过度拟合的,不能对新数据产生好的效果。因此,我们需要对树进行预剪枝。
我们设置max_depth=3,限制树的深度以减少过拟合。这会使训练集的准确度降低,但测试集准确度提高。
tree=DecisionTreeClassifier(max_depth=3,random_state=0)
tree.fit(x_train,y_train)
print("Accuracy on training set:{:.3f}".format(tree.score(x_train,y_train)))
print("Accuracy on test set:{:.3f}".format(tree.score(x_test,y_test)))
Accuracy on training set:0.773
Accuracy on test set:0.740
决策树中的特征重要度:
决策树中的特征重要度是用来衡量每个特征对于预测结果的重要性的。对每个特征有一个从0到1的打分,0表示“一点也没用”,1表示“完美预测”。各特征的重要度加和一定是为1的。
print("Feature importances:\n{}".format(tree.feature_importances_))
Feature importances: [ 0.04554275 0.6830362 0. 0. 0. 0.27142106 0. 0. ]
然后我们能可视化特征重要度:
def plot_feature_importances_diabetes(model):
plt.figure(figsize=(8,6))
n_features=8
plt.barh(range(n_features),model.feature_importances_,align='center')
plt.yticks(np.arange(n_features),diabetes_features)
plt.xlabel("Feature importance")
plt.ylabel("Feature")
plt.ylim(-1,n_features)
plot_feature_importances_diabetes(tree)
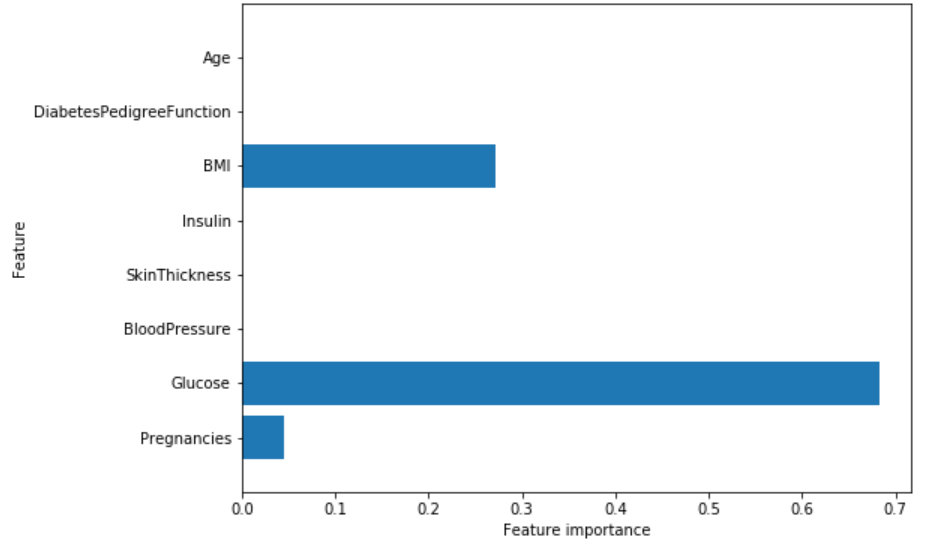
特征“血糖”是目前最重要的特征。
随机森林:
让我们在糖尿病数据集中应用一个由100棵树组成的随机森林:
from sklearn.ensemble import RandomForestClassifier
rf=RandomForestClassifier(n_estimators=100,random_state=0)
rf.fit(x_train,y_train)
print("Accuracy on training set:{:.3f}".format(rf.score(x_train,y_train)))
print("Accuracy on test set:{:.3f}".format(rf.score(x_test,y_test)))
Accuracy on training set:1.000
Accuracy on test set:0.786
没有更改任何参数的随机森林有78.6%的准确度,比逻辑回归和单一决策树的预测效果更好。然而,我们还是可以调整max_features设置,看看效果是否能够提高。
rf1=RandomForestClassifier(max_depth=3,n_estimators=100,random_state=0)
rf1.fit(x_train,y_train)
print("Accuracy on training set:{:.3f}".format(rf1.score(x_train,y_train)))
print("Accuracy on test set:{:.3f}".format(rf1.score(x_test,y_test)))
Accuracy on training set:0.800
Accuracy on test set:0.755
结果并没有提高,这表明默认参数的随机森林在这里效果很好。
随机森林的特征重要度:
plot_feature_importances_diabetes(rf1)

与单一决策树相似,随机森林的结果仍然显示特征“血糖”的重要度最高,但是它也同样显示“BMI(身体质量指数)”在整体中是第二重要的信息特征。随机森林的随机性促使算法考虑了更多可能的解释,这就导致随机森林捕获的数据比单一树要大得多。