Referencia de este blog: iris-análisis de casos clásicos-aprendizaje automático

Los problemas que queremos resolver son los siguientes:
Se sabe que el iris se divide en tres tipos diferentes: iris de montaña Setosa, iris que cambia de color Versicolor y iris de Virginia Virginica.Esta clasificación se basa principalmente en la longitud y anchura del cáliz y la longitud y anchura de los pétalos.Cuatro métricas (y posiblemente otras referencias también). No conocemos los criterios de clasificación exactos, pero los botánicos han clasificado 150 iris diferentes y podemos medir con precisión los pétalos del cáliz de cada iris.
Entonces aquí viene la pregunta, un lirio en casa de tu novia ha florecido, ella lo midió, el largo y el ancho del cáliz y los pétalos son 3.1, 2.3, 1.2, 0.5, y luego te pregunta: "Este lirio en mi casa ¿A qué categoría pertenece?"
Directorio de artículos
1. Verifica los datos
¿Hay algún problema con el formato de datos?
¿Hay algún problema con los valores de los datos?
¿Es necesario reparar y eliminar los datos?
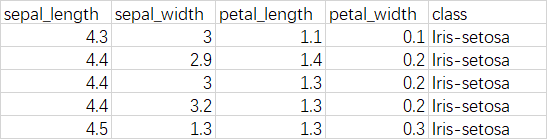
表格说明:横行属于一朵花的数据
Sepal length/width:花萼的长度/宽度数据
Petal length/width:花瓣的长度/宽度数据
class:植物学家鉴定的花的类型
import seaborn as sns
import numpy as np
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
%matplotlib inline
plt.rcParams['font.sans-serif']=['Microsoft YaHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False # 用来正常显示负号
from datetime import datetime
plt.figure(figsize=(16,10))
import pyecharts.options as opts
from pyecharts.charts import Line
from pyecharts.faker import Faker
from pyecharts.charts import Bar
import os
from pyecharts.options.global_options import ThemeType
iris_data=pd.read_csv("iris.csv",na_values='NA') # na_values:在读取的时候直接将空值赋值为NA
iris_data.head()
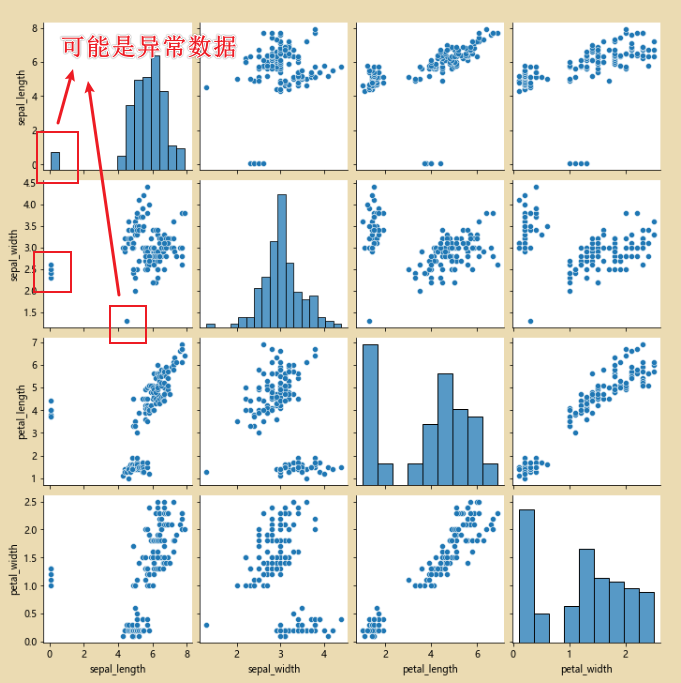
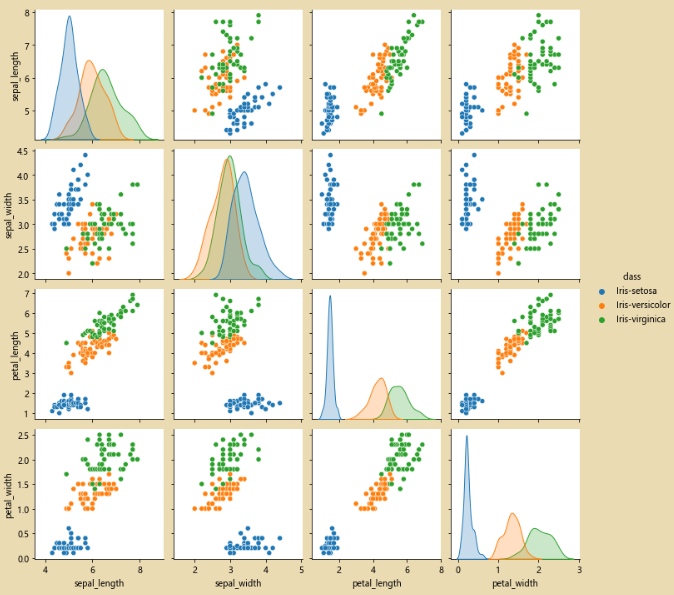
# 使用pairpoint进行检查,看看原始的数据是否有问题
sns.pairplot(iris_data.dropna())
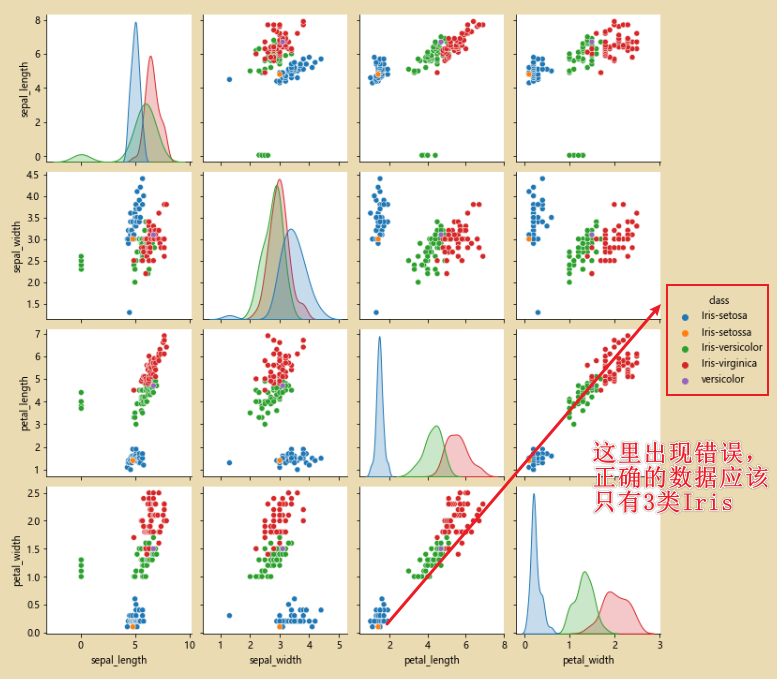
sns.pairplot(iris_data.dropna(),hue='class')# hue='class' 按照class进行分类
2. Limpiar los datos
# 3 对数据进行修正
cond=(iris_data['class']=='Iris-setosa') & (iris_data['sepal_width']<2.5)
iris_data.loc[cond]
iris_data.loc[iris_data['class']=='versicolor','class']='Iris-versicolor'
iris_data.loc[iris_data['class']=='Iris-setossa','class']='Iris-setosa'
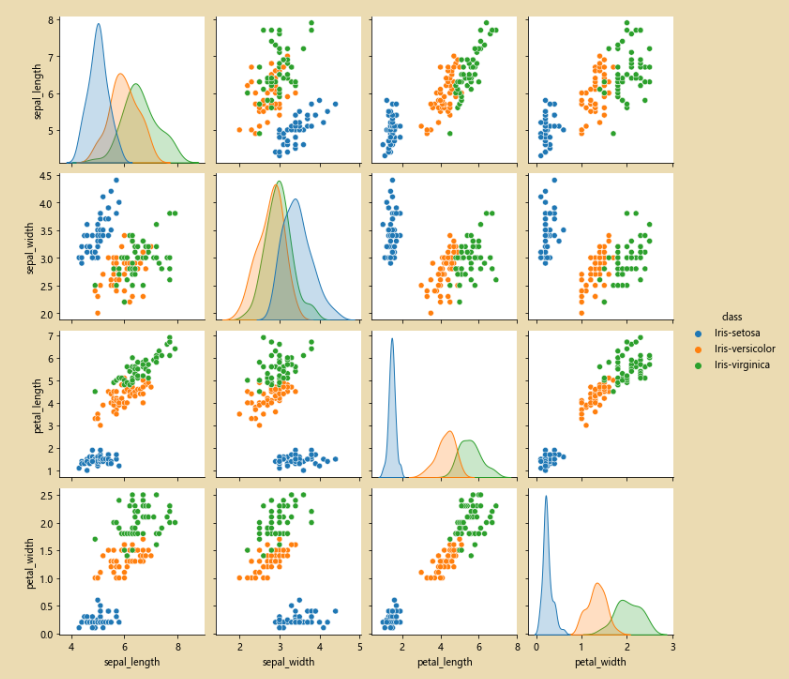
sns.pairplot(iris_data.dropna(),hue='class')
En este punto, después de la corrección, solo hay tres tipos de
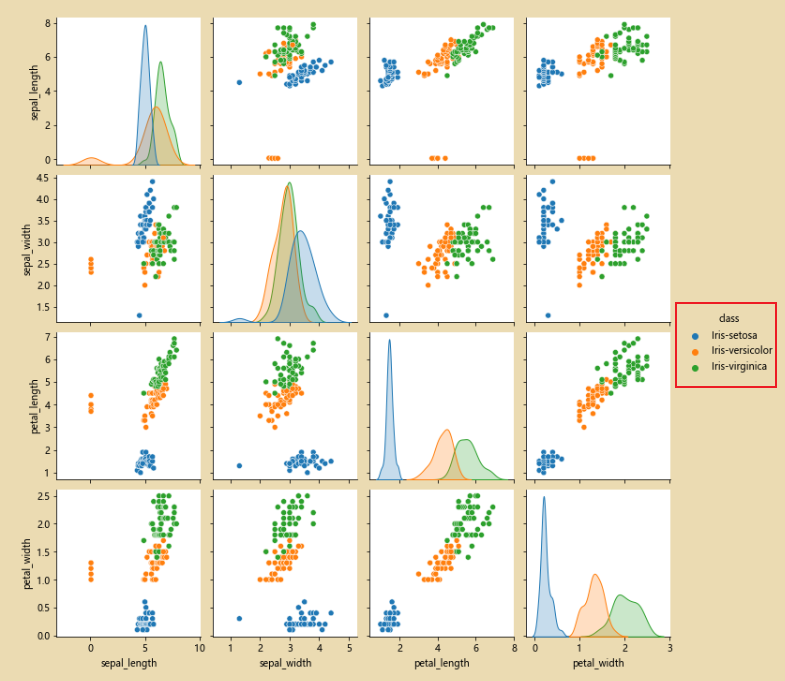
datos.Mostrar imágenes:
from PIL import Image
img = Image.open('test.jpg')
plt.imshow(img)
plt.show()

1. Corrija el valor faltante NAN
# 对缺失值NAN进行修正(找出所有字段的缺失值)
iris_data.loc[
(iris_data['sepal_width'].isnull()) |
(iris_data['sepal_length'].isnull()) |
(iris_data['petal_width'].isnull()) |
(iris_data['petal_length'].isnull())
]
Valores atípicos encontrados:
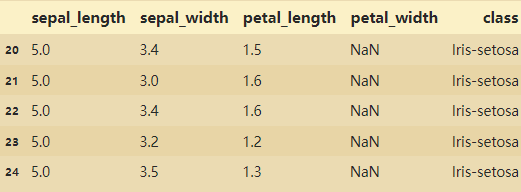
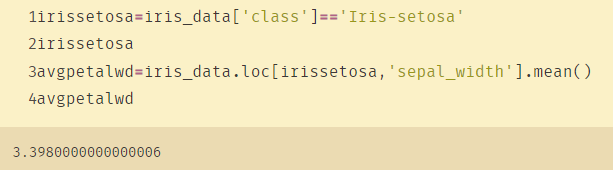
# 使用mean均值对nan进行替换
irissetosa=iris_data['class']=='Iris-setosa'
irissetosa
avgpetalwd=iris_data.loc[irissetosa,'petal_width'].mean()
avgpetalwd
iris_data.loc[irissetosa & (iris_data['petal_width'].isnull()),'petal_width']=avgpetalwd

2. Corrija los datos anormales de sepal_width
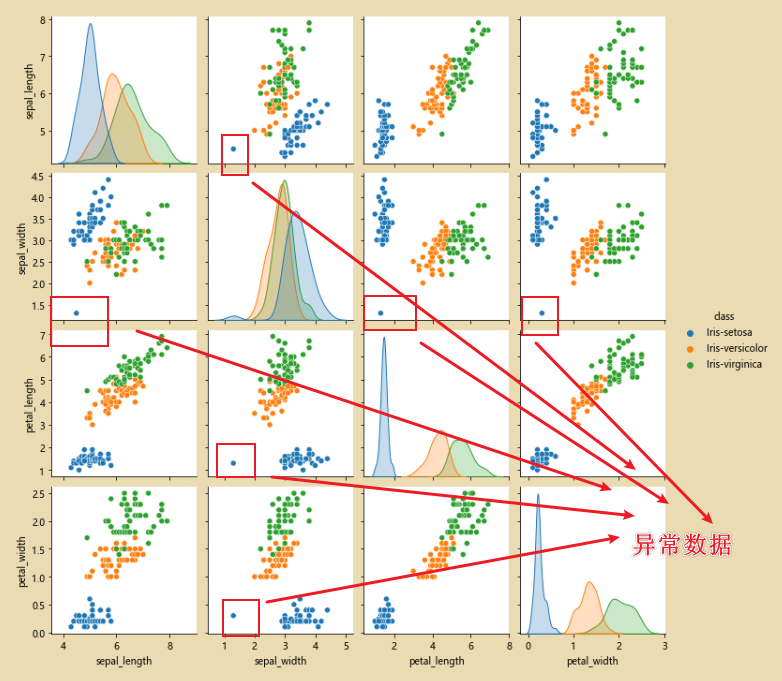
Análisis: De acuerdo con el color del punto anormal en la figura, los datos pertenecen a Iris-setosaDespués de encontrar los datos anormales, llénelos con el valor medio.
Encuentre esta anomalía cortando:
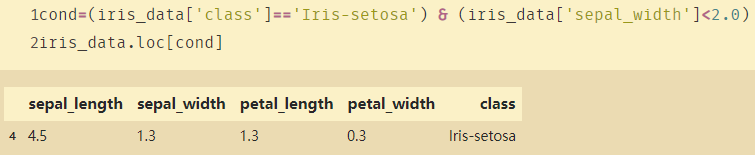
cond=(iris_data['class']=='Iris-setosa') & (iris_data['sepal_width']<2.0)
iris_data.loc[cond]
sepal_widthLa media a buscar a continuación :
Asigne valores atípicos a la media:
iris_data.loc[irissetosa & (cond),'sepal_width']=avgpetalwd
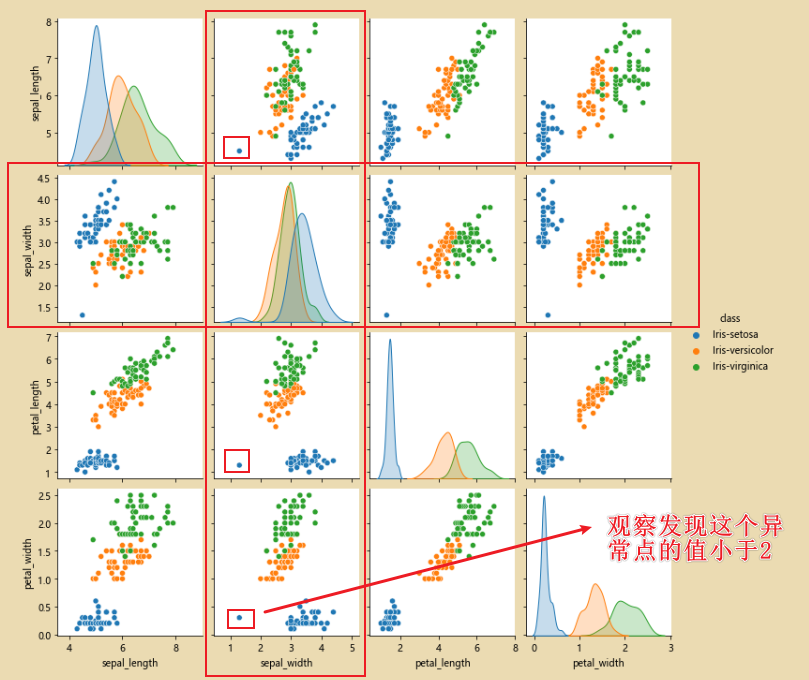
En este punto, no hay datos anormales:
observe la imagen, como se muestra a continuación, el punto anormal desaparece:
3. Árboles de decisión y bosques aleatorios
1. Dibujar gráficos
%matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sb
sb.pairplot(iris_data.dropna(),hue='class')
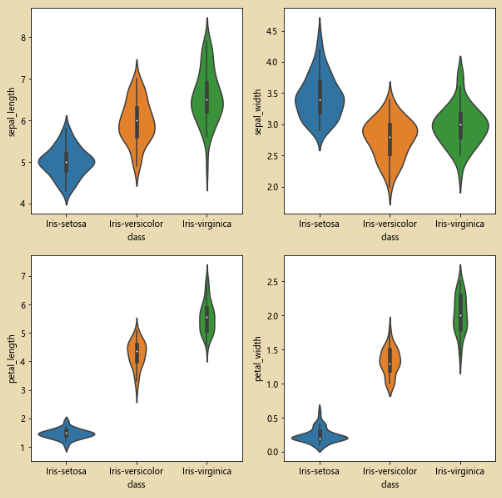
Trazar la trama del violín:
plt.figure(figsize=(10,10))
for column_index,column in enumerate(iris_data.columns):
if column == 'class':
continue
plt.subplot(2,2,column_index + 1)
sb.violinplot(x='class' ,y=column,data=iris_data)
2. Comparar parámetros
from sklearn.model_selection import train_test_split
all_inputs=iris_data[['sepal_length','sepal_width','petal_length','petal_width']].values
all_inputs
all_classes=iris_data['class'].values
(training_inputs,
testing_inputs,
training_classes,
testing_classes)=train_test_split(all_inputs,all_classes,train_size=0.75,random_state=1)
all_classes
training_inputs
# train_size=0.75 :75%作为训练集
from sklearn.tree import DecisionTreeClassifier
decision_tree_classifier=DecisionTreeClassifier()
decision_tree_classifier.fit(training_inputs,training_classes)
-
criterio: gini o entropía
Criterio: coeficiente de Gini o entropía
Ejemplo:criterion='gini'(indica que el coeficiente de Gini se utiliza para implementar un árbol de decisión) -
Divisor: mejor o aleatorio
Encuentre el mejor punto de división (mejor) o seleccione aleatoriamente (aleatorio) entre todas las características -
max_features : indica si es necesario agregar todas las características al árbol de decisión.
Tiene parámetros:None(all), log2, sqrt, N. Cuando la característica es menor de 50, generalmente se toman todas, y si es mayor de 50, se puede considerar log2 como parámetro. -
Este valor se puede ignorar cuando hay pocos datos o características de profundidad mínima
. Si hay muchas muestras de modelos, este valor se puede limitar adecuadamente -
max_ depth : la profundidad máxima del árbol
-
min_samples_split :
si el número de muestras de un nodo es menor quemin_samples_spliteso, no continuará intentando seleccionar la característica óptima para la división. Si la muestra no es grande, no necesita preocuparse por este valor. Si la magnitud de la muestra es muy grande, se recomienda aumentar este valor. -
min_samples_leaf : el número mínimo de muestras para los nodos hoja.
Este valor limita el número mínimo de muestras de nodos Tez. Si el número de nodos hoja es menor que el número de muestras, se podará junto con los nodos hermanos. Si el tamaño de la muestra no es grande, no necesita controlar este valor. Si es más grande, como 100,000, puede probar 5. -
El valor de min_weight_fraction_leaf
limita todos los pesos de muestra y los valores mínimos de los nodos hoja, si es menor a este valor, se podará junto con los nodos hermanos, el valor por defecto es 0, es decir, no se considera el problema de peso En términos generales ,
si tenemos más muestras con valores faltantes, o la categoría de distribución de la muestra del árbol de clasificación tiene una gran desviación, se introducirá el peso de la muestra, que es prestar atención a este valor. -
max_leaf_nodes :
al limitar el número máximo de nodos de hoja , se puede evitar el sobreajuste . El valor predeterminado es 'Ninguno', es decir, el número máximo de nodos de hoja no está limitado. Si se agrega el límite, el algoritmo reanudará el árbol de decisión óptimo dentro del número máximo de nodos hoja. Si no hay muchas funciones, este valor se puede ignorar, pero si hay muchos componentes de funciones, se pueden agregar restricciones y el valor específico se puede obtener a través de la validación cruzada. -
class_weight
especifica el peso de cada categoría de la muestra, principalmente para evitar demasiadas muestras de algunas categorías en el conjunto de entrenamiento, lo que resulta en que el árbol de decisiones de entrenamiento esté demasiado sesgado hacia estas categorías
Aquí puede especificar el peso de cada muestra usted mismo. Si usa 'equilibrado', el algoritmo calculará el peso usted mismo, el peso de la muestra correspondiente a la categoría con la menor cantidad de muestras será mayor -
El valor de min_inpurity_split
limita el crecimiento del árbol de decisión. Si la impureza (coeficiente de Gini, ganancia de información, error cuadrático medio, valor absoluto) de una orden dada es menor que este umbral
, el nodo ya no generará nodos secundarios, lo cual es un nodo hoja. -
train_size=0.75 : 75% como conjunto de entrenamiento
-
min_impurity_decrease : La pureza del nodo. Es posible que los nodos no se dividan lo suficientemente limpios. Esto conduce al problema de que los nodos de hoja no se pueden generar en la parte de generación posterior de los nodos de hoja o son demasiado delgados.
-
random_state : si su valor cambia, los datos obtenidos también cambiarán.
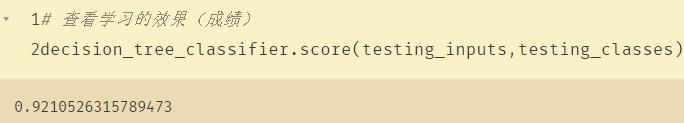
Ver el efecto del aprendizaje (grados):
decision_tree_classifier.score(testing_inputs,testing_classes)
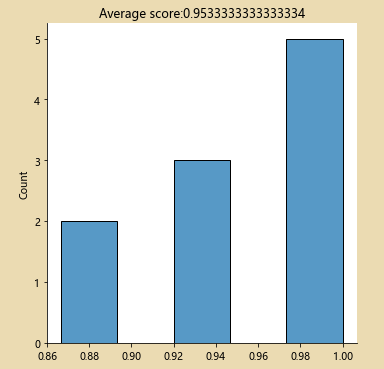
Echa un vistazo a los resultados después de la comparación cruzada:
from sklearn.model_selection import cross_val_score
import numpy as np
decision_tree_classifier=DecisionTreeClassifier()
cv_scores=cross_val_score(decision_tree_classifier,all_inputs,all_classes,cv=10)
print(cv_scores)
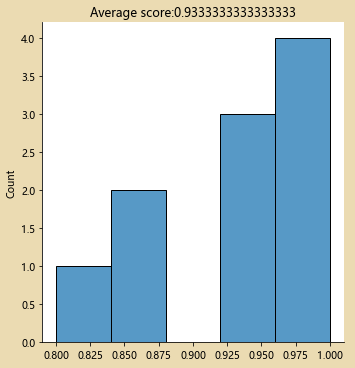
sb.displot(cv_scores)
plt.title('Average score:{}'.format(np.mean(cv_scores)))
decision_tree_classifier=DecisionTreeClassifier(max_depth=3) # max_depth:表示分类是否有区别,分几类
cv_scores=cross_val_score(decision_tree_classifier,all_inputs,all_classes,cv=10)
print(cv_scores)
sb.displot(cv_scores,kde=False)
plt.title('Average score:{}'.format(np.mean(cv_scores)))
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import StratifiedKFold
decision_tree_classifier=DecisionTreeClassifier()
parameter_grid={
'max_depth':[1,2,3,4,5],
'max_features':[1,2,3,4]}
# 将每个max_depth与每个max_features都组合一遍,找出其中最好的
skf=StratifiedKFold(n_splits=10)
cross_validation=skf.get_n_splits(all_inputs,all_classes)
grid_search=GridSearchCV(decision_tree_classifier,
param_grid=parameter_grid,
cv=cross_validation)
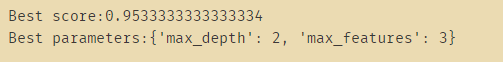
grid_search.fit(all_inputs,all_classes)
print('Best score:{}'.format(grid_search.best_score_)) # 得分最高
print('Best parameters:{}'.format(grid_search.best_params_)) # 得分最好
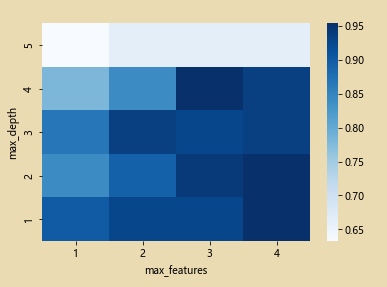
Dibujar un mapa de calor
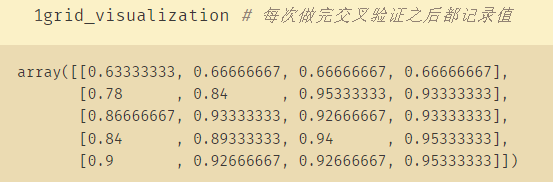
grid_visualization=[]
for grid_pair in grid_search.cv_results_['mean_test_score']:
grid_visualization.append(grid_pair)
grid_visualization=np.array(grid_visualization)
grid_visualization.shape=(5,4)
sb.heatmap(grid_visualization,cmap='Blues') # 蓝色
plt.xticks(np.arange(4)+0.5,grid_search.param_grid['max_features'])
plt.yticks(np.arange(5)+0.5,grid_search.param_grid['max_depth'][::-1])
plt.xlabel('max_features')
plt.ylabel('max_depth')
3. Llame a Graphviz para dibujar un árbol de decisión
decision_tree_classifier=grid_search.best_estimator_
decision_tree_classifier # 拿到得分最高的数据
import sklearn.tree as tree
from six import StringIO
with open('iris_dtc.dot','w') as out_file:
out_file=tree.export_graphviz(decision_tree_classifier,out_file=out_file)
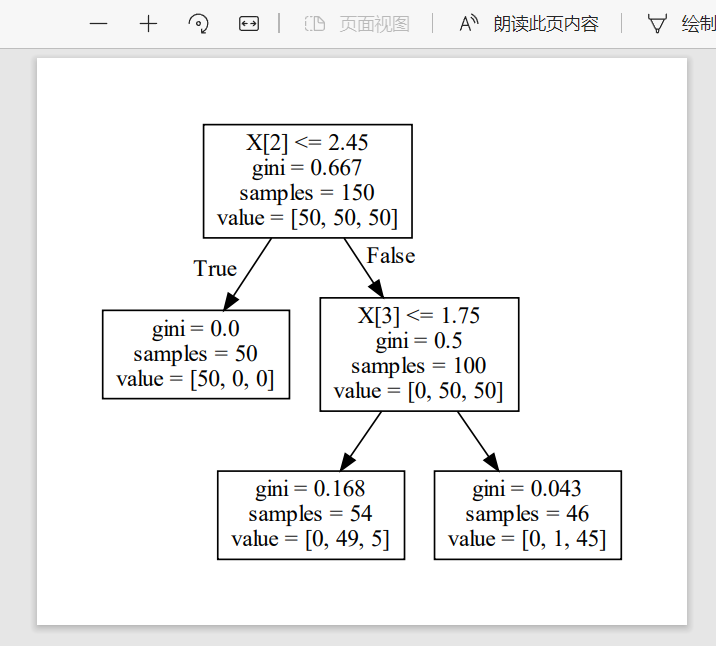
En este punto, se genera un documento: (se seleccionan los nodos y se escribe el resultado en el documento de puntos)

El contenido del documento:
digraph Tree {
node [shape=box] ;
0 [label="X[2] <= 2.45\ngini = 0.667\nsamples = 150\nvalue = [50, 50, 50]"] ;
1 [label="gini = 0.0\nsamples = 50\nvalue = [50, 0, 0]"] ;
0 -> 1 [labeldistance=2.5, labelangle=45, headlabel="True"] ;
2 [label="X[3] <= 1.75\ngini = 0.5\nsamples = 100\nvalue = [0, 50, 50]"] ;
0 -> 2 [labeldistance=2.5, labelangle=-45, headlabel="False"] ;
3 [label="gini = 0.168\nsamples = 54\nvalue = [0, 49, 5]"] ;
2 -> 3 ;
4 [label="gini = 0.043\nsamples = 46\nvalue = [0, 1, 45]"] ;
2 -> 4 ;
}
Utilice Graphviz para dibujar una imagen de árbol de decisión. Tenga en cuenta que primero debe instalar Graphviz, configurar las variables de entorno y abrirlo dot.exe.
En la línea de comando, vaya al iris_dtc.dotdirectorio donde se encuentra el archivo y luego llame al siguiente comando

dot -Tpdf iris_dtc.dot -o irisdt.pdf
En este punto, se genera el archivo pdf que necesitamos: