文章目录
前言
Unet++是2018年的论文,这篇论文主要是针对生物医学图像的分割,在以下四个分割任务做了实验:
1、胸部低剂量CT扫描中的结节分割
2、显微镜图像中的细胞核分割
3、腹部CT扫描中的肝脏分割
4、结肠镜检查中的息肉分割
概述
对以前的论文做了一个简短的总结:对于分割任务,都会有一个共识,就是skip connection。目前对自然图像的分割效果蛮好,对生物医学图像的分割一般。(自我理解)主要的原因可能还是(1)数据集的量(2)生物医学图像相较于自然图像来说,本身分割难度大,体现在生物图像中分割目标边界模糊、变形一类的复杂情况。
解决的问题?
对生物医学图像的分割,具体的已经在上面列出来了。
使用的方法?
Unet的增强版,灵感来源于DenseNet。但实际上,在此之前有一个思想是一样的工作,可以说是Unet的工作与其如出一辙,根据任务进行了调整而已。
深度监督
result
想对于UNet和wide UNet(wide IOU是相对与UNet的参数增加,使其与Unet++在参数上相差无几,减少相互对比中的不一样的条件)各自有3.9和3.4的平均IOU的提升。
More
文中提到可以将Unet++作为Mask rcnn的backbone architecture。但是文中没有给出具体做法
加中间的Dense block所基于的一个假设:让received encoder feature maps和the corresponding decoder feature maps are semantically similar,这样会使优化器更好的优化。
细节部分
1、一张结构图 & 解释:

其中的蓝色和绿色部分就是Unet++相对UNet添加的部分。在右侧有L1, L2等,这些是Unet++对网络不同深度的设置。其中绿色的箭头表示上采样,同UNet,黑色的下箭头表示下采样,同UNet。
蓝色箭头则表示skip connection,每一个水平层就是非常标准的DenseNet的结构。每个原型单元代表了卷积+激活函数一系列的操作。
对其中的等式进行解释:


2、loss:binary cross-entropy和dice coefficient两部分的组合。以下的loss应用到每一个不同层次的输出。
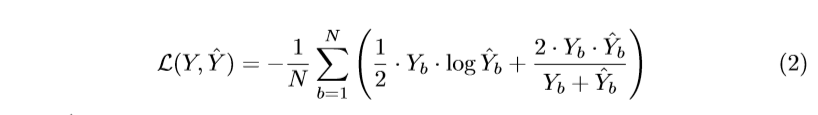
3、深度监督:
上述的每个X0,j(j!=0)X^{0,j(j!=0)}X
0,j(j!=0)
的loss其实就是深度监督的做法,其实就在最后一个X0,?X^{0, ?}X
0,?
做loss也可以,深度监督可以说是一种小trick。
文中对深度监督的描述:使用深度监督是为了可以让模型可以在两种方式下有选择的进行操作:(1)Accurate mode:做上述的所有分割分支做一个平均再得到最后输出结果(2)Fast mode:从其中的所有的分支输出选择一个作为输出。
Another:Deep supervised enables model pruning and improves or in the worst case achieves comparable performance to using only one loss layer.
result
1、几个数据集的信息

2、一些效果图


3、效果的对比
其中colon polyp(结肠息肉)是最难有好效果。而且表明对于不同的数据集,不同深度的网络对其能做到的程度时不一样的。再cell nuclei和colon polyp中加上了深度监督反而没有没有加深度监督的效果好,文中也给出了解释:This is because polyps and liver appear at varying scales in video frames and CT slices。

