有医院的朋友,需要帮忙完成一个图像分割的任务,提供了一些数据,看了下数据,灰度图,觉得设计特征再做分割太麻烦。直接整神经网络吧。不用费神设计特征,省事,毕竟只是帮个忙而已。
1. 查找方案
显然,这个任务,早有前人做过无数次了,这么热点的领域,简直一搜一大把。搜索结果,是用 u-net 做医学图像分割的较多,于是决定使用u-net。关于FCN的介绍,看这个博客吧,本文着重于代码实现!
考虑到任务的价值和撸代码的便利性,决定使用keras,毕竟这只是一个任务。
使用kears 做图像分割,CSDN 有一篇很容易搜到的文章(文章链接在本文末尾),还附了github地址,简直得来全不费工夫,立马下下来,准备直接换数据跑完代码收工。显然… 我还是太年轻。
2. 坑
原作者的代码的测试是二分类的,但我要跑的数据与标记如下:
注:左边原图,右边mask,三类,mask=0,128,255 各为一类。
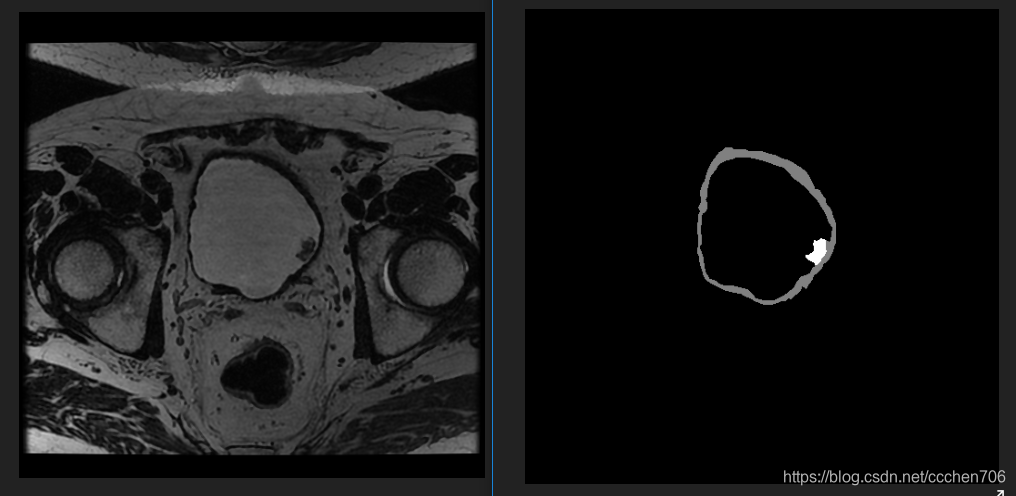
数据是多分类的,从此埋下了深深地祸根!
先来一个个看吧。
-
ValueError: Error when checking target: expected conv2d_24 to have 4 dimensions, but got array with shape (2, 65536, 3)
这个问题其实见得比较多了,神经网络图像初学时比较容易出现类似的问题,于是检查代码,根据提示定位到如下代码段:
def adjustData(img,mask,flag_multi_class,num_class):
if(flag_multi_class):
img = img / 255
mask = mask[:,:,:,0] if(len(mask.shape) == 4) else mask[:,:,0]
new_mask = np.zeros(mask.shape + (num_class,))
for i in range(num_class):
#for one pixel in the image, find the class in mask and convert it into one-hot vector
#index = np.where(mask == i)
#index_mask = (index[0],index[1],index[2],np.zeros(len(index[0]),dtype = np.int64) + i) if (len(mask.shape) == 4) else (index[0],index[1],np.zeros(len(index[0]),dtype = np.int64) + i)
#new_mask[index_mask] = 1
new_mask[mask == i,i] = 1
new_mask = np.reshape(new_mask,(new_mask.shape[0],new_mask.shape[1]*new_mask.shape[2],new_mask.shape[3])) if flag_multi_class else np.reshape(new_mask,(new_mask.shape[0]*new_mask.shape[1],new_mask.shape[2]))
mask = new_mask
elif(np.max(img) > 1):
img = img / 255
mask = mask /255
mask[mask > 0.5] = 1
mask[mask <= 0.5] = 0
return (img,mask)
由于是多分类,设置flag_multi_class=True,num_class=3,可以看到代码将走向前段,这样mask将会被reshape成(65536,3),至于前面的2 是 batch_size,此时明确了label的形状,就证明网络输出层与label不匹配导致错误,于是查看模型代码。
conv10 = Conv2D(1, 1, activation='sigmoid')(conv9) #其实conv2d_24 就是这里的conv10
model = Model(input=inputs, output=conv10)
model.compile(optimizer=Adam(lr=1e-4), loss='binary_crossentropy', metrics=['accuracy'])
根据model的输出形状可推出 label形状应该是(2,256,256,1)。而我们提供的mask是(2,256*256,3),所以报错。
明确了错误就很好修改了!
for i in range(num_class):
new_mask[mask == i,i] = 1 #去掉了注释
这段代码是指mask中有与类型相等的值时,添加为这层的label。 我的mask怎么会是0,1,2这种呢,而且一般的mask都是0,128,256这种易于区分的值啊…尴尬
自己撸代码:
def adjustData(img,mask,flag_multi_class,num_class):
if(flag_multi_class):
img = img / 255.
mask = mask[:,:,:,0] if(len(mask.shape) == 4) else mask[:,:,0]
new_mask = np.zeros(mask.shape + (num_class,))
new_mask[mask == 0,0] = 1
new_mask[mask == 128, 1] = 1
new_mask[mask > 200, 2] = 1
mask = new_mask
# print('new 0 :',np.sum(mask==1))
# print('new 128 :', np.sum(mask ==2))
# print('new 255 :', np.sum(mask == 3))
# print('new sum :',np.sum(mask==1)+np.sum(mask==2)+np.sum(mask==3))
elif(np.max(img) > 1):
img = img / 255.
mask = mask /255.
mask[mask > 0.5] = 1
mask[mask <= 0.5] = 0
return (img,mask)
这个修改不再reshape mask,并且将mask处理成one-hot编码。即按照数据集中mask的值 0,128,256 进行分类叠加。如果mask图像相应的值是 0
,那么,处理后的值为[1,0,0],当mask=128时为[0,1,0],255时为[0,0,1]。这样就完成了标记数据的转换。处理后的label shape 为(2,256,256,3),与网络输出还有区别网络为(2,256,256,1)所以我们要对网络再进行改进。
conv10 = Conv2D(3, 1, activation='sigmoid')(conv9) #修改原来的1为3,此时网络有3通道输出。
model = Model(input=inputs, output=conv10)
model.compile(optimizer=Adam(lr=1e-4), loss='binary_crossentropy', metrics=['accuracy'])
这样就能完成多分类的训练了。
后面发现,训练了多个epoch(40次左右),输出图像仍为纯白,再次回顾网络结构,与loss函数。发现居然用的binary_crossentropy,坑了个爹的,只能用于二分类。修改成categorical_crossentropy 又不收敛,唉,本来不想花时间的东西,居然已经弄了几个小时。
至此,已经发现要改的东西比较多。老老实实再继续吧弄吧。
3. 目标很明确,端到端多类型图像分割!
后面借鉴github的代码做了端到端的多分类,将在(二)里面介绍,考虑用u-net做多分类的可以看下,github地址:Keras-u-net,欢迎star。
注:分析代码原文地址:文章链接
端到端多分类代码地址:Keras-u-net