04-SNAP处理Sentinel-2 L2A级数据(二)
(原创文章,转载请注明来源!)
前言
03篇中谈到了利用SNAP对Sentinel-2进行的一些预处理,但是还有一些预处理没有谈到。这篇是为下篇的Sentinel-2监督分类做准备工作,主要是特征工程,或者说构造分类特征。这篇博客会介绍一下操作波段指数特征计算,灰度纹理特征提取,主成分变换,波段叠加等操作。在计算之前希望,你能把SNAP相关的插件装上(建议全部装上),否则下面的操作可能进行不了。可以在tools—>Plugins打开插件的相关操作面板,如下图所示
数据集
03篇用的数据集有比较多的云量,这次我们选择一个云量较少的Sentinel-2数据进行处理。下面的数据集,我已经进行如下处理:大气校正,重采样,裁剪,镶嵌,最终得到一幅覆盖整个崇明岛、分辨率为10m的无云Sentinel-2影像。(我还想强调一下,Sentinel-2 由于存在三种不同分辨率的波段,在大气校正后,通常的第一步操作都是重采样,因为SNAP(或者别的软件)相关操作都要求波段的分辨率(影像大小)是相同的才能进行。 除了重采样这种方式外,你还可以使用SNAP中的超分辨率插件Sen2Res获取分辨率一样(10m)的波段(见03篇)。
这里使用的是重采样工具获取相同的分辨率,因为超分辨率合成需要耗费比较多的时间。不过如果想获得更好的效果,并且时间充裕的话,推荐使用超分辨率合成工具处理。这里仅仅用于介绍SNAP的一些操作,就不那么讲究了。
本文使用的SNAP版本为Windows系统的SNAP V6.0,电脑运行内存为16G。
本文的数据集来自以下两幅影像(上海市2019年12月10日的无云影像):
S2B_MSIL1C_20191210T024109_N0208_R089_T51SUR_20191210T041040.SAFE
S2B_MSIL1C_20191210T024109_N0208_R089_T51RUQ_20191210T041040.SAFE
处理的结果数据集:
chongming_mosaic.dim
本文涉及的数据见百度云盘链接(数据集说明见云盘中的说明.txt):
链接:https://pan.baidu.com/s/1WfLrhjEFz3sT66StQ8pXZQ
提取码:1pya
波段指数计算
植被指数NDVI计算
法1----使用NDVI Processsor工具
这应该是这简单的方式了,因为只需要动几下鼠标即可(不用写代数式计算,并且不用你考虑计算时的逻辑,如分母为零、类型转换等问题)。选择Optical—>Thematic Land Processing—>Vegetation Radiometric Indices—>NDVI Processor。
此外,还可以在Vegetation Radiometric Indices(植被辐射指数)找到许多定义好的植被指数计算工具,更多的说明以及算法请查看帮助文档。
I/O Parameters(输入/输出参数)保留默即可
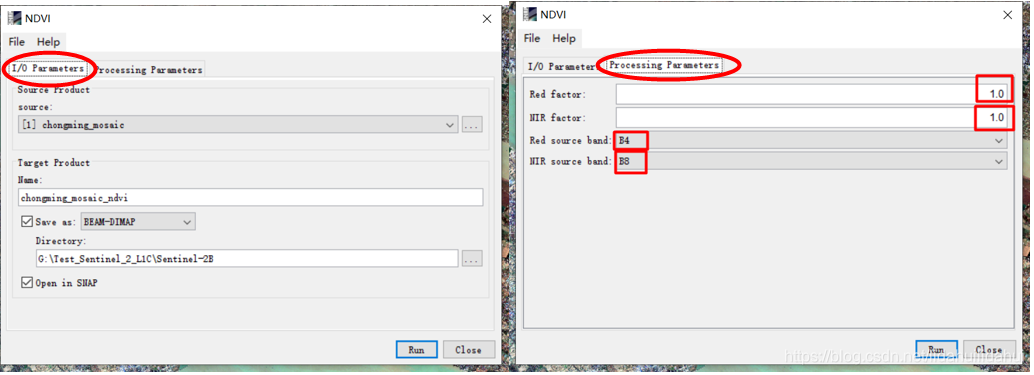
我想对Processing Parameters参数面板的Red factor和NIR factor,简单说明一下,Red factor是红光波段的比例调节因子,NIR是近红外波段的比例调节因子。其它的指数计算参数面板中,也会有类似的比例因子。以前在一些论文中会采用比例因子对NDVI的计算进行改进(或者说是修正),以获得更好的效果。如果你想获得最好的比例因子,通常都需要有实测植被等地物的光谱曲线数据。或者你可以点击Help,看一下NDVI的算法(计算的表达式)。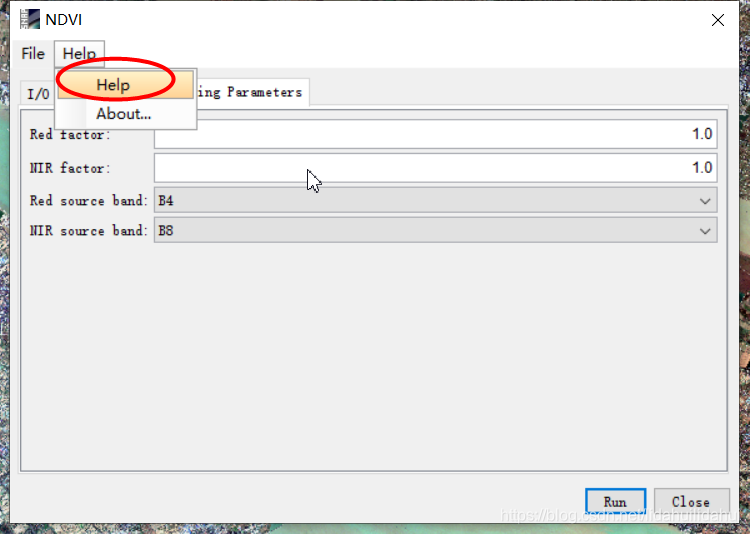
第一个红色框中就是其计算NDVI时的表达式:IR_facctor(这里应该是NIR_factor)。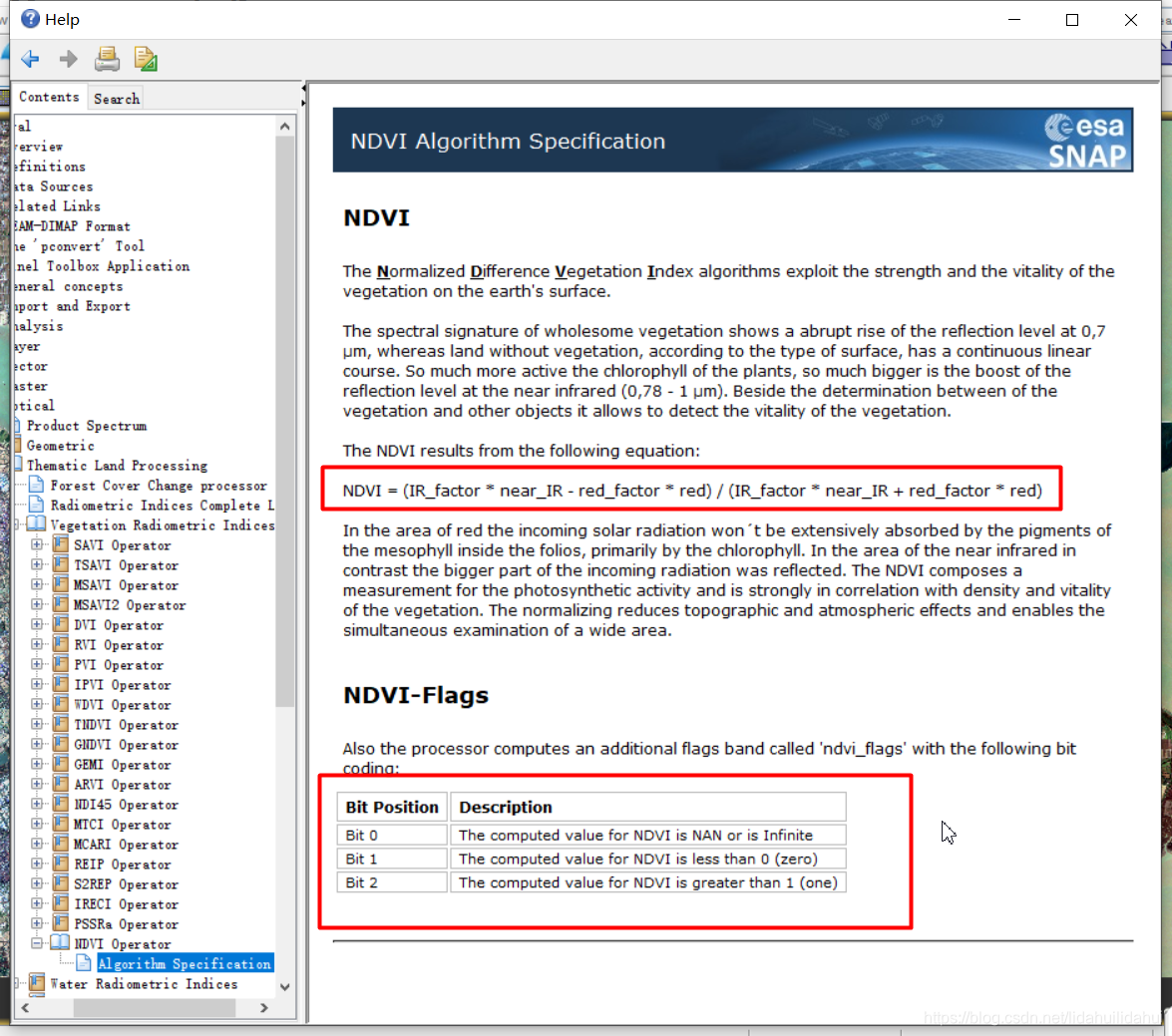
第二个红色框部分是对计算结果NDVI_flag波段的说明。SNAP中许多操作都会有一个flag标志波段,来说明其结果的质量,通常这个值是0-1之间的概率,但有时有不是,例如这里的计算结果。通常可以利用这个来创建掩膜文件(通过波段运算Band Maths工具可以实现)。
点击"Run",执行计算,我们再来看其结果,完成后的提示界面,可以看到计算的速度还是比较快的。完成后可以关闭NDVI Processor的界面。
看下结果,再来解释ndvi_flags波段的意义(建议点开Masks文件夹,以便看下掩膜文件名称)。效果如图:
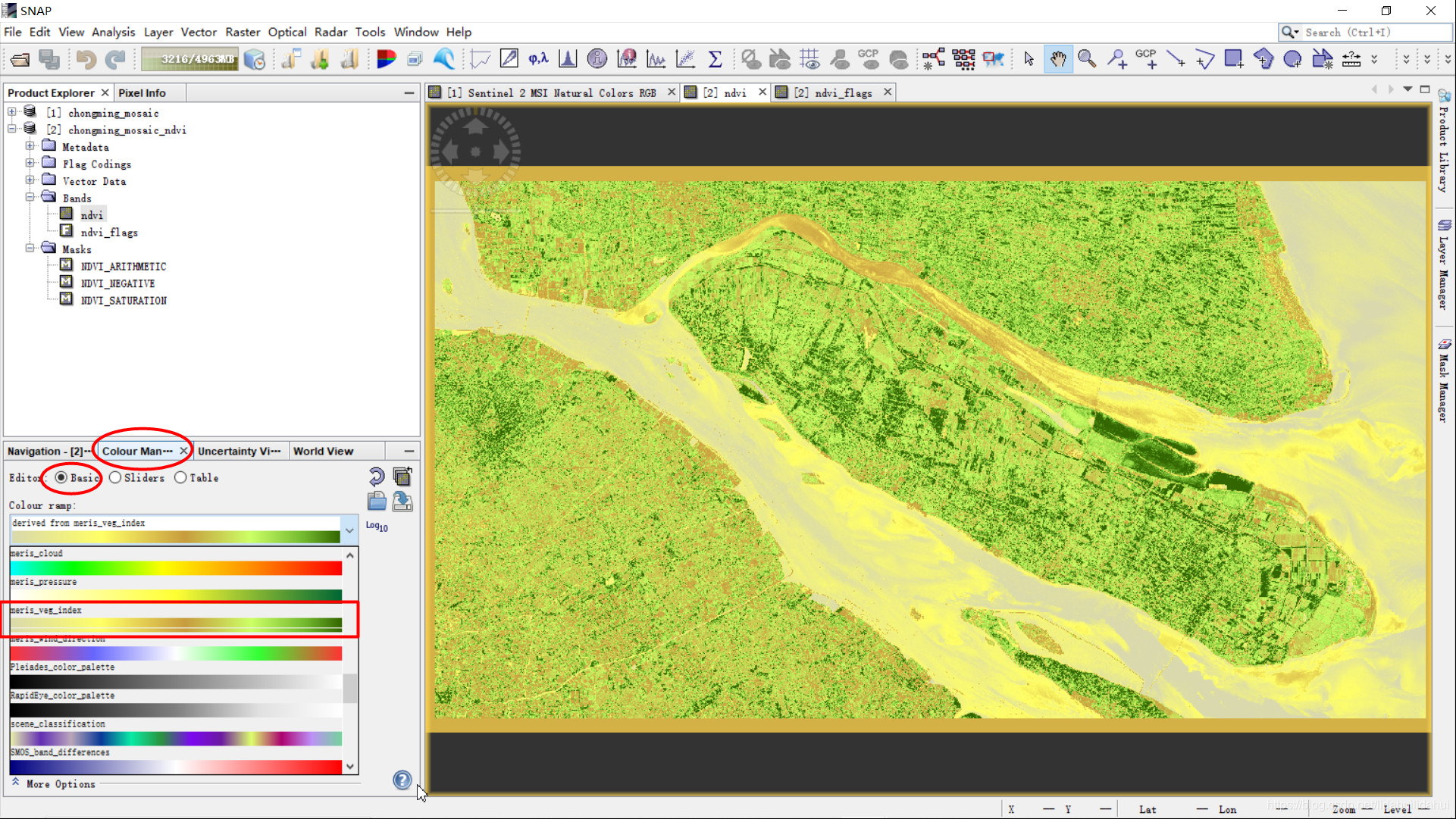
对ndvi波段更换伪彩色带,使其更直观显示植被状况(尽管不能绝对准确显示出植被的分布)。这里使用meris_veg_index色带,SNAP自带的植被指数色带。通常意义下,颜色越绿表示其是植被的概率越高,植被植被健康状况越好(但是这不是绝对的)。
 水平分屏显示效果:
水平分屏显示效果:
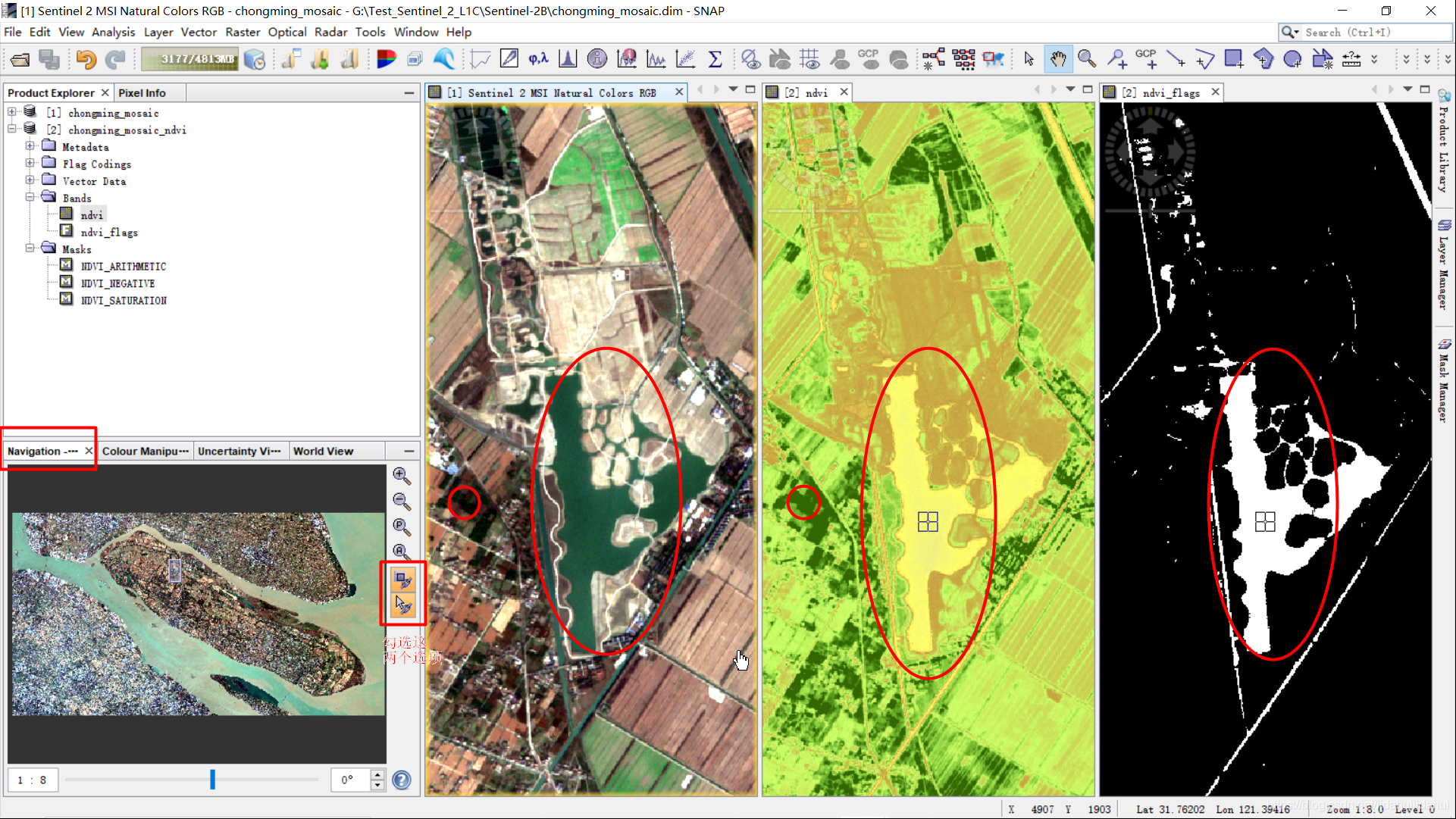
小红色圈中墨绿色区域(看起来像黑色)竟然是植被,反而RGB上绿色显示的是水体(尽管眼睛目视能分辨清楚),有意思的是ndvi_flags标志波段和水体区域表现出几乎一致的相似性,某种意义上或许可以利用flag波段来提取水体。当然如果你对水体提取更感兴趣的话,或许计算水体指数能获得更好的效果。
好了,现在来解释一下ndvi_flags标志波段的值的意义。
可以关闭前面显示的RGB影像,只保留ndvi和ndvi_flags波段显示。
鼠标左键单击一下波段名ndvi_flags,然后选择I标志图标查看该栅格波段的元数据信息,我比较关心的是其数值类型,考虑到其是一种编码值(Help中所示),应该是整型,确实如此,为int32。如果你查看ndvi波段的元数据信息,看到的应该是float32的类型:
 选择Pixel Info面板查看ndvi和ndvi_flags波段的值。注意观察下图红色框部分的值的变化。
选择Pixel Info面板查看ndvi和ndvi_flags波段的值。注意观察下图红色框部分的值的变化。
这里需要解释下图最后一个框中Flags标志字段的意义:
“NDVI_ARITHMETIC”指的是“An arithmetic exception occurred.”。这和NDVI算法Help中NDVI_flags中bit 0是对应的,指的是可导致算术出现异常的数值例如背景(NAN)和无穷大值(Infinite)。
"NDVI_NEGATIVE"指的是“NDVI value is too low.” 这和NDVI算法Help中NDVI_flags中bit 1是对应的,指的NDVI中小于0的值。
"NDVI_NEGATIVE"指的是“NDVI value is too low.” 这和NDVI算法Help中NDVI_flags中bit 1是对应的,指的NDVI中小于0的值,负值。这里的"0"相当于NDVI低值分界点对应阈值。
"NDVI_SATURATION"指的是“NDVI value is too high.” 这和NDVI算法Help中NDVI_flags中bit 2是对应的,指的NDVI中大于1的值,饱和值。这里的“1”相当于NDVI高值分界点对应阈值。
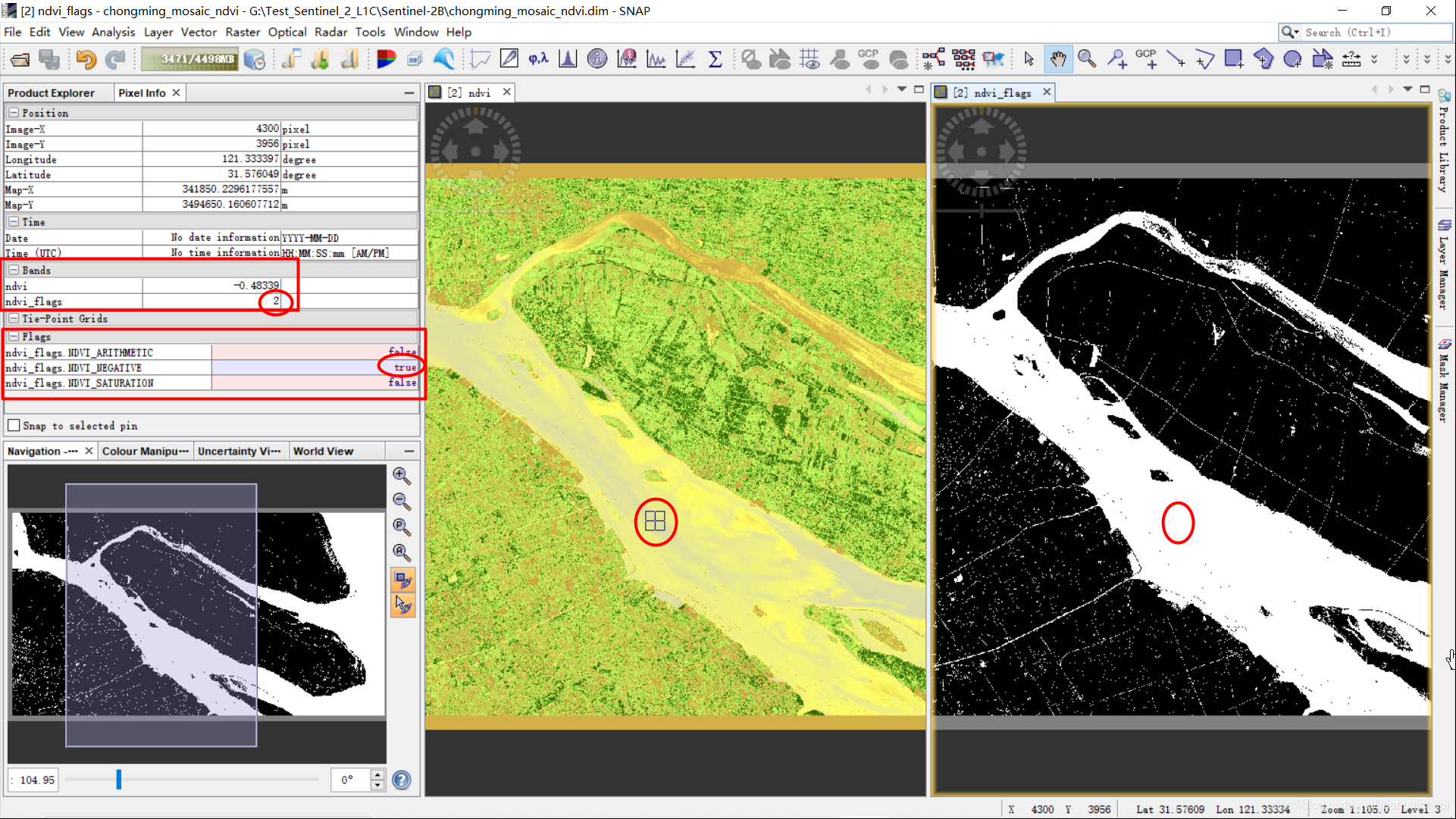
例如这里鼠标移动到水体部分,可以看到ndvi的是负值,ndvi_flags为2,指示其为ndvi_flags.NDVI_NEGATIVE,该值显示为true,其它两个标记显示为false。
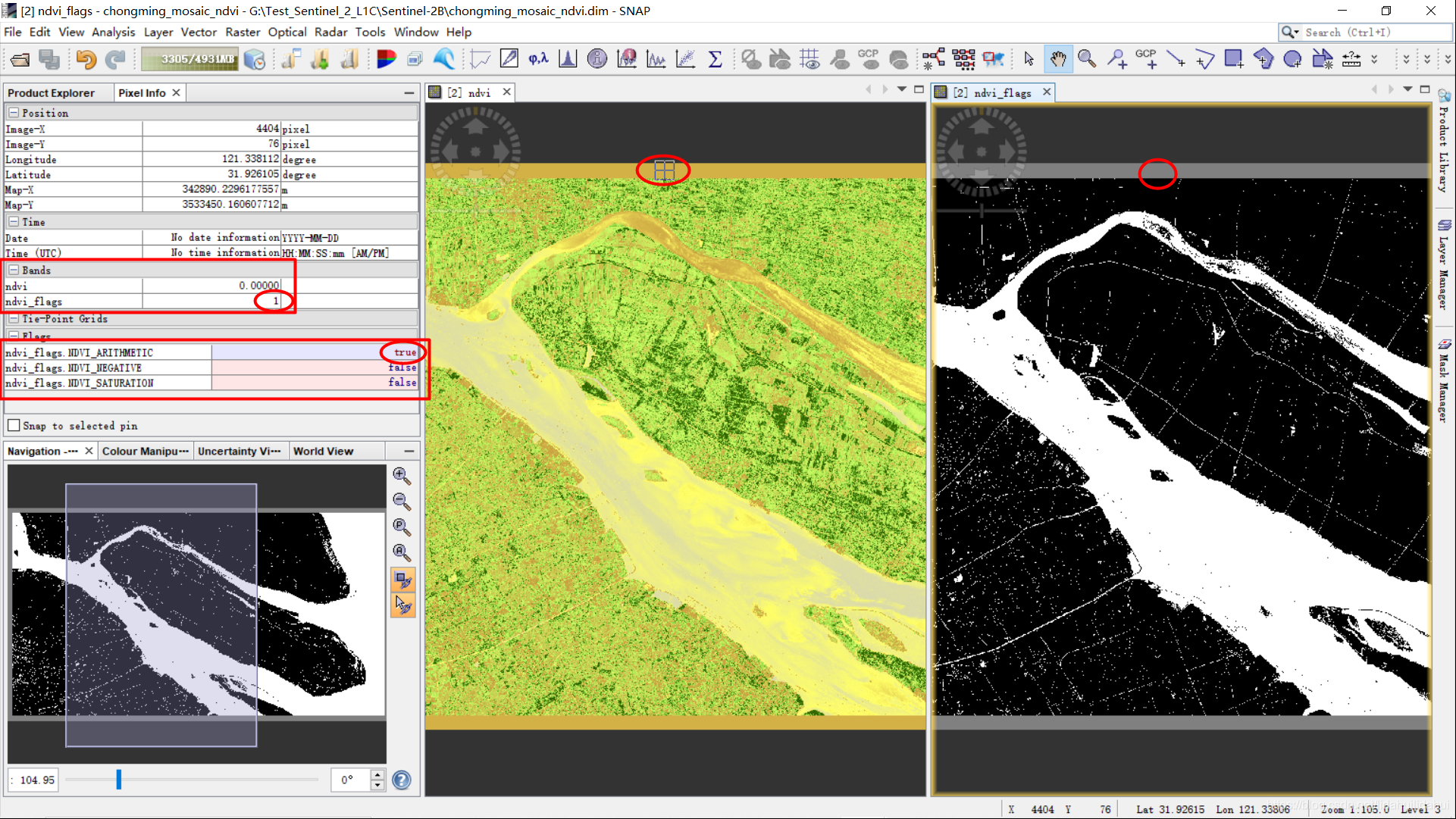
再如,我将鼠标移至背景值处。可以看到:
ndvi_flags为1,指示其为ndvi_flags.NDVI_ARITHMETIC,该值显示为true,其它两个标记显示为false。
 当其在正常范围时,ndvi_flags取值为0,三个ndvi_flags字段均为false。当ndvi_flags取值为3,ndvi_flags.NDVI_SATURATION取值为true,另两个取值为false,表示大于1的NDVI的值。
当其在正常范围时,ndvi_flags取值为0,三个ndvi_flags字段均为false。当ndvi_flags取值为3,ndvi_flags.NDVI_SATURATION取值为true,另两个取值为false,表示大于1的NDVI的值。
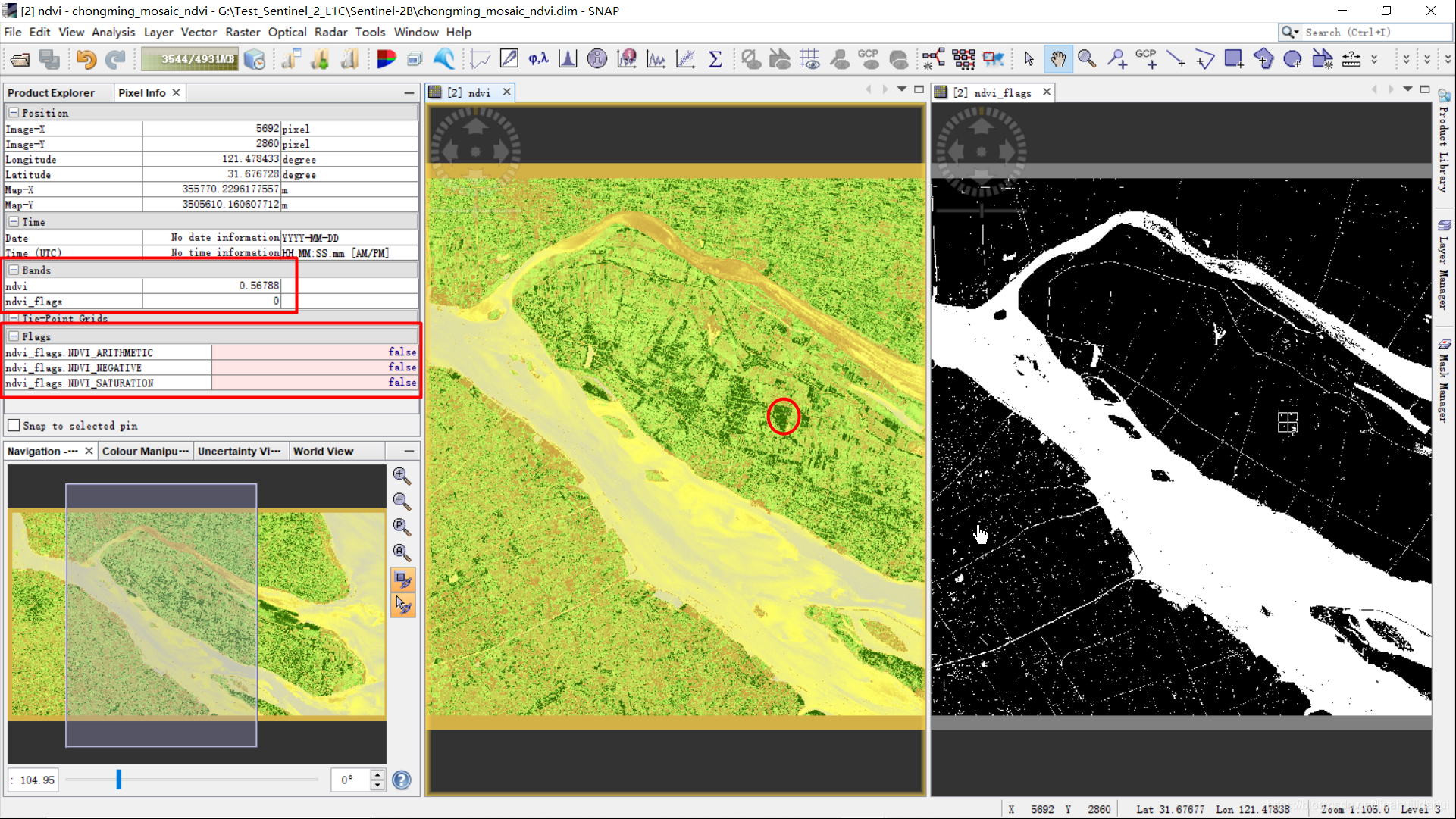
也就说ndvi_flag的黑色部分(0值)是正常值,白色部分是某种意义的异常值(或是算术上的异常或是阈值下的异常值)。
其它指数计算结果的标志波段解释是相类似的。我后面不再解释其它波段指数的计算结果。
另外,这里0和1阈值可以通过snappy包处理更换为其他数值。
法2----使用Band Maths工具
使用波段代数(Band Maths)工具可以自定义计算各种代数表达式(数值表达式,关系表达式、逻辑表达式等)。这个工具使用在03篇去云掩膜,波段运算创建掩膜操作提到,我不再重复赘述这个操作面板的参数说明。
关闭前面的ndvi和ndvi_flags波段。选中数据集chongming_mosaic重新打开RGB真彩色合成图。
两种方式打开Band Maths:
方式1: 方式2菜单栏启动:
方式2菜单栏启动:
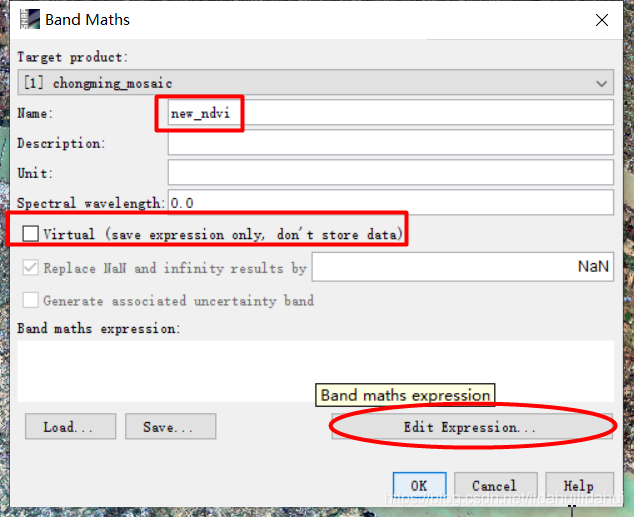
 打开Bands Maths 面板后,Name字段改为new_ndvi,把Virtual前面的勾去掉(使其保存到数据中,而不是作为虚拟波段存储在内存中)。
打开Bands Maths 面板后,Name字段改为new_ndvi,把Virtual前面的勾去掉(使其保存到数据中,而不是作为虚拟波段存储在内存中)。
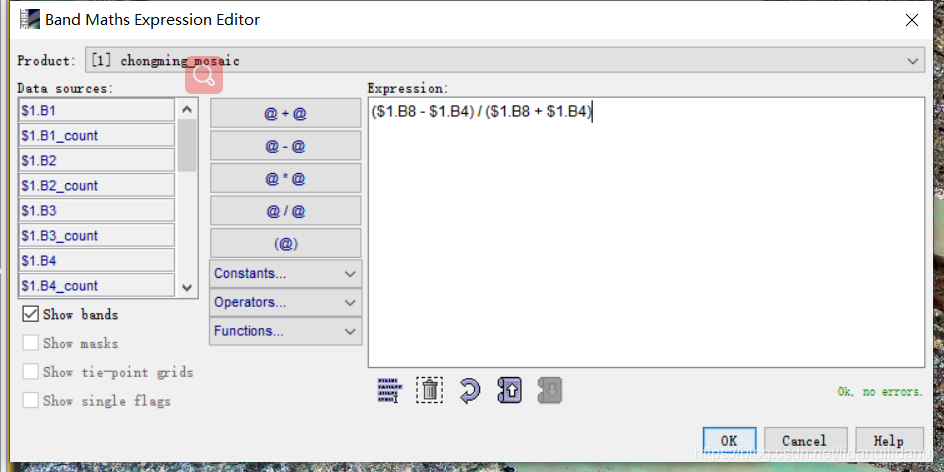
点击Edit Expression。输入如下表达式(注意观察右下角的绿色的"Ok,no rrors"字眼提示,确保表达式无误),点击OK:
点击“OK”.,执行计算。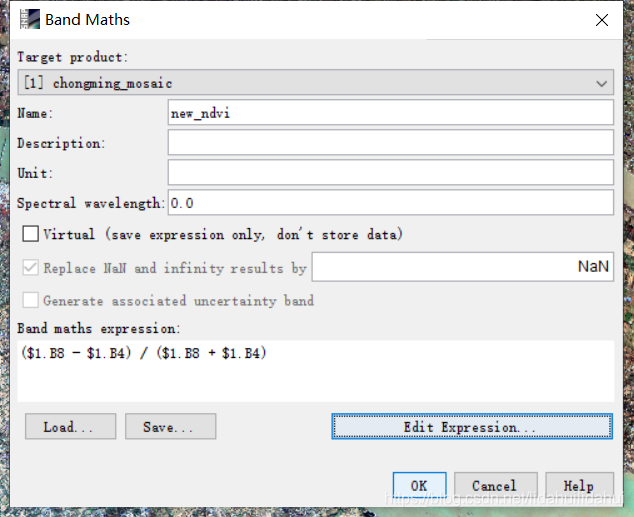
结果,可以看到数据集中多了一个波段:
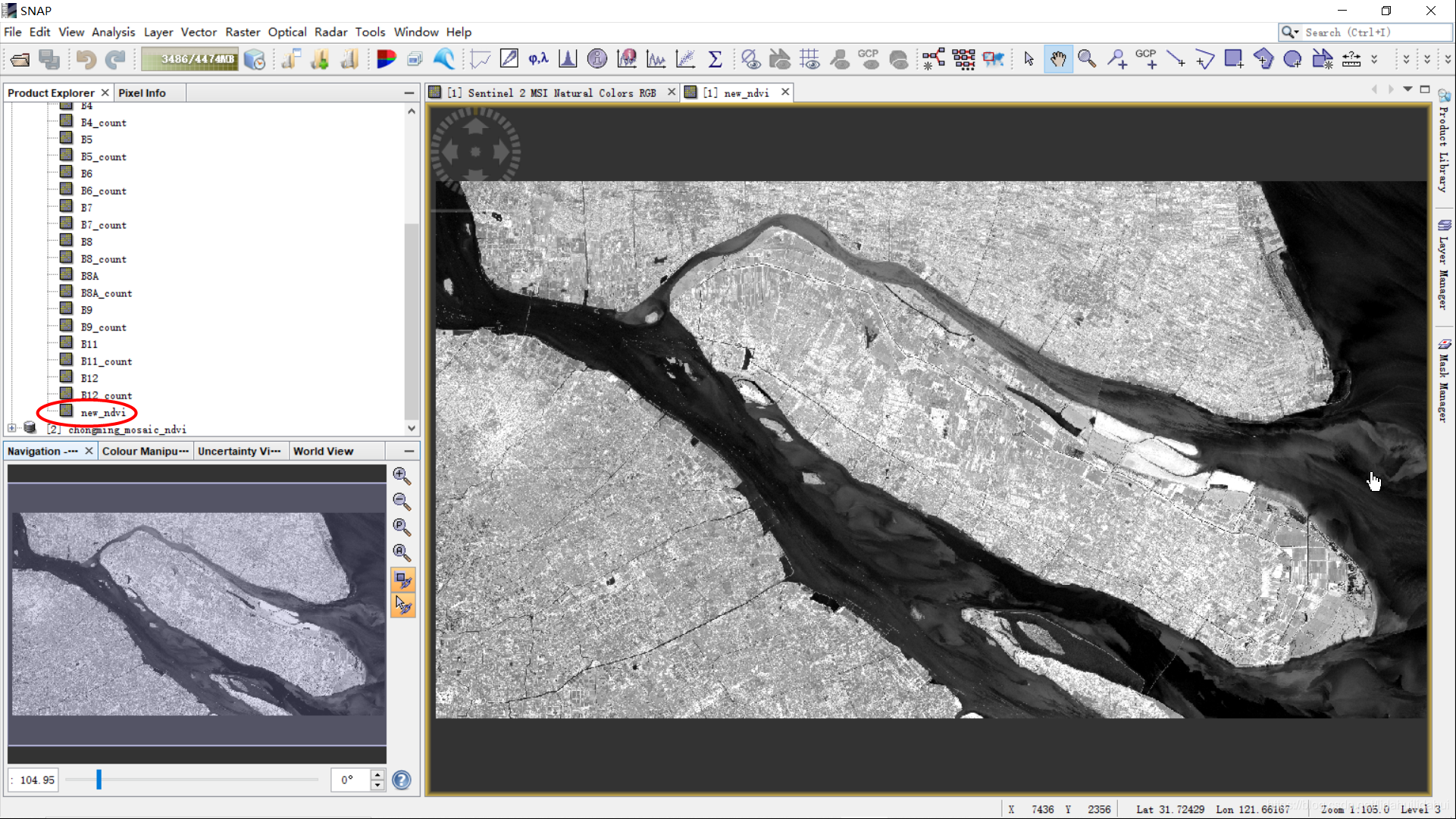
相比其他软件来说,SNAP计算NDVI的等表达式是非常直观的,不要你考虑异常值的情况,系统已经代替了你考虑到这些异常的情况。
法1(NDVI Processor)与法2(Band Maths)的区别:
- 法1计算的结果是单独作为新一个数据集(chongming_mosaic_ndvi)存储起来的,法2是存储在原来的数据集(chongming_mosaic)中
- 法1计算的结果含有flags标志波段,并且带有掩膜文件。而波段运算结果是没有这些文件。而法2中是没有这些文件的。
植被红边位置指数REPI计算
Sentinel-2在红光与近红外波段的红边区域(670—760 nm)内具有3个波段,这里利用它来计算红边波段被认为对植被分类具有更好的分辨能力【1】。这里使用欧空局经过测试确定得到的红边位置指数REPI(Red-Edge Position Index)计算工具(特别针对Sentinel-2而设计的)来计算。红边波段以及短波红外波段都是欧空局等研究机构经过大量研究和实践而设定(具体频谱范围,数量多少都是有讲究的)。既然如此,提取其用植被分类等地物分类应该具有不错的效果。这里利用S2REP Processor来计算(这个是针对Sentinel-2而设计,对于Landsat等卫星可能不一定合适)。
S2REP Processor工具位置, 如图:
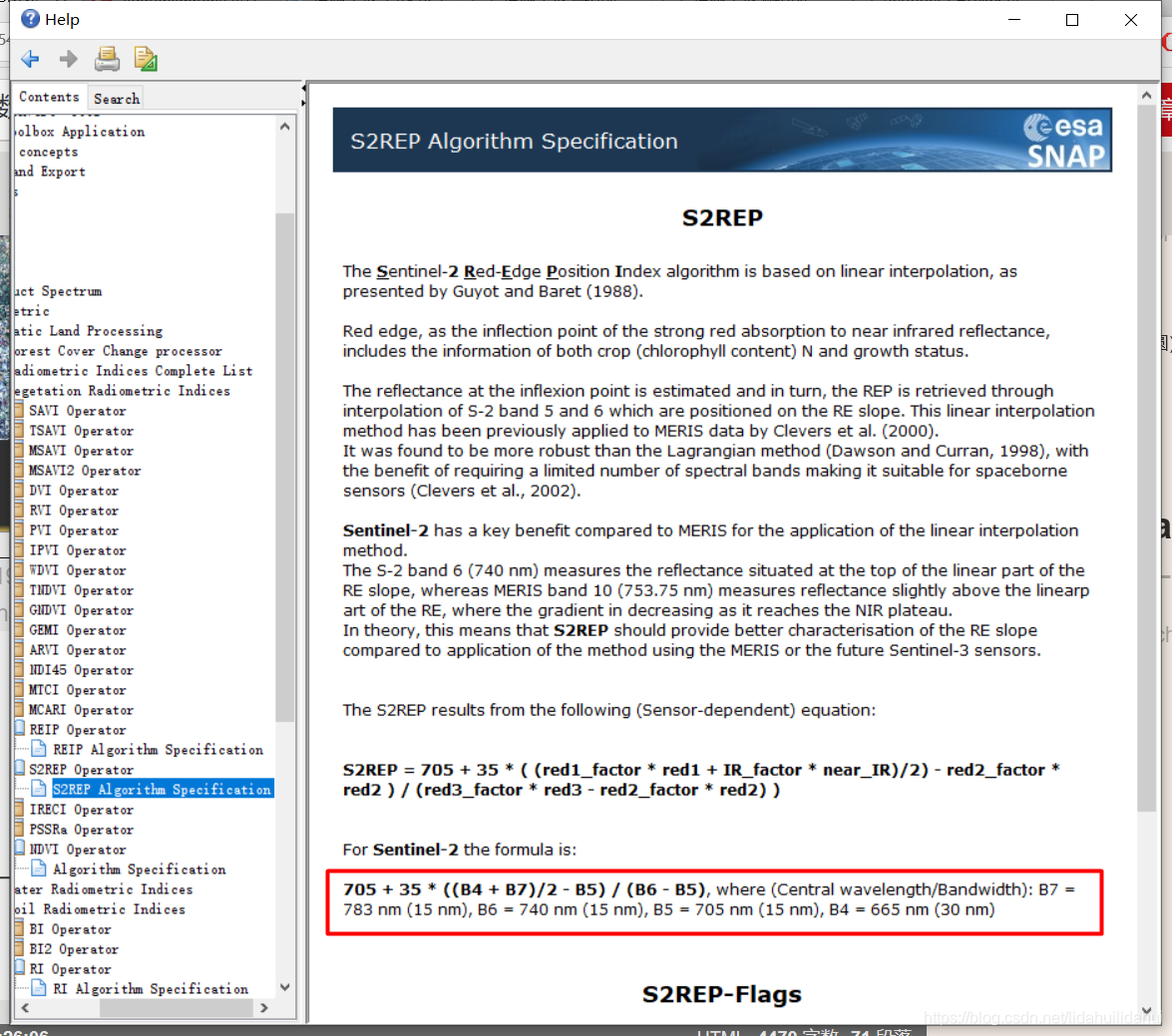
 具体算法可以查看Help中的说明:
具体算法可以查看Help中的说明:
参数面板设置:
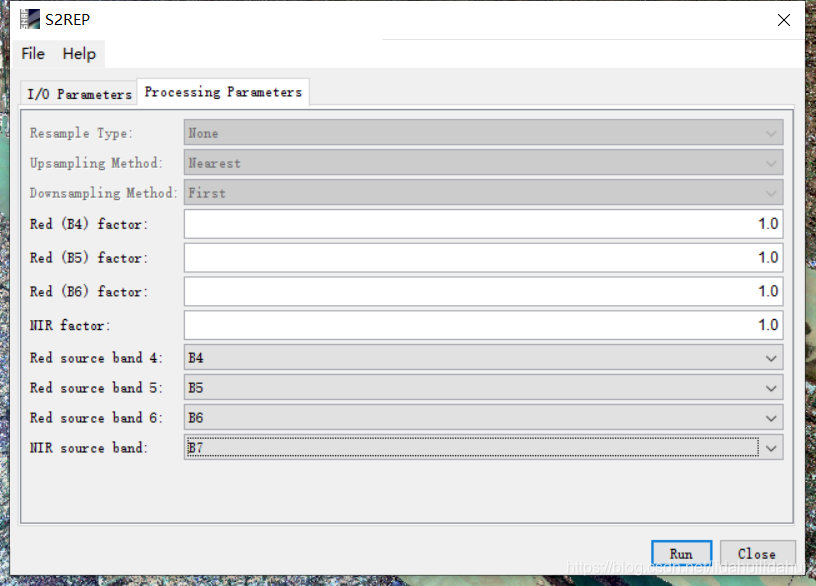
需要注意的是,对于Sentinel-2来说,这里的NIR波段选择的是B7波段,而不是B8,参见Help中的说明
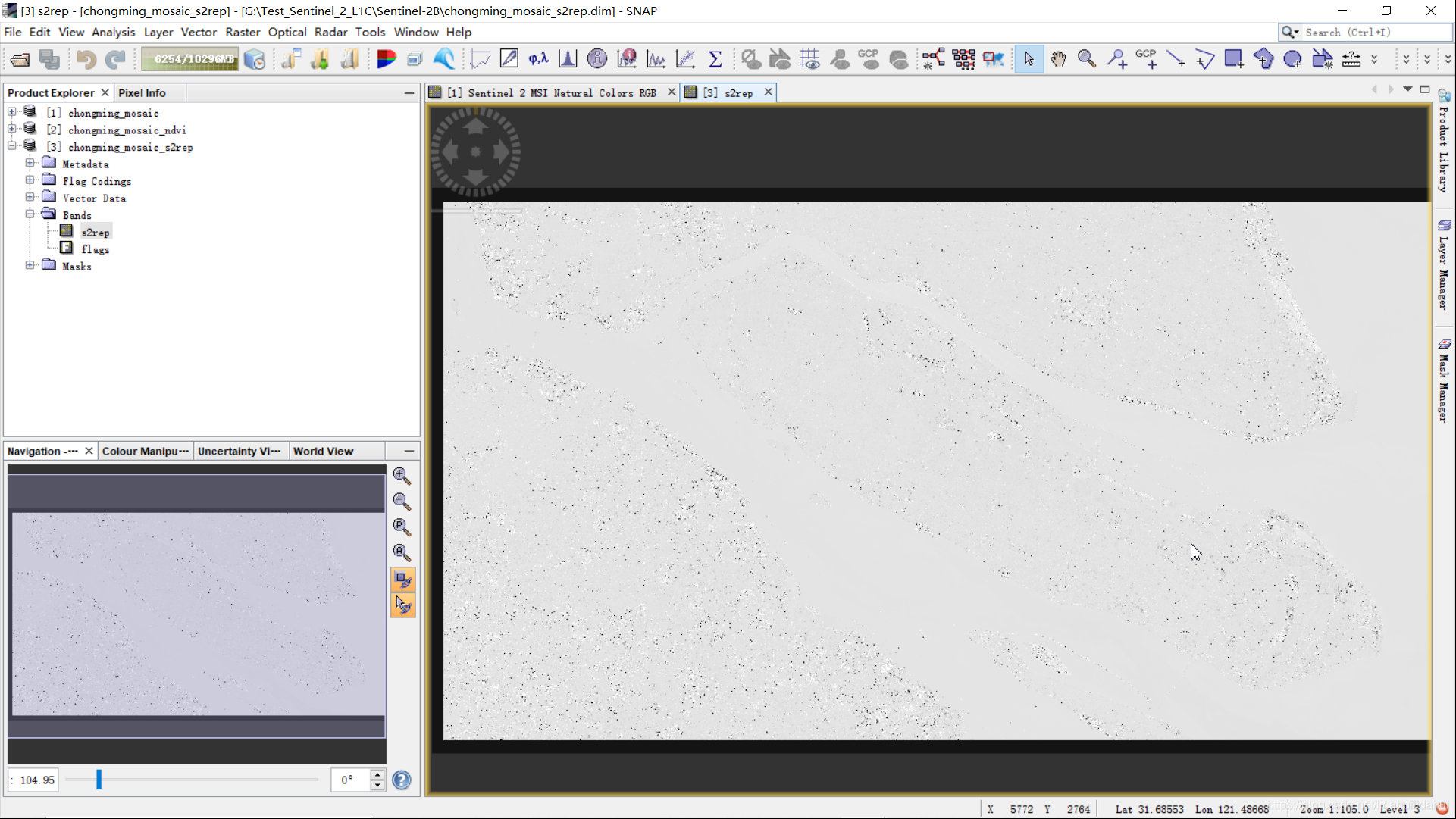
结果:
水体指数MNDWI计算
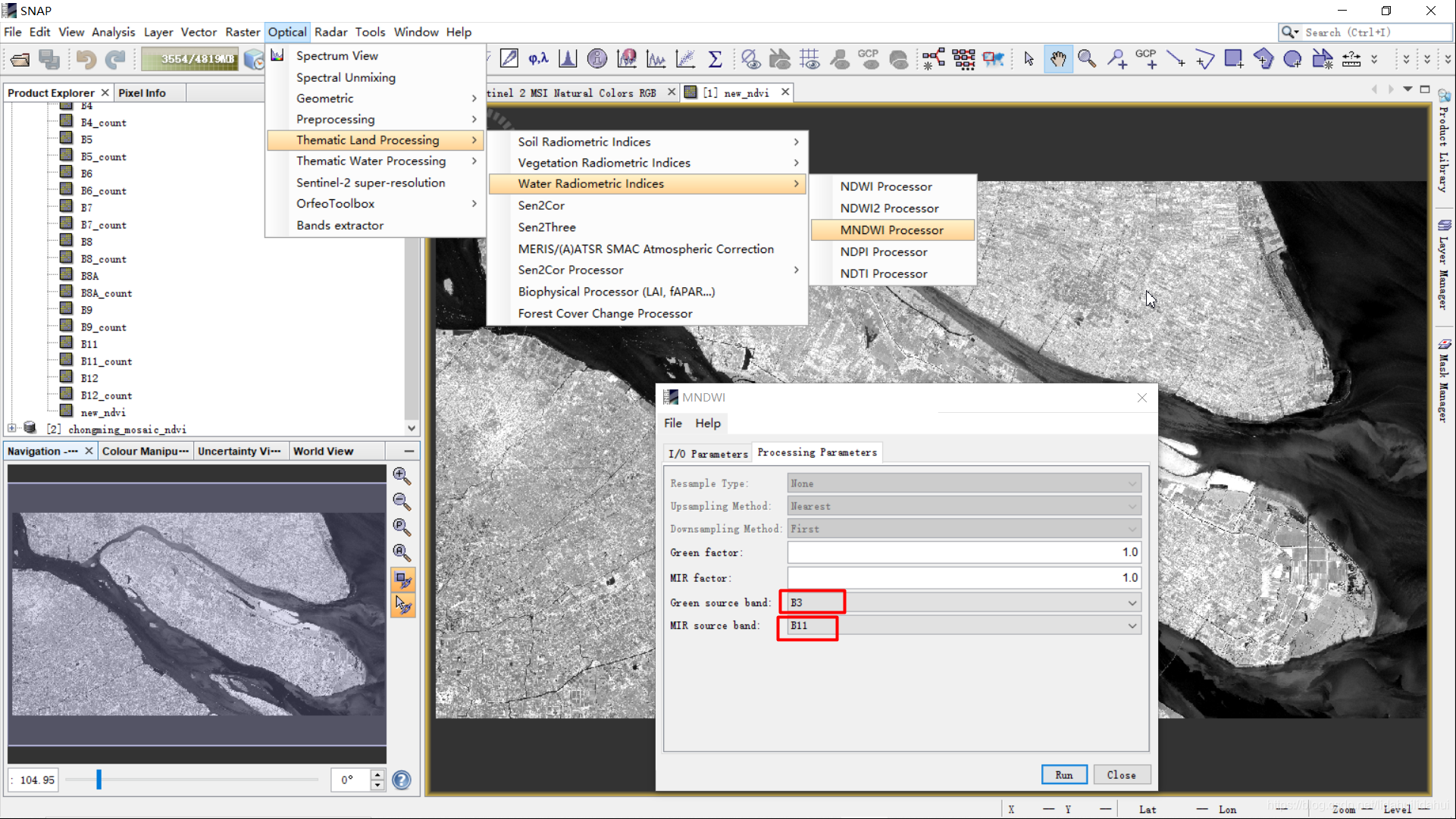
考虑到重采样后具有中红外波段,这里使用它们计算改进的归一化水体指数MNDWI,其计算表达式为:
Green指绿光波段,SWIR(Short Wave Infrared)指短波红外波段。对于Sentinel-2来说是分别对应B3和B11。
MNDWI Processor工具位置,如图:
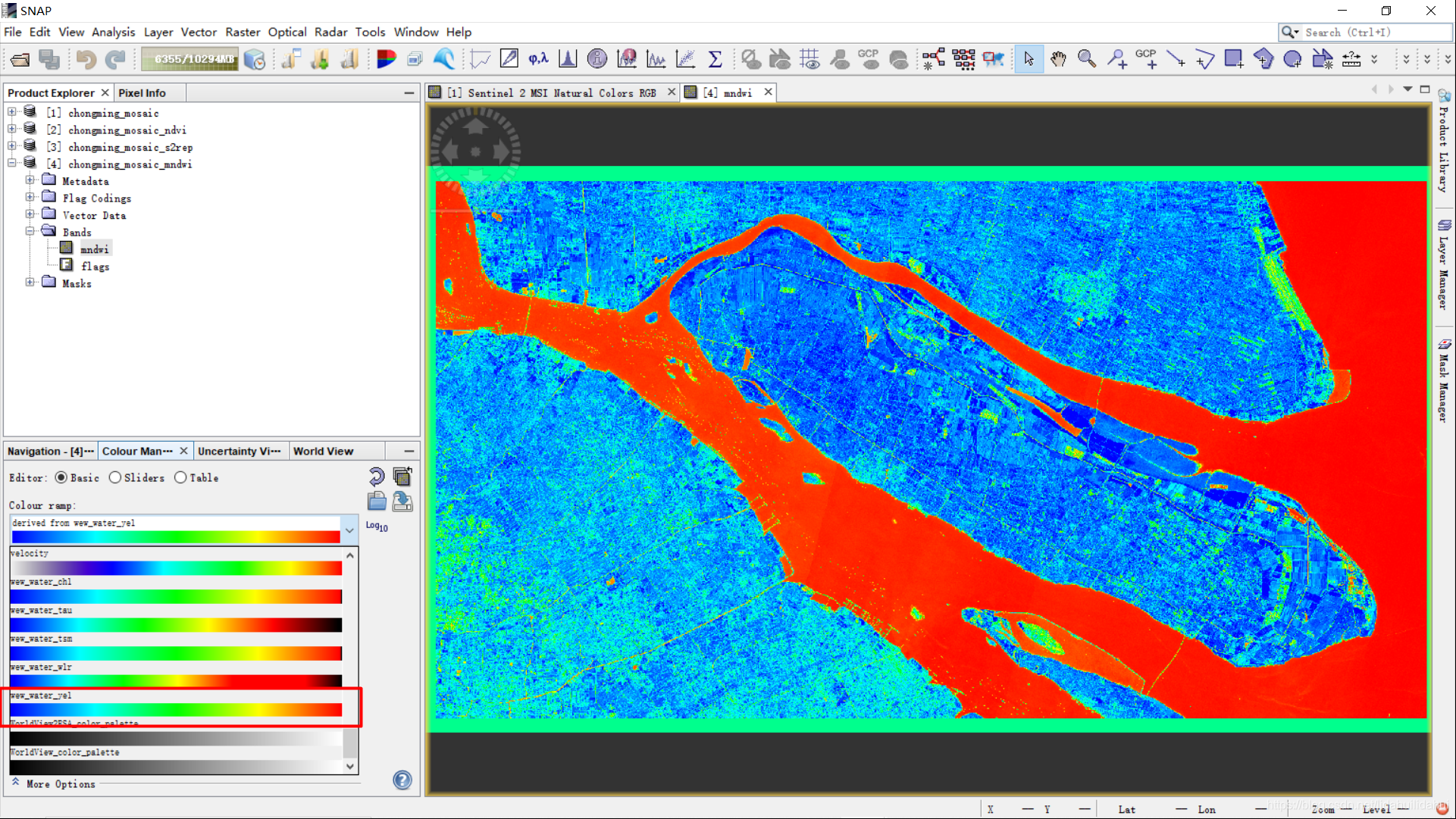
结果:
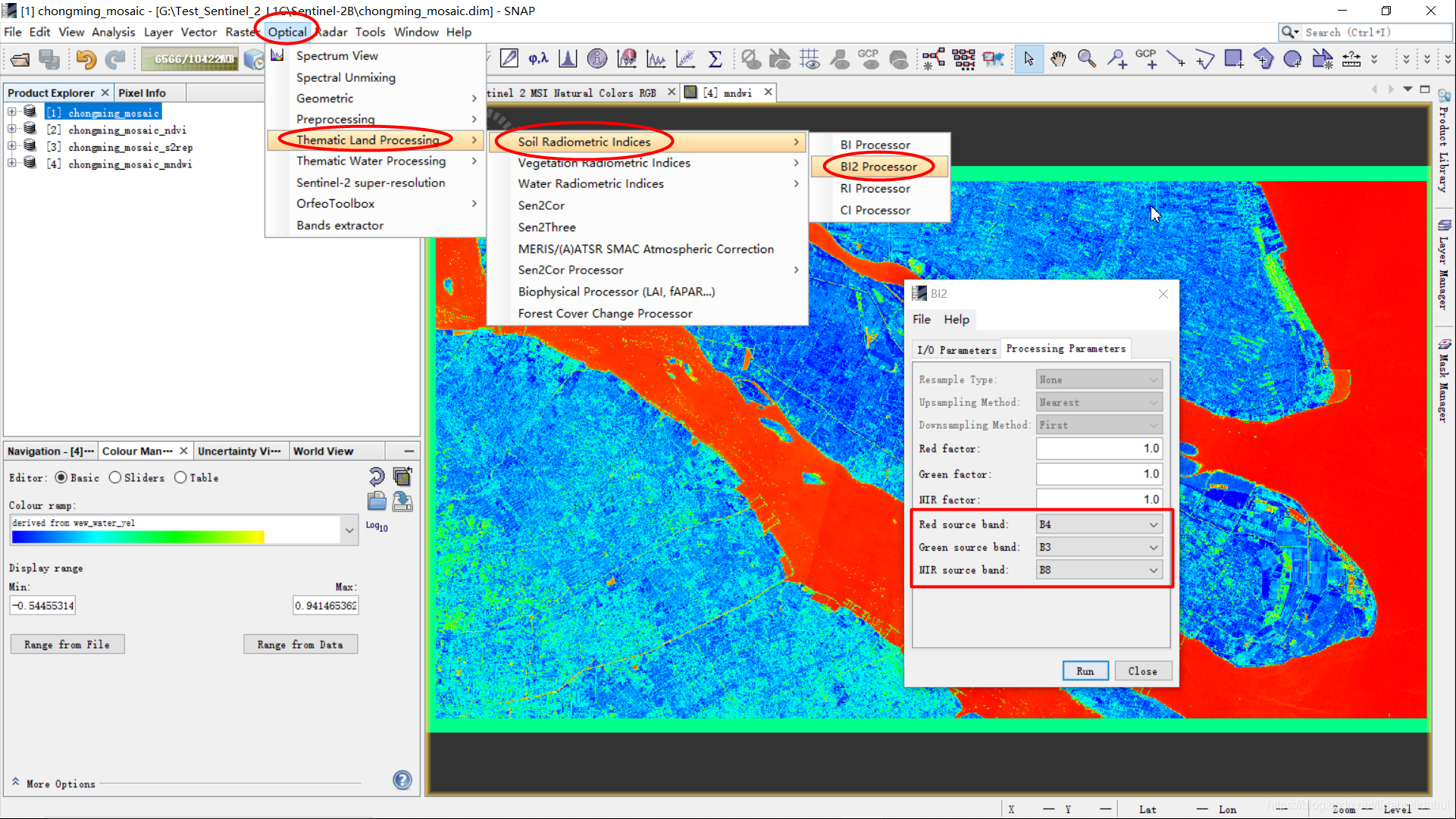
土壤亮度指数BI计算
这里使用BI2指数( The second Brightness Index),该指数对土壤的亮度很敏感,有助于我们区分土壤与其它地物。
BI2 Processor工具位置,以及参数面板:
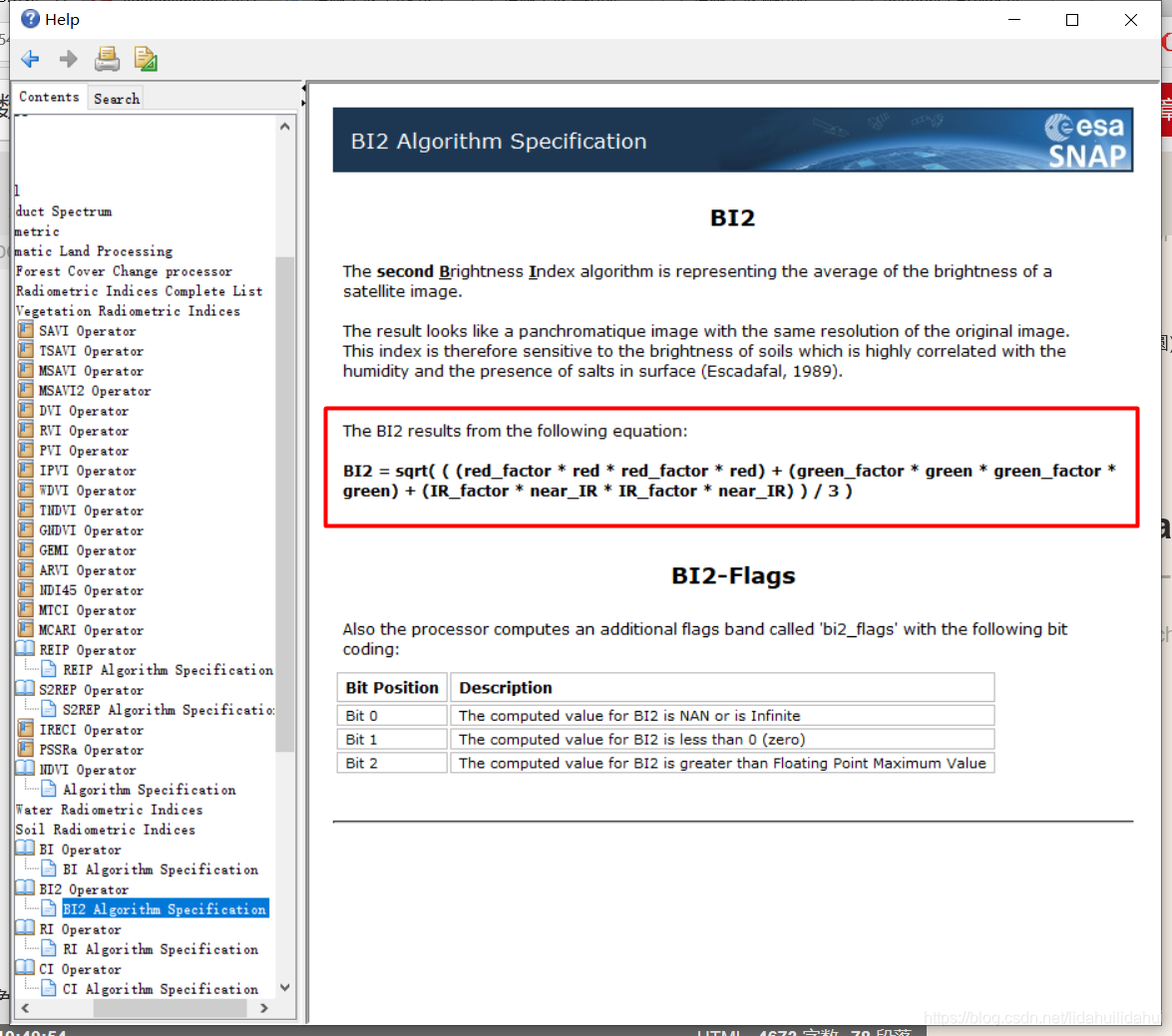
具体算法参见Help的帮助文档
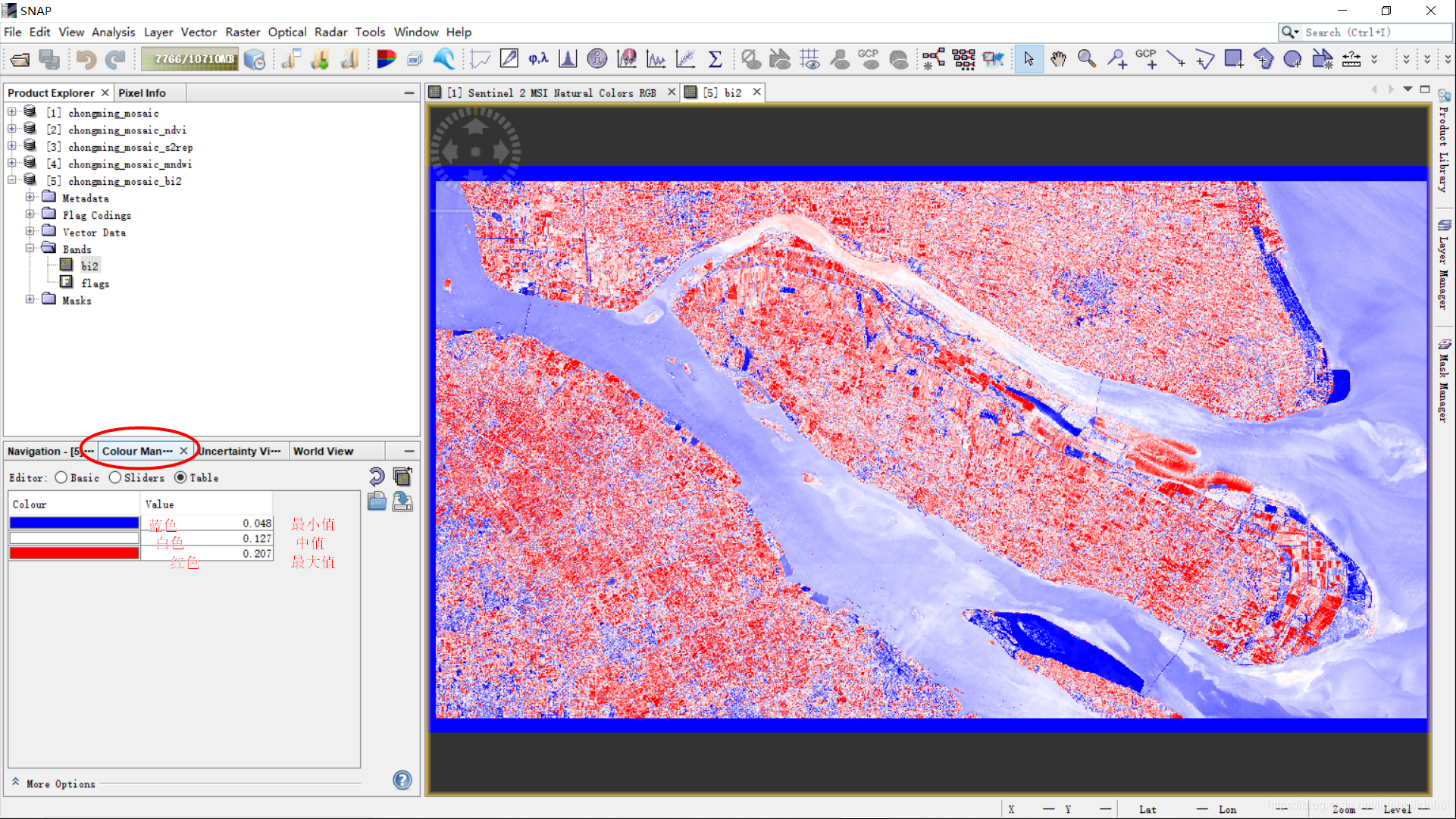
结果(红色越亮表示其是土壤的可能性越大):
好了,指数特征提取到这里结束。你还可以探索别的指数特征。
主成分变换
在多光谱(尤其是高光谱)图像中,邻近波段之间往往具有高度的相关性,存在着大量冗余和重复的信息,需从这些数据中提取那些无冗余的有效信息来识别目标地物。主成分变换(PCA, principal components analysis),可以提取几个不相关的成分,从而有效压缩冗余信息。
博主这里做这个步骤是为提取纹理特征服务的,主要是为避免纹理分析产生的统计分量较多(每一个波段都可以提取纹理特征,如果每一个波段都提取的话,纹理特征将会非常多)且存在一定的信息交叉,参考前人研究经验,在对原始影像进行主成分分析之后选取第一主成分(第一个主成分方差占比均大于0.75)获取来提取纹理特征[1]。一般而言,用分辨率最高大的波段提取纹理特征较为合适,因为分辨率越高,能反映的纹理特征便更多,且更细致。例如,Landsat 8中含有全色波段(分辨为15m),通常可以用这个波段来提取纹理特征。遗憾的是,哨兵2号没有全色波段,不过,其多数波段分辨率优于Landsat 8,整体上说,哨兵2图像性能要好于Landsat 8,哥白尼项目(哨兵1号-6号)出台时,美国人都感到妒忌。
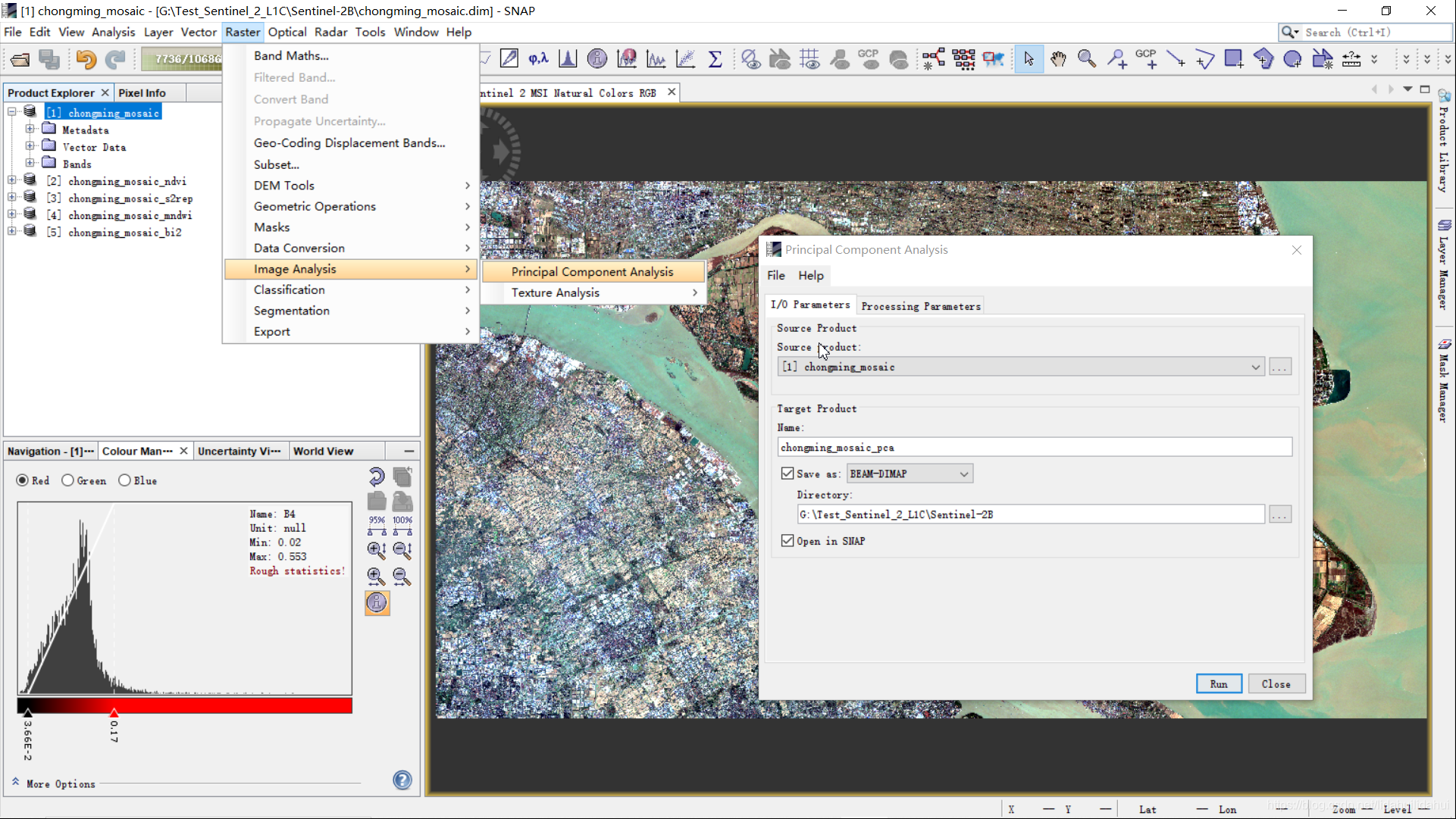
SNAP中的主成分分析操作见下图:
这个步骤比较慢,因为波段数量比较多,分辨率较高,数据量是比较大的。需要保持耐心。(不过,这个模块最初是针对针对哨兵1数据处理而开发的,似乎对哨兵2号处理很慢,博主等了一天多还没有结果,后来放弃用这个做处理,改用开源的Orfeo Toolboxs做的主成分分析,此外,GRASS GIS,ENVI等也可以做。博主最近在研究OrfeoToolboxs(下称OTB),日后也可能对OTB做下介绍。其实,SNAP也集成了一部分的Orfeo Toolboxs的图像分割功能(Optical—>Orfeo Toolboxs)。OTB支持面向对象分类,当然,它也支持面向过程分类,关键其是开源,是开放的C++源码,采用流数据等处理技术,运行效率很高。SNAP也是支持添加第三方的插件,事实上,可以以bat脚本(shell脚本)的形式将第三方的插件添加到SNAP中,类似于其Sen2Cor插件等)。
虽然最后是使用OTB做的主成分变换,但是这里还是将其参数简要介绍一下(SNAP论坛中说的,可以将数据类型类型转为uInt8,以便加快主成分变换,但我试了没有效果,操作:Raster—>Data Conversion—>Convert Datatype)。
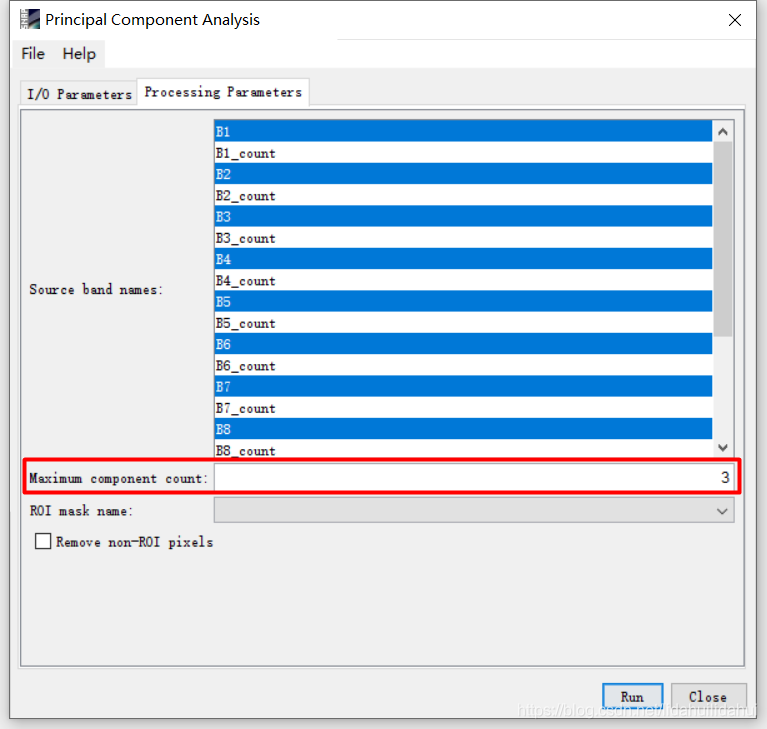
参数面板中只选择光谱光谱波段(B1,B2,B3,B4,B5,B6,B7,B8,B8A,B9,B11,B12,按住Ctrl键可以选择,鼠标左键单击,可以实现选择多个波段);Maximum component count指的是主成分的数量,这里设置的是3。
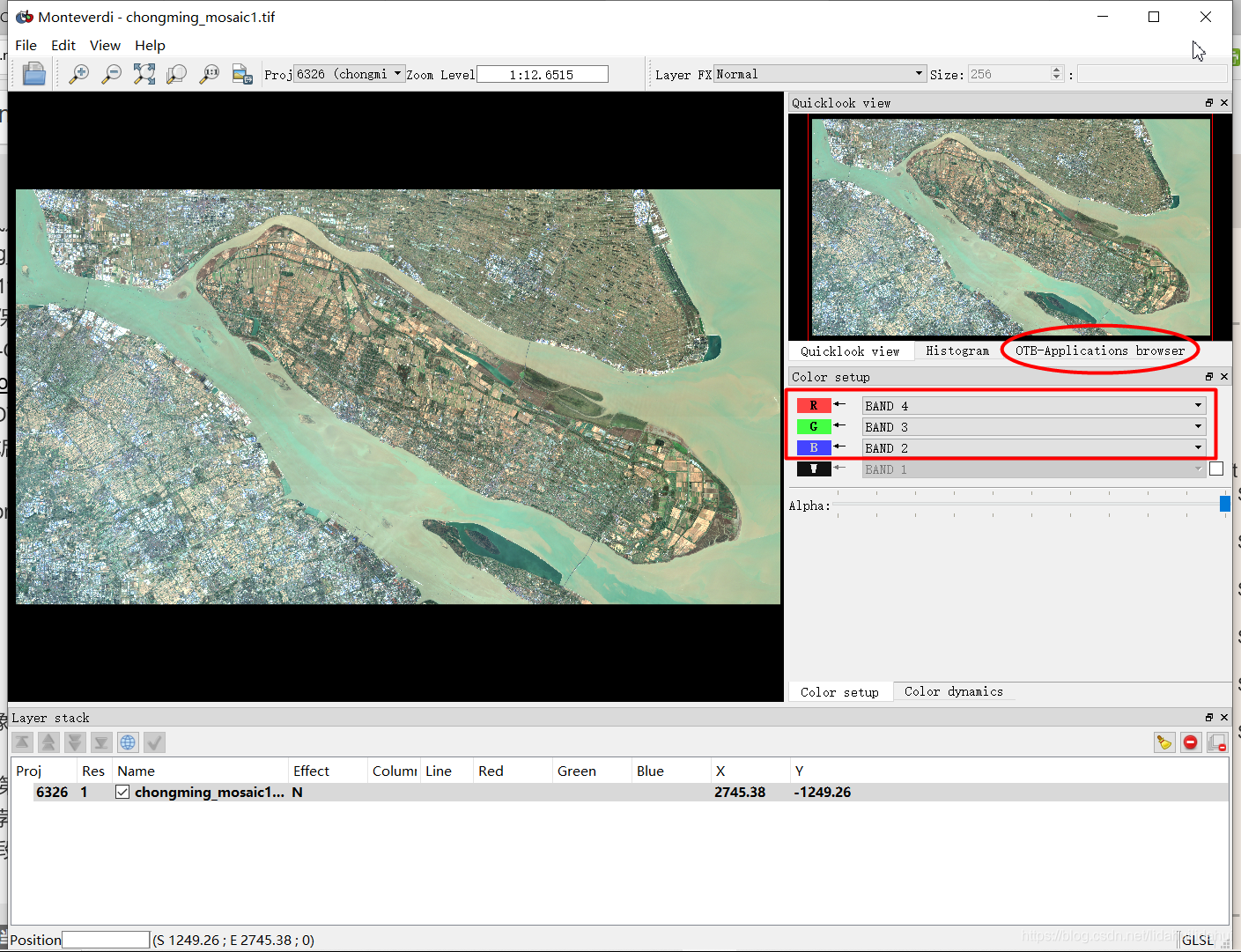
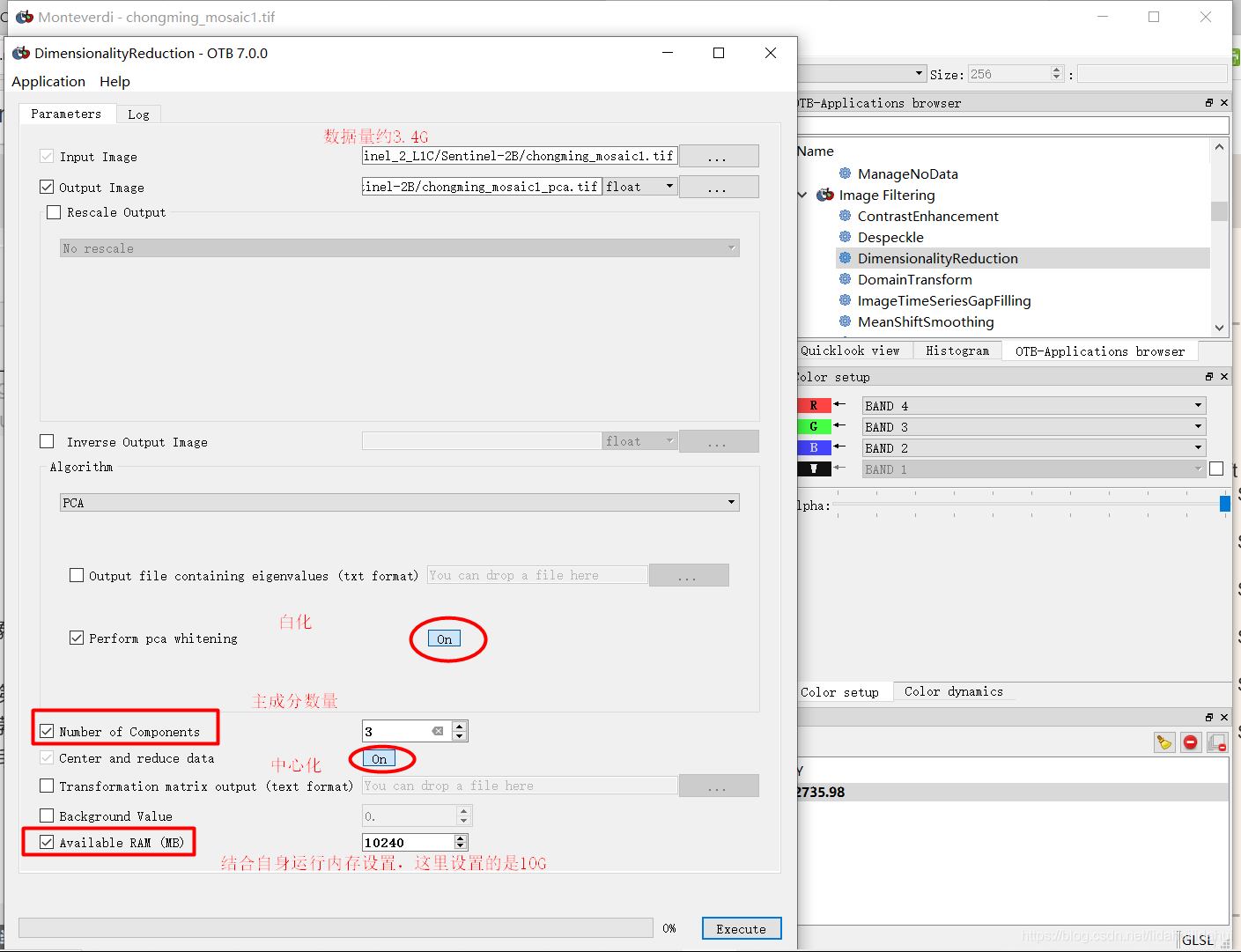
点Run后就是漫长的等待过程。后来我是用Orfeo Toolbox 7.0.0(OTB)做的(我简单说下过程,现将chongming_mosaic数据集中B1,B2,B3,B4,B5,B6,B7,B8,B8A,B9,B11,B12提取出来(Raster—>Data Conversion—>Band Select),保存为Geotiff的形式,注意Save As那个下拉选项需要使用GDAL-GTiff-WRITER,别的GeoTiff格式驱动可能出错;下载Orfeo Toolbox 7.0.0,解压;找到OTB-7.0.0-Win64文件夹下的monteverdi.bat脚本(这个是OTB的GUI界面脚本,这个我也不知道为啥起这个名字),以命令行的方式启动monteverdi.bat(注意,不要关闭命令行窗口);File—>Open,打开转换好的geotiff图像;在View下选项中勾选OTB-Applications browser);在OTB-Applications browser—>Image Filtering—>DemensionalityReduction,启动降维操作面板,按后最后两个图设置参数,点击Execute,运行速度超快。最后可以在SNAP打开主成分变换结果。
 降维操作
降维操作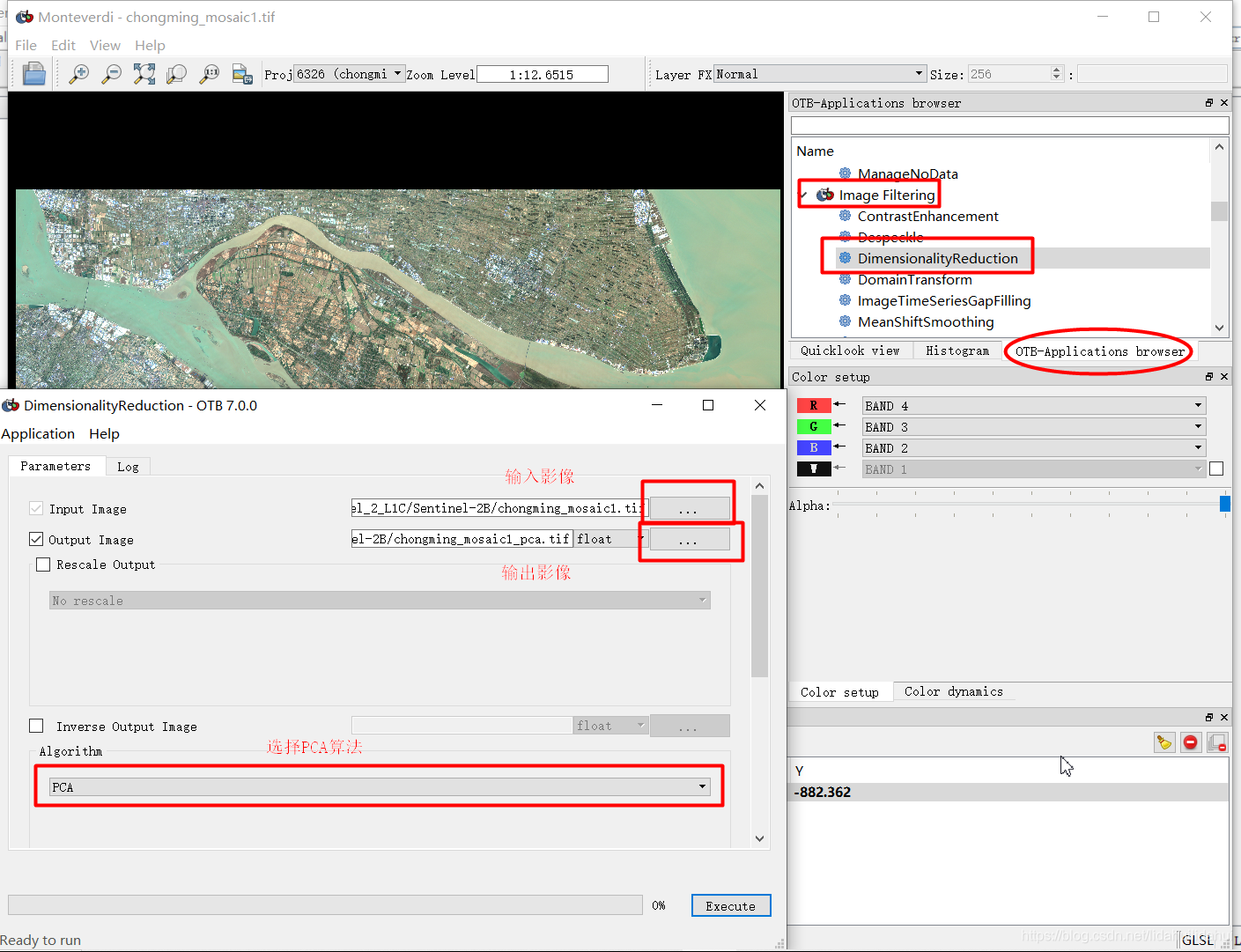
纹理特征提取
纹理反映了图像灰度模式的空间分布,包含了图像的表面信息及其与周围环境的关系,更好地兼顾了图像的宏观结构与微观结构[2].
这里利用Orfeo Toolbox 主成分变换的第一个波段来提取纹理特征。如果你没有做主成分变换,我推荐你使用可以使用NDVI来提取纹理特征(NDVI的计算源自红光波段和近红外波段,这两个波段都是10m分辨率的,原始Sentinel-2最高分辨率的两个波段,并且含有较丰富的光谱信息)。
打开PCA变换band_1的结果如图:
提取纹理特征,操作见下图
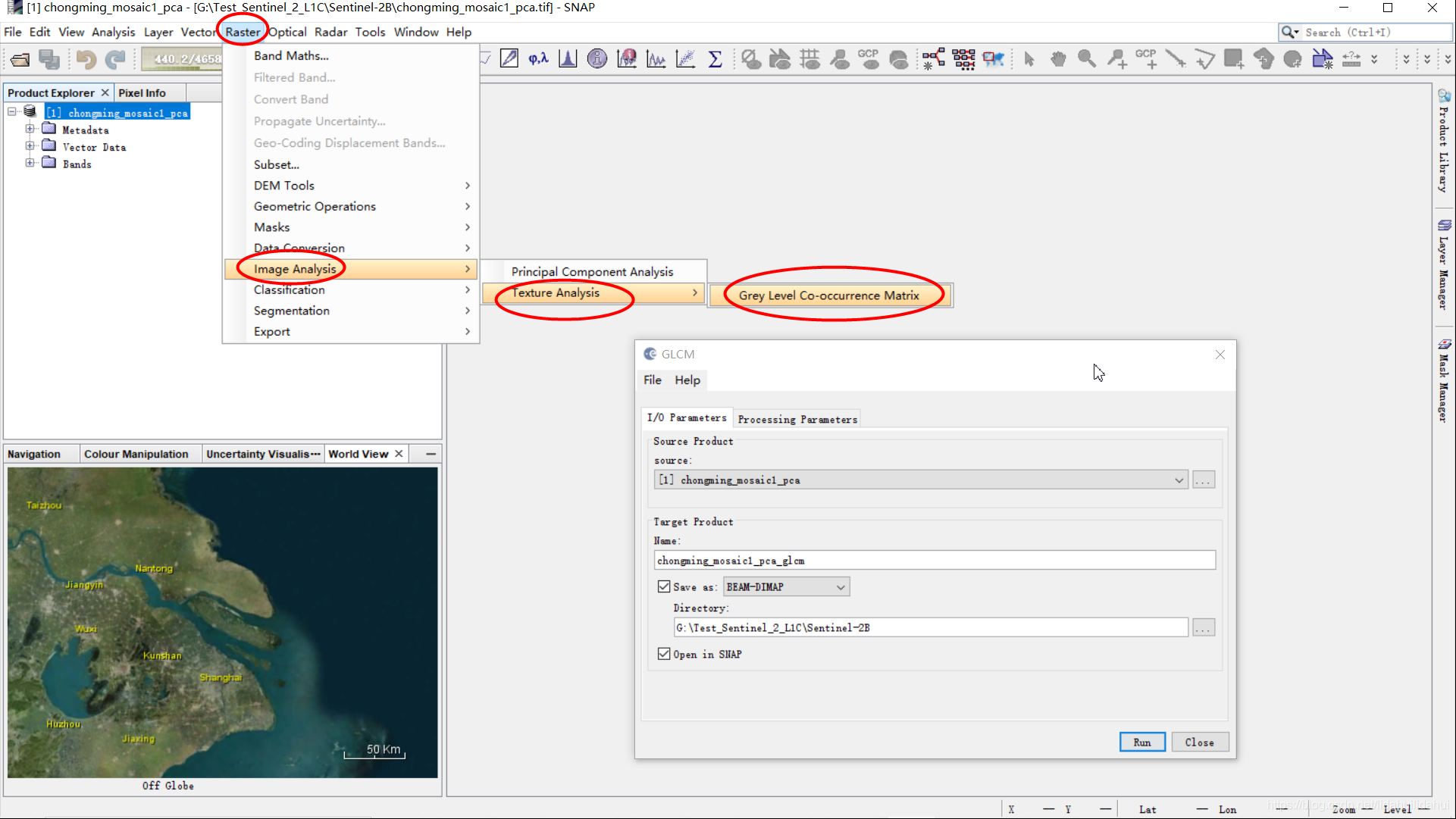 参数面板设置:
参数面板设置:
该操作是基于灰度共生纹理矩阵提取的,其原理介绍结见文献2。
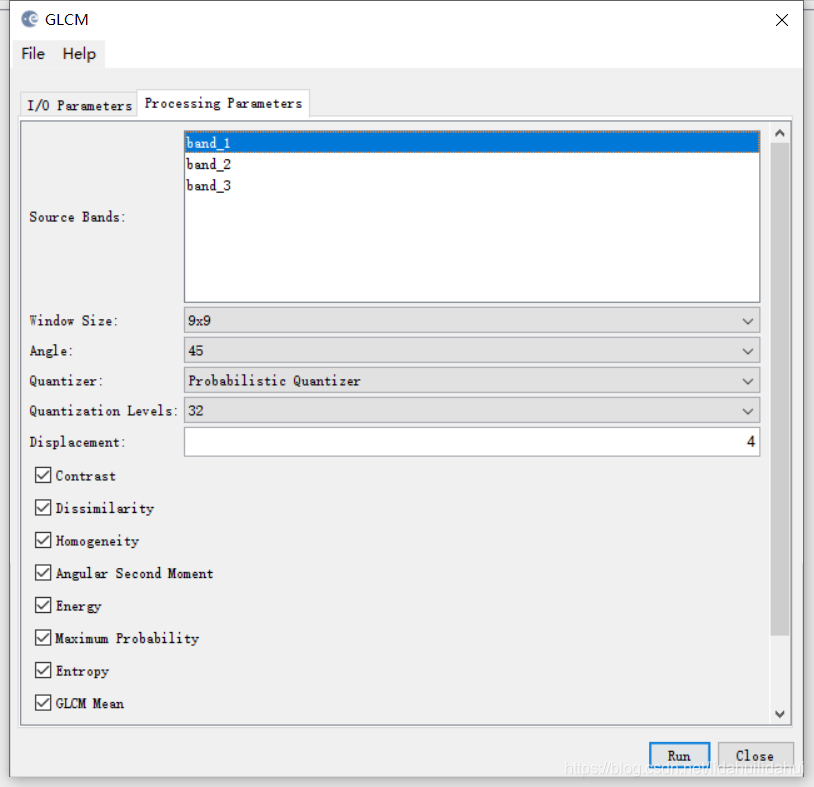
Window Size:统计像元的窗口大小;Angle:角度(0°,45°, 90°,135°,All)沿那个方向(角度)提取纹理;因为崇明岛大致是沿东南-西北走向的,这里选择45;Quantizer:量化器(划分器),选择默认值Probabilistic Quantizer(等概率量化,可以理解为等比例划分),Equal Distance Quantizer(等距离量化,可以理解为等区间划分);Quantizations Level:指灰度量化级(为2的倍数),选择默认值32。displace,这个参数我也没有搞明白是干啥的,保持默认就行。
source band:只选择band_1。
如图:
点击Run:
得到的纹理特征结果(参考Help以及文献2):
Contrast Group Features(对比度组特征):
Contrast(对比度),Dissimilarity(非相似性), Homogeneity(均匀性);
Orderliness Group Features(有序性组特征):
Angular Second Moment(二阶矩), Maximum Probability(最大概率),Entropy(熵);
Statistics Group Features(统计组特征):
GLCM Mean(均值),GLCM Variance(方差),GLCM Correlation(相关性);
主成分第一分量Contrast(对比度)特征展示: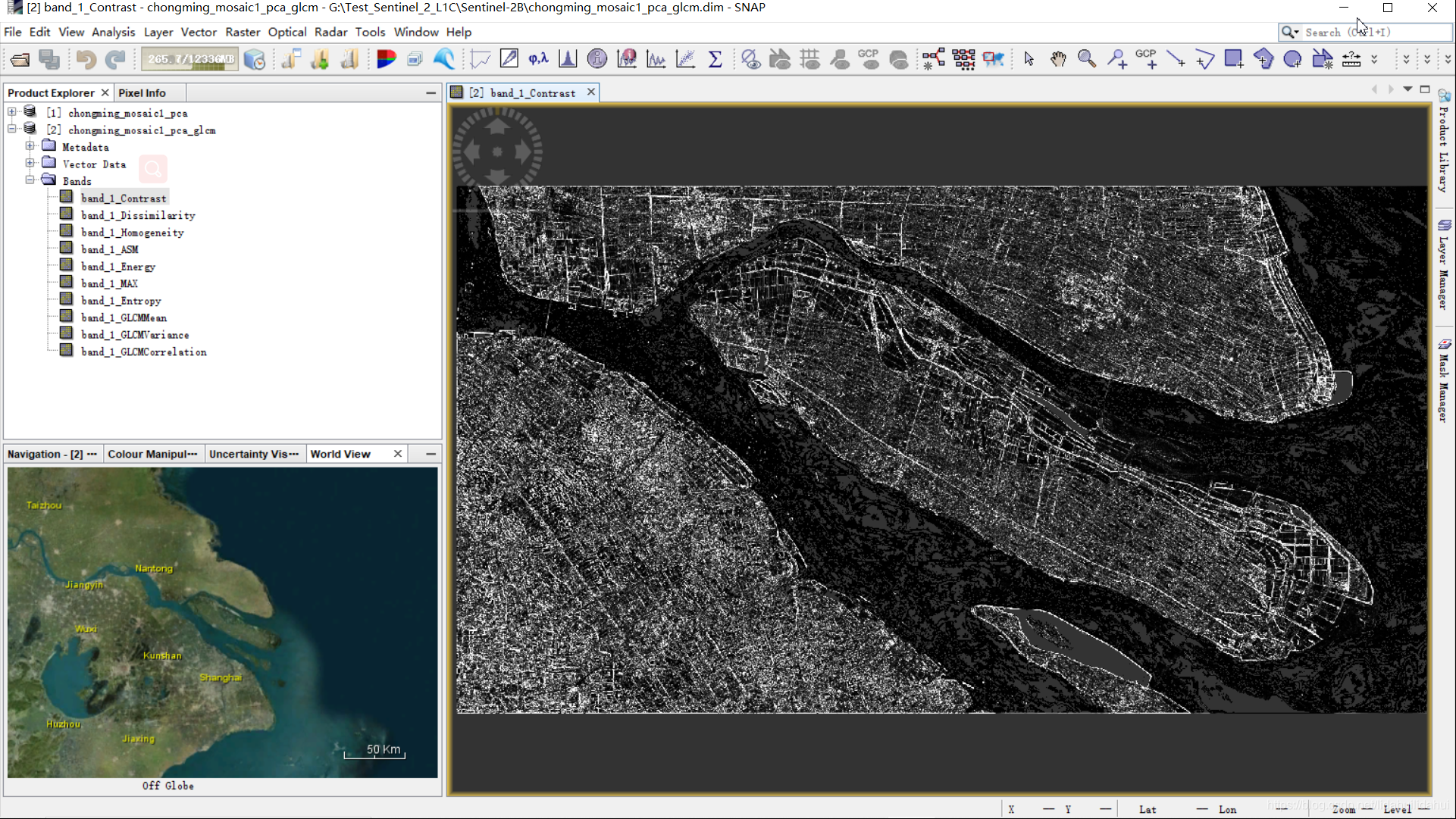
其它的特征可以自行探索。
波段叠加
特征工程最后一步,我们可以将三种类型的特征叠加起来:光谱特征、指数特征、纹理特征叠加起来。这个操作可以通过BandMerge操作可以实现。这个操作在菜单栏中是无法找到的,需要从流程图中才能找到。
打开上述原始的以及处理得到的数据(chongming_mosaic.dim,chongming_mosaic_ndvi.dim,chongming_mosaic_s2rep.dim,chongming_mosaic_mndwi.dim,chongming_mosaic_bi2.dim,chongming_mosaic1_pca_glcm.dim),打开流程图窗口:
法1:点击工具栏快捷图标

法2:Tool菜单栏的GraphBuilder打开:
BandMerge工具
按上图的红色框指示,打开流程图窗口:
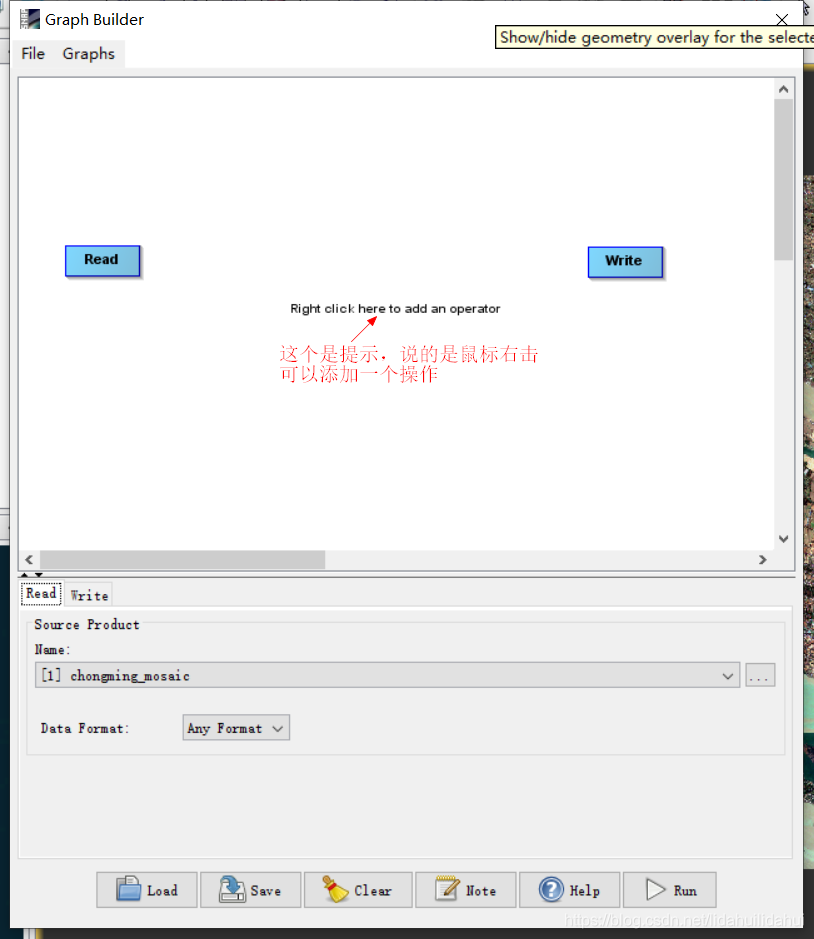
在流程图窗口中,鼠标右击,添加操作BandMerge(鼠标左键长按在某个流程框上,可以拖动流程框在窗口的位置,从而调整流程布局):
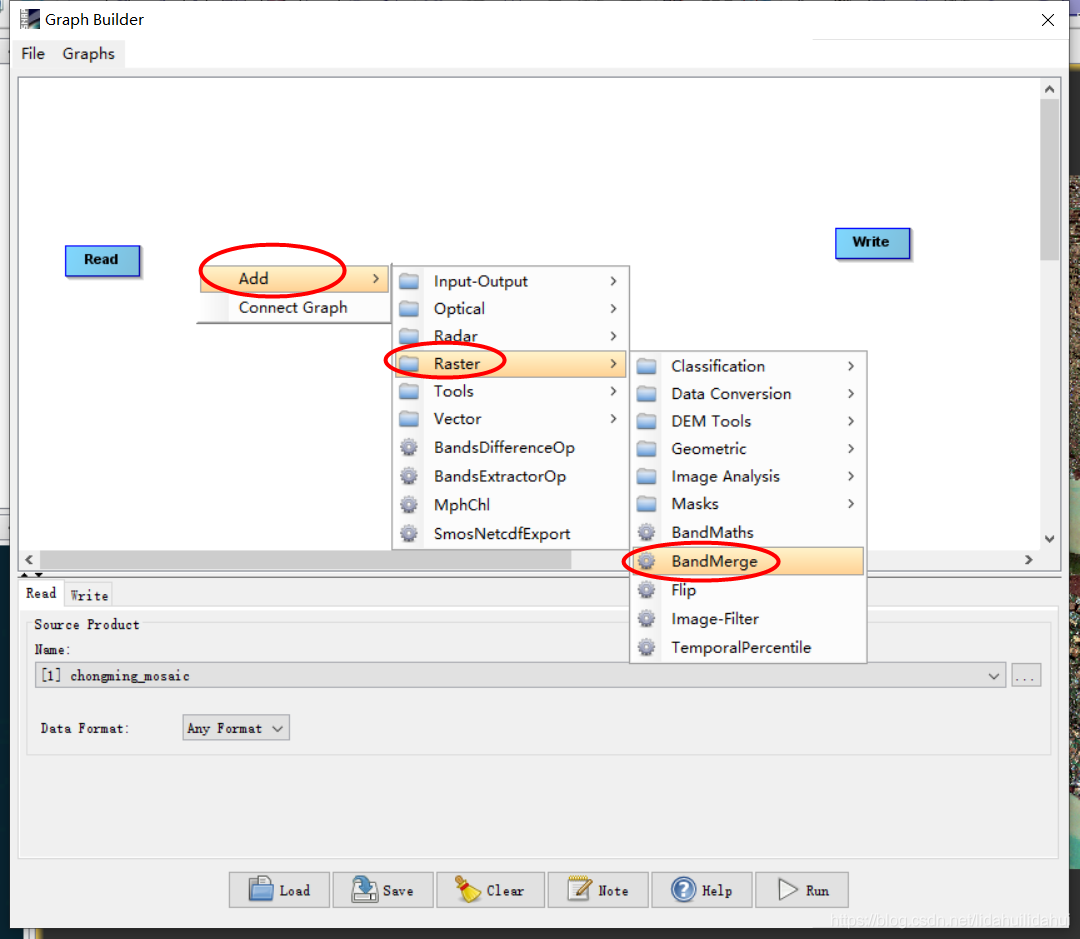
添加好BandMerge操作的效果:
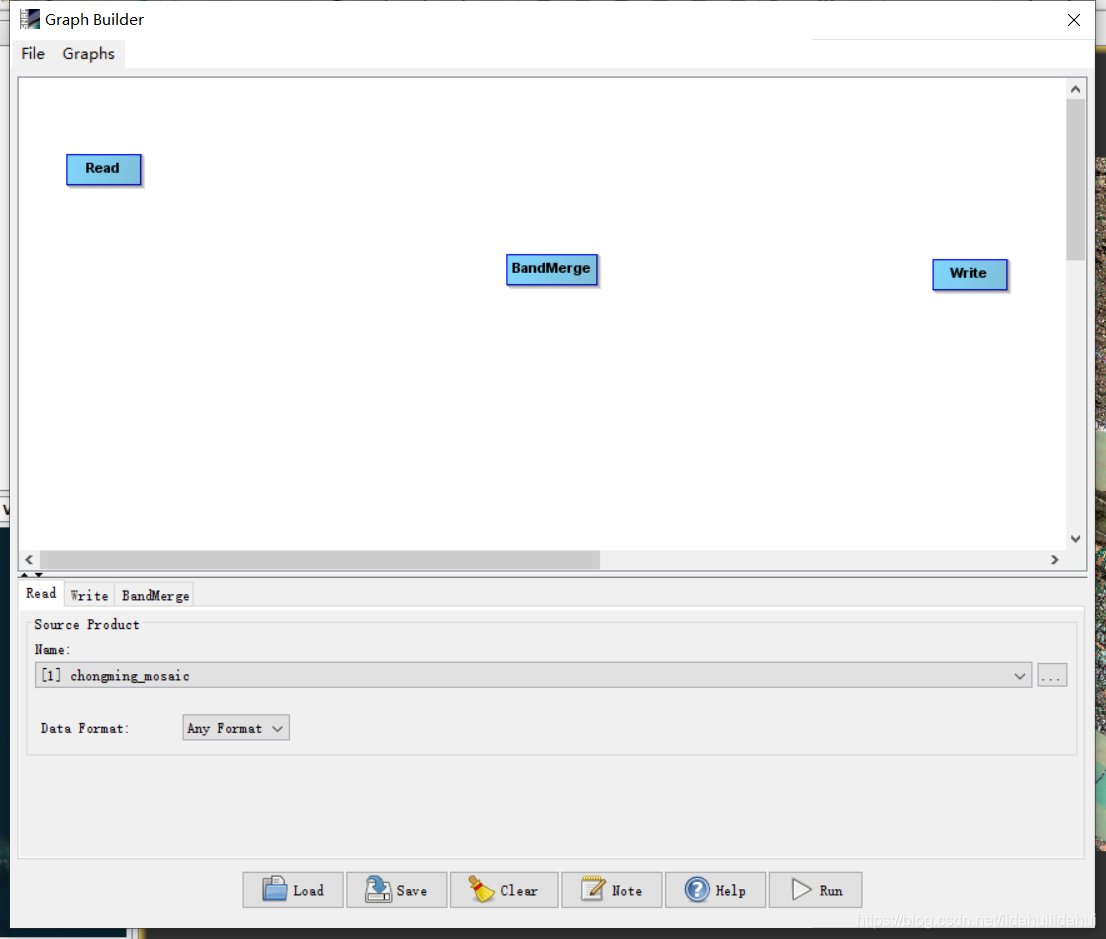
由于我们有6个输入的数据集,一个叠加的输出数据集,所以,我们需要6个Read操作(输入),一个Write操作(输出),现在还差5个Read操作。(纹理矩阵特征是第三方的软件产生tif影像可能会丢失地理坐标系信息,如果丢失了,直接将其读入进行BandMerge时会出错的),所以我们要分两个流程图进行合并第一个流程图合并所有的指数特征ndvi,s2rep,mndwi,bi2(即产品数据集2,3, 4,5),第一个流程图还需要3个Read操作:
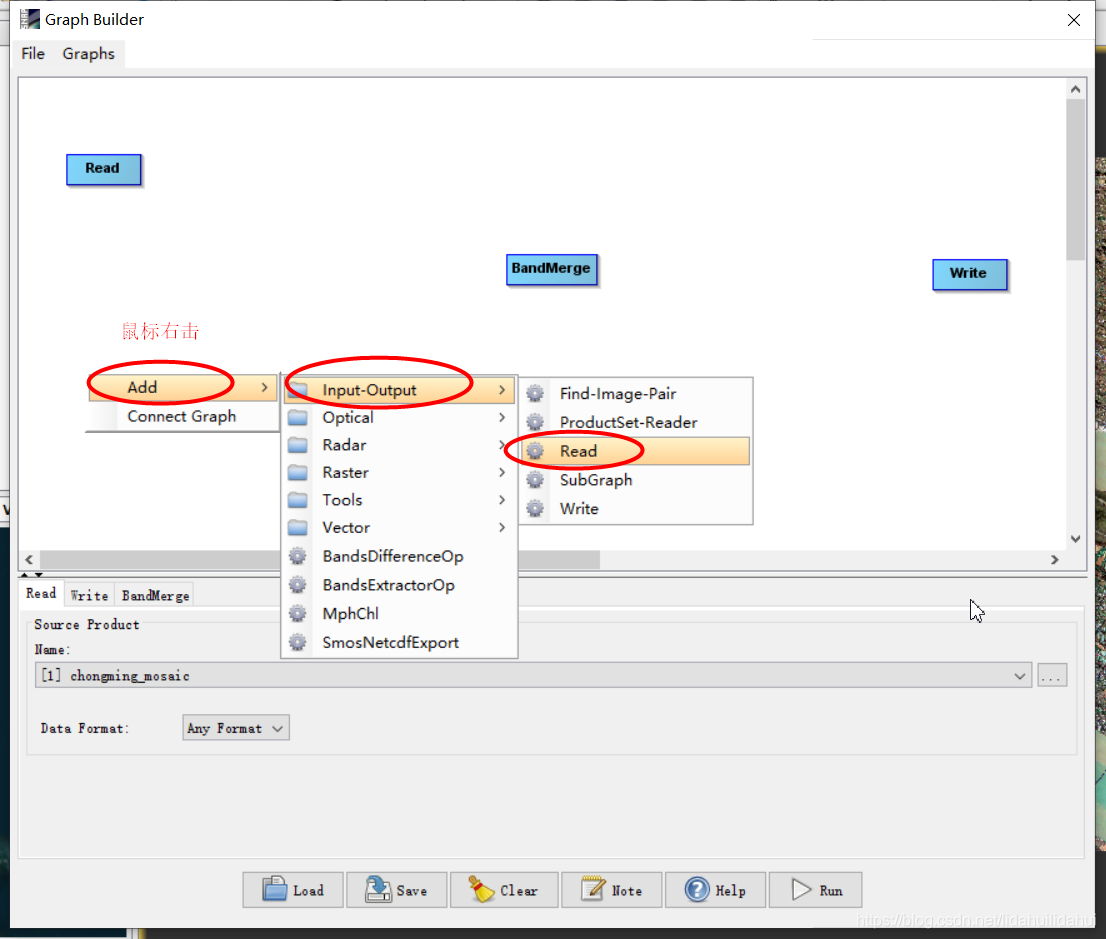
链接两个流程框:
鼠标左键移动到前一个流程框右边界(大致中间位置),长按住鼠标左键,向要连接的下一个流程框拖动出一个连接线,直至连接线箭头部分与下一个流程框左边界(上下边界中部位置也是可以的,只是有时识别不是很好)接触(或超过一部分)。如果是新手建议多操作几遍,以便熟悉流程图操作因为流程图是SNAP中一个非常重要的功能,可以借助流程图实现批量处理,参见下图提示:
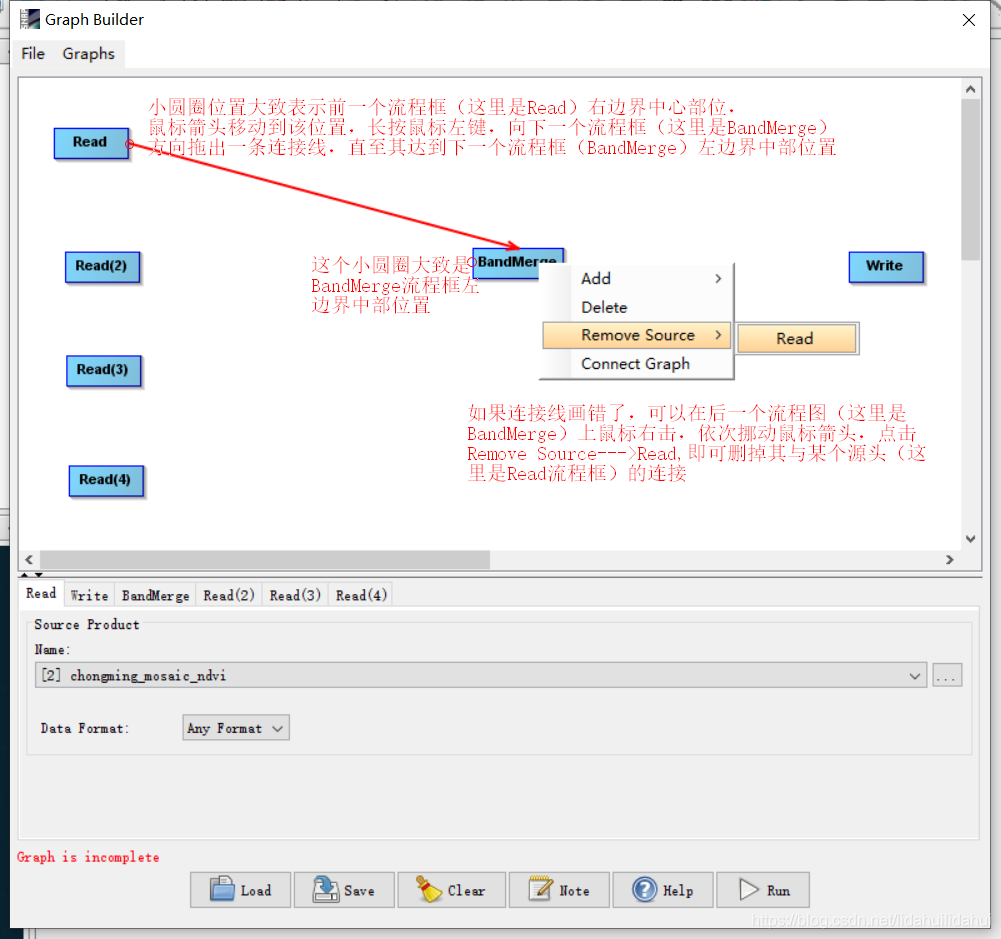
(此外,还可以看到流程图窗口的红色预警提示:Graph is incomplete,提醒我们流程图没有完成)
参见下图的连接图,自主完成流程图的创建:
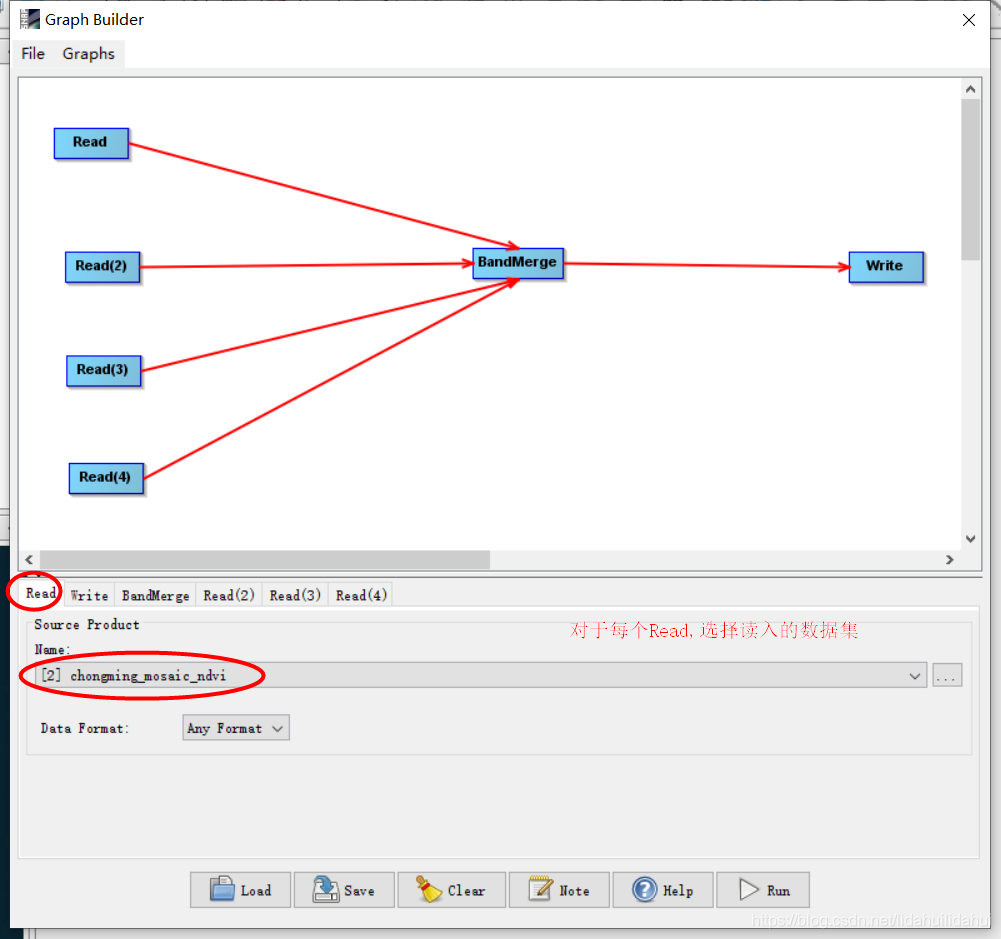
(完成后可以看到红色的提示消失了)
Read选择的是数据集2(ndvi),Read2选择的是数据集3(s2rep),Read3选择的是数据集4(mndwi),Read4选择的是数据集5(bi2)。
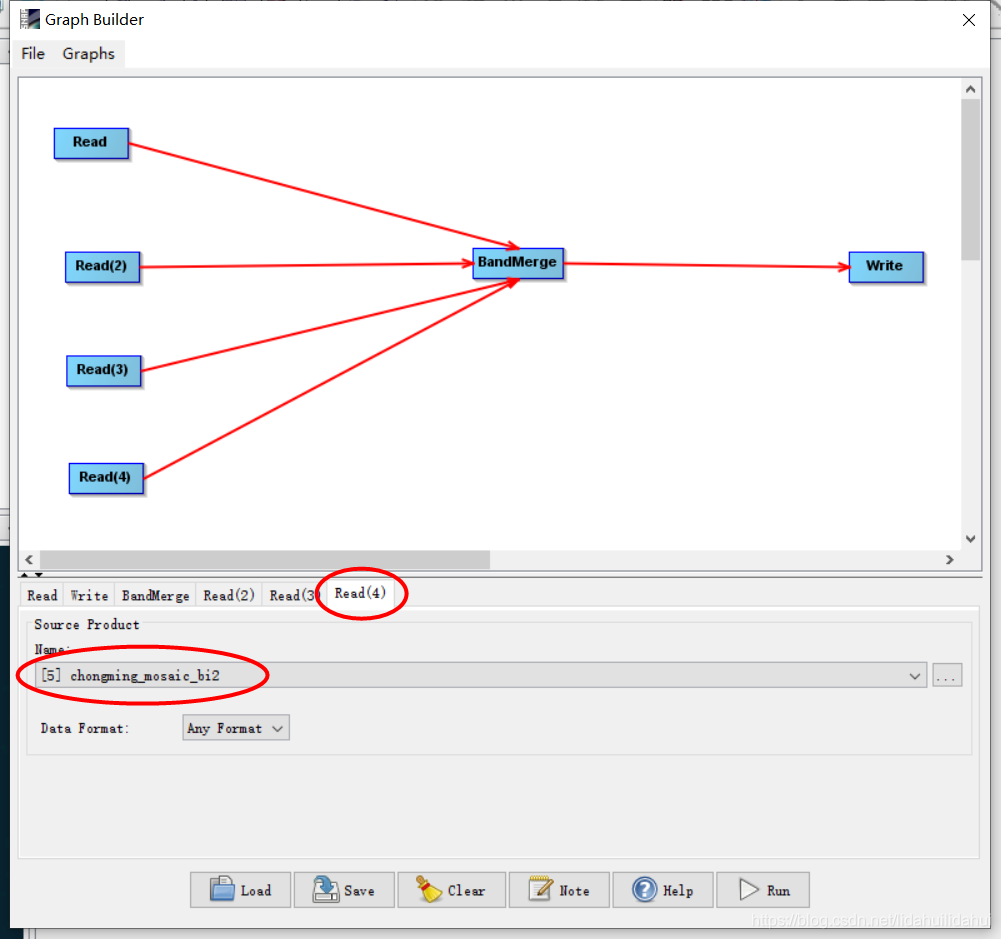
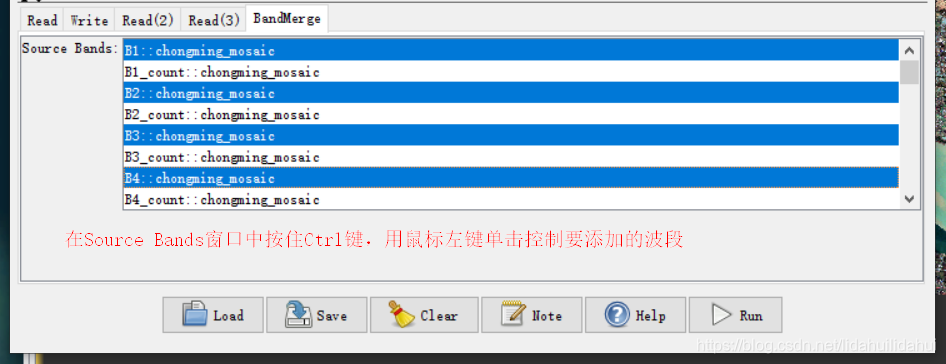
再回到BandMerge操作上,选择需要提取的波段(这里不选择flags波段):

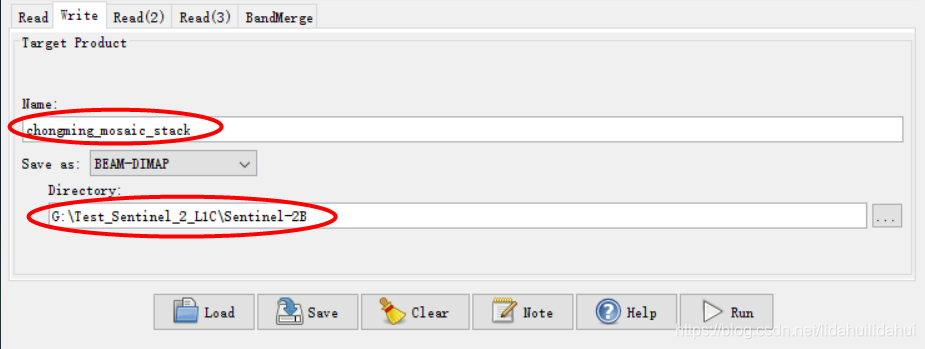
再回到Write操作,修改输出的数据集文件名以及输出的数据目录:
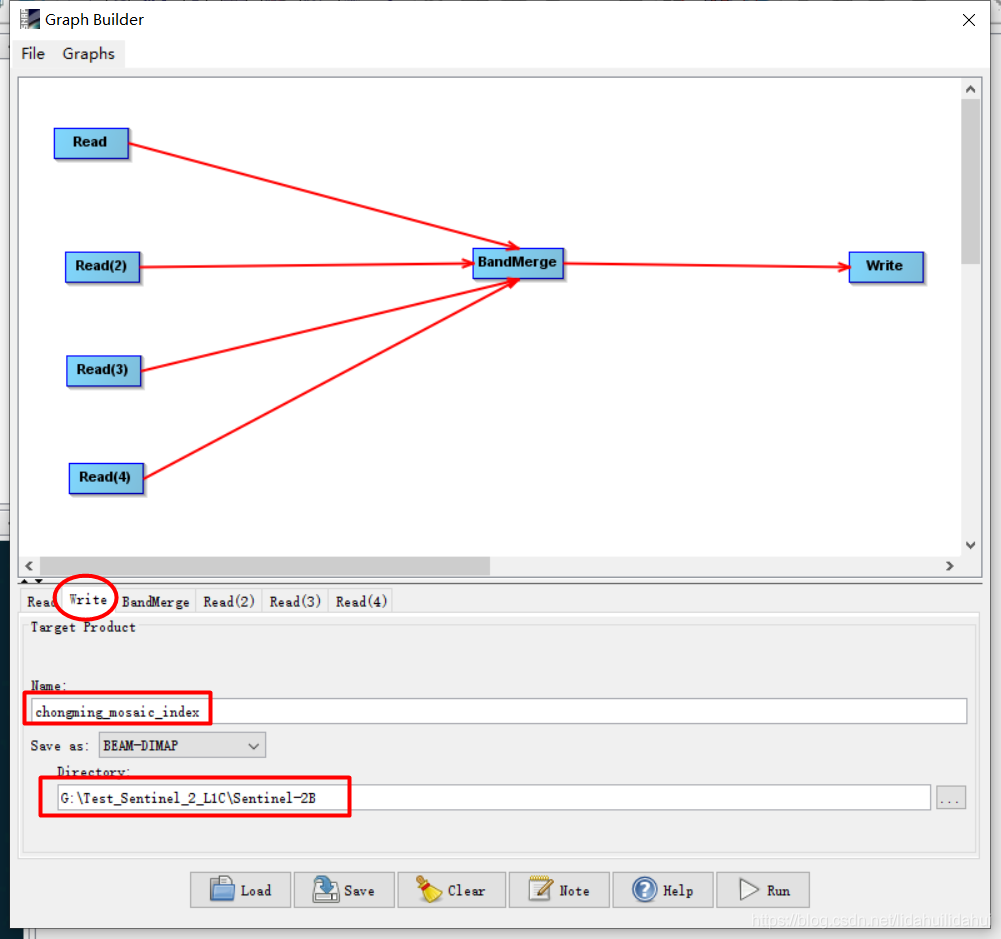
完成后点击Run(下方会出现进度条),等待二十多分钟左右,提示完成: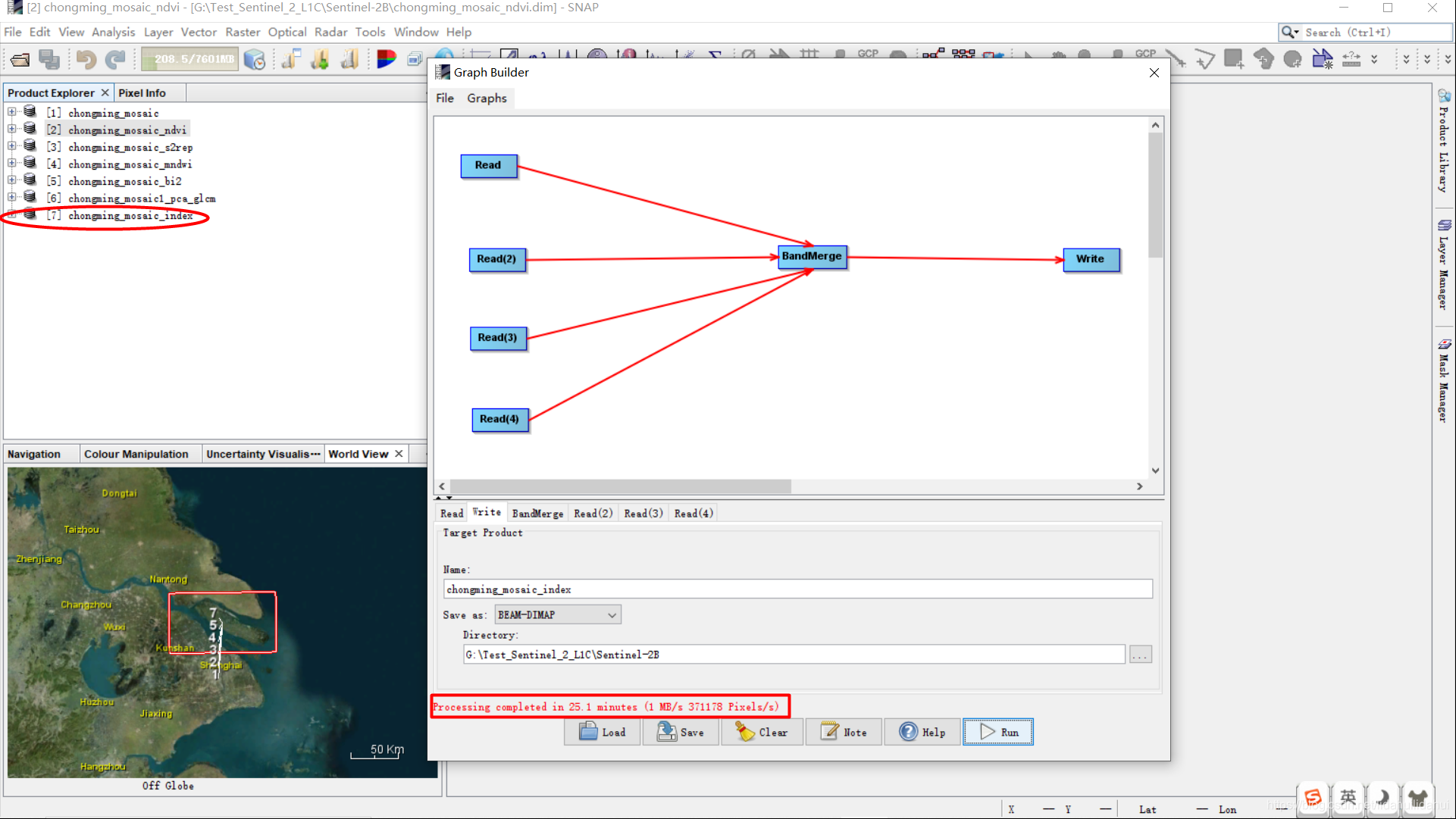
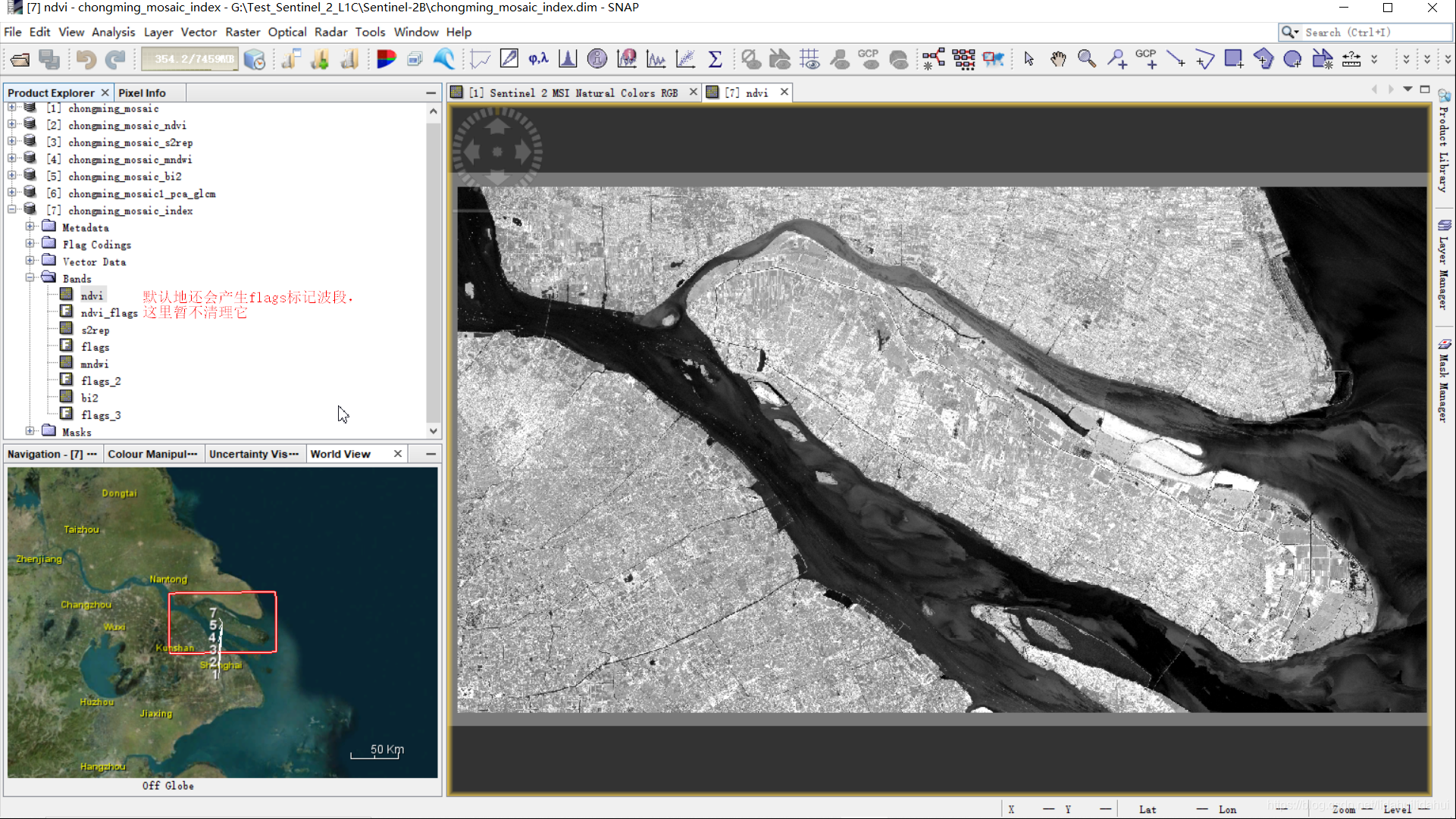
可以打开数据集7某个波段看下效果(如ndvi):
再用一次流程图,将数据集1(原始的光谱特征集)、6(纹理特征集),7(指数特征集)叠加起来, 借助BandMerge操作完成。
连接好的流程图如下图所示:
(其中Read对应数据集1(光谱特征集),Read2对应数据集6(纹理特征集),Read3对应数据集7(指数特征集))
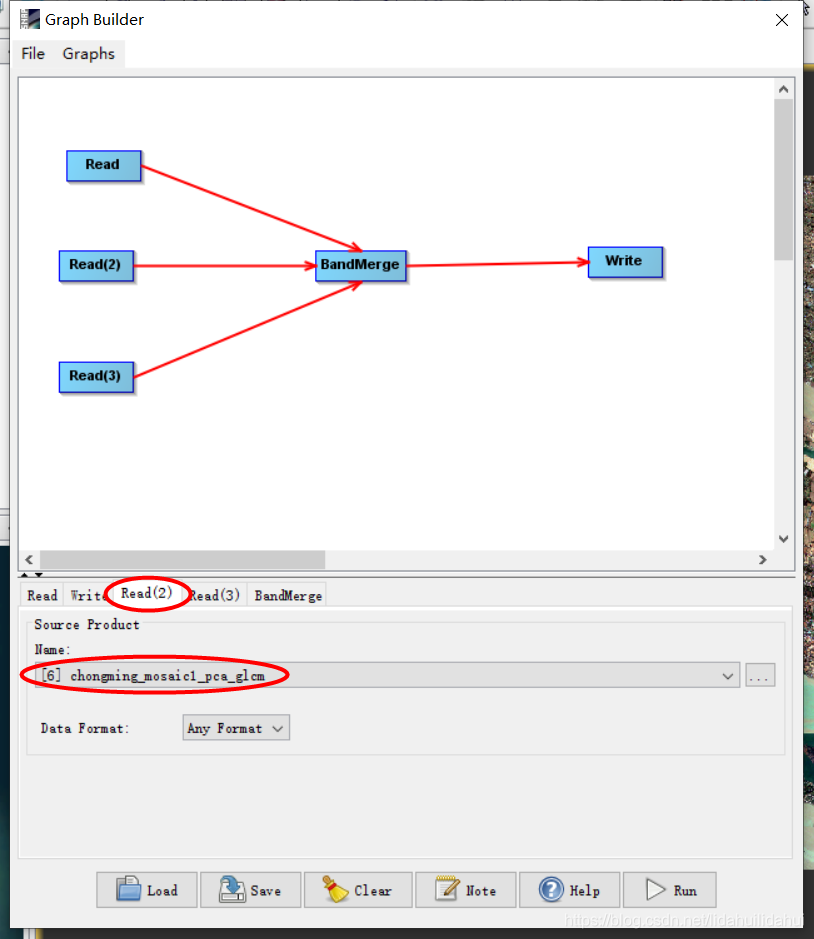
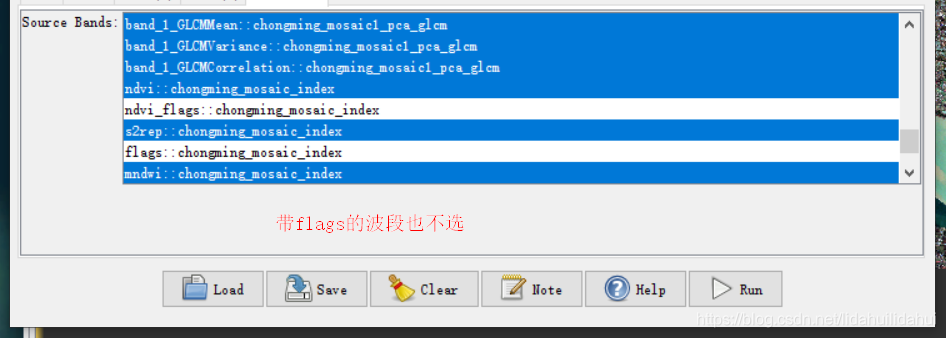
设置好3个Read操作后,再回到BandSelect操作,选择适当的波段进行叠加(其中count波段和flags波段都不选):
回到Write操作,修改输出文件名和文件路径:
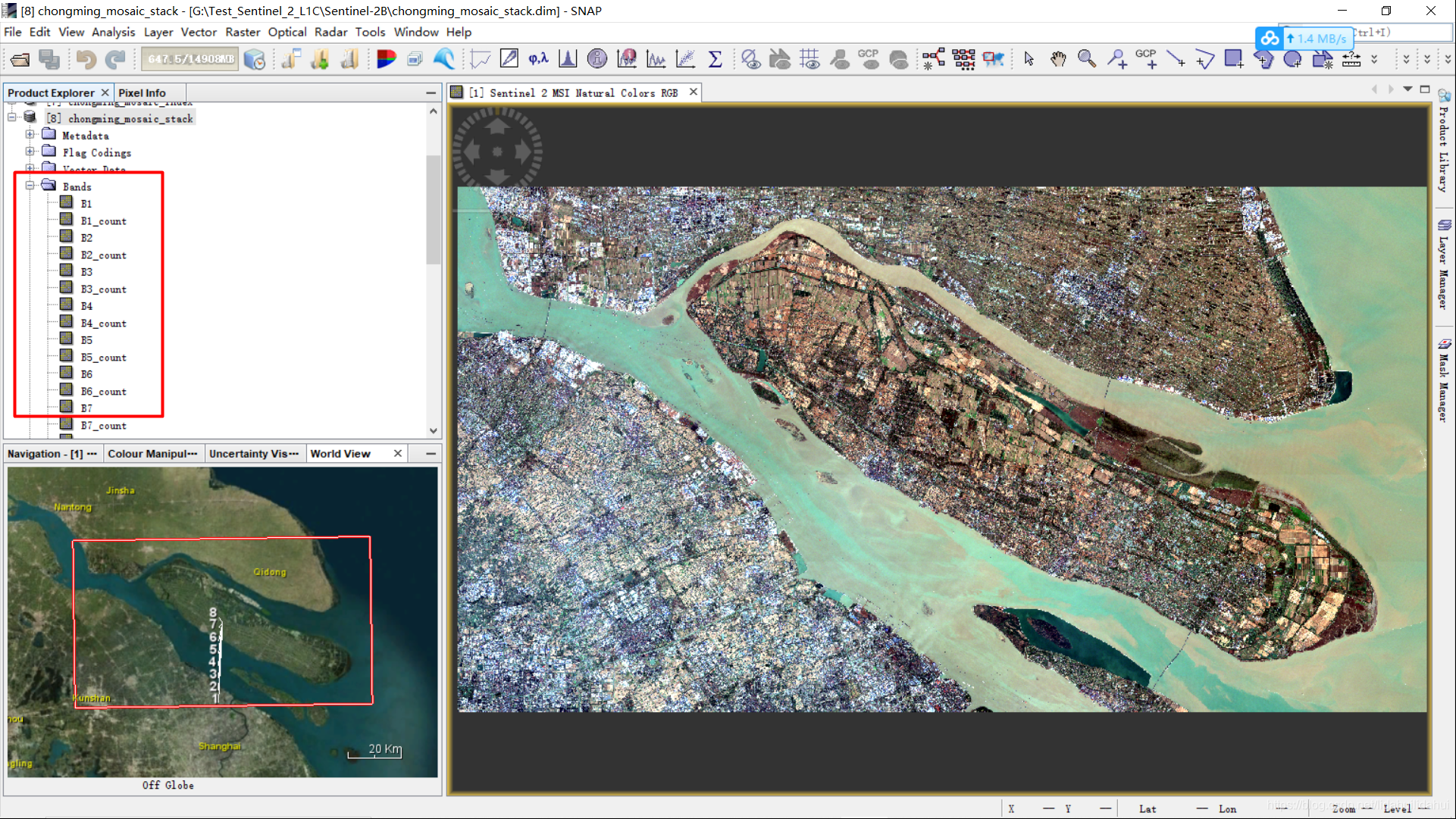
确认无误后点击Run,由于特征数量比较多,需要等待更多的时间,大概一个小时。完成后的效果(奇怪的是它似乎会保留原来的波段,后来发现这些count和flags标记都是某种掩膜标记,不过不影响后续操作,只是占了不少内存):
BandSelect工具
前面,建立的特征工程中含有较多的无用波段,可以借助BandSelect工具将它们去掉:
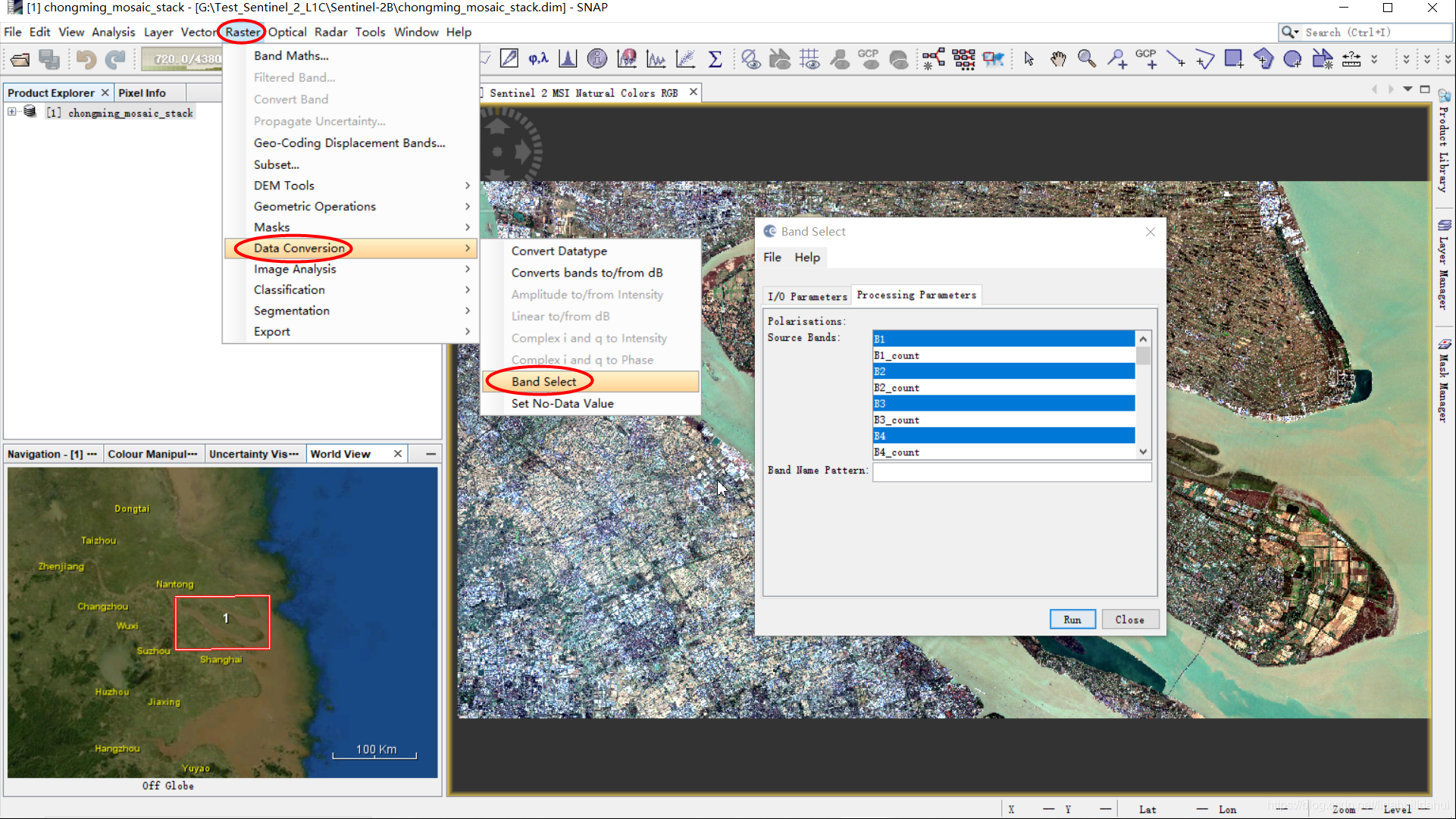 在Processing Paramters参数面板中挑选对应的波段(带“count”和“flags”字眼的波段的都不选)。
在Processing Paramters参数面板中挑选对应的波段(带“count”和“flags”字眼的波段的都不选)。
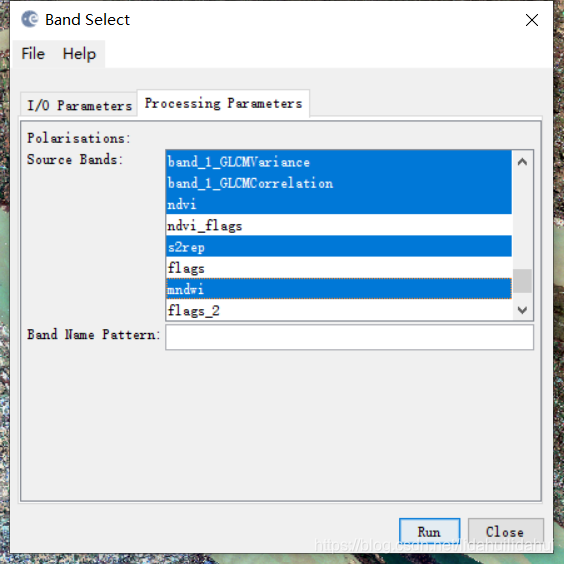
I/O Paramters修改输出的文件名和输出文件夹(这里只修改了输出文件夹)。
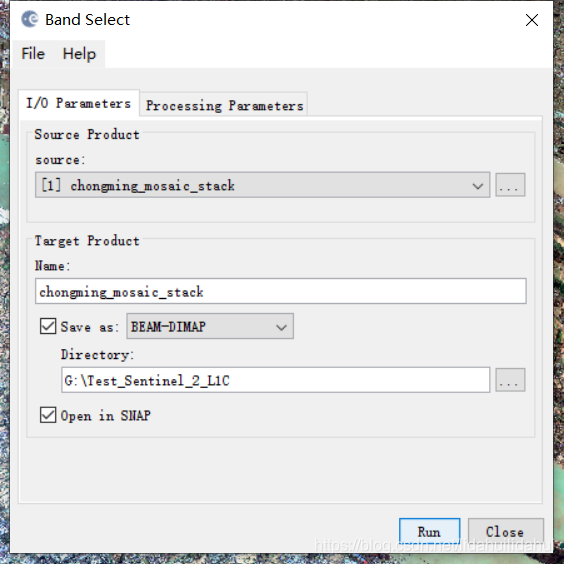
确认无误后,点击Run,耐心等待二十多分钟。打开处理后的波段(例如打开B1波段),会碰到一个错误(原因是我们删掉count和flags与掩膜有关的波段,而元数据数据仍然保留了这两种标记,很多时候图像读取出错都是在元数据出现问题,多数时候可以通过修改元数据得以解决,例如坐标系丢失或错误等):
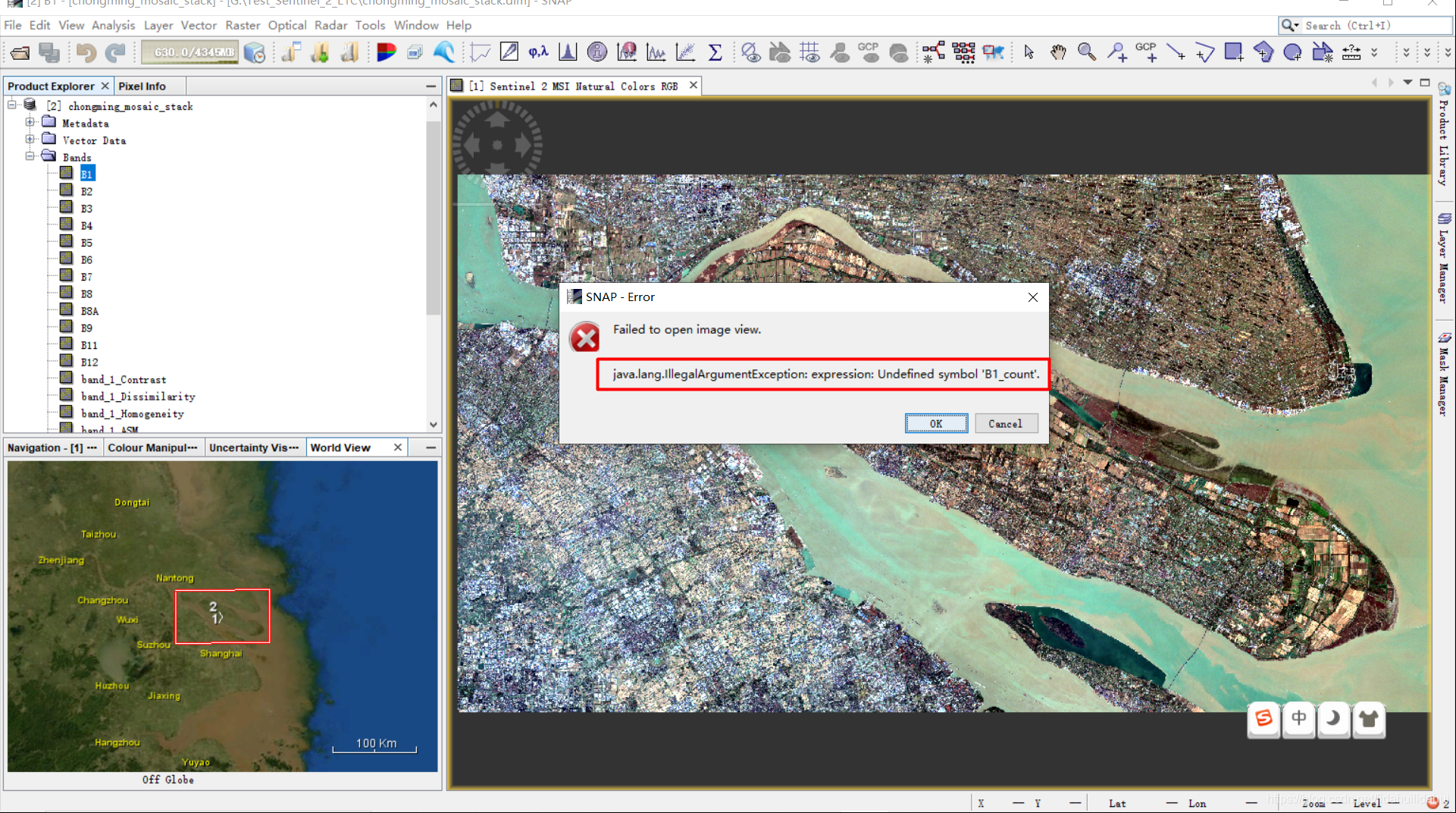
我们通过修改元数据文件(chongming_mosaic_stack.dim),来消除这个错误:
先关闭这个数据集:
找到其元数据文件(chongming_mosaic_stack.dim),.dim文件为BEAM-DIMAP格式(这种格式最初来自于法国Spot卫星,后来由欧空局进一步改进,最后成为SNAP默认数据格式 ),.dim元数据文件可以记事本(写字板等编辑器打开):
.dim文件可以看到其本质上一个.xml文件(尽管尚不清欧空局作了何种修改):
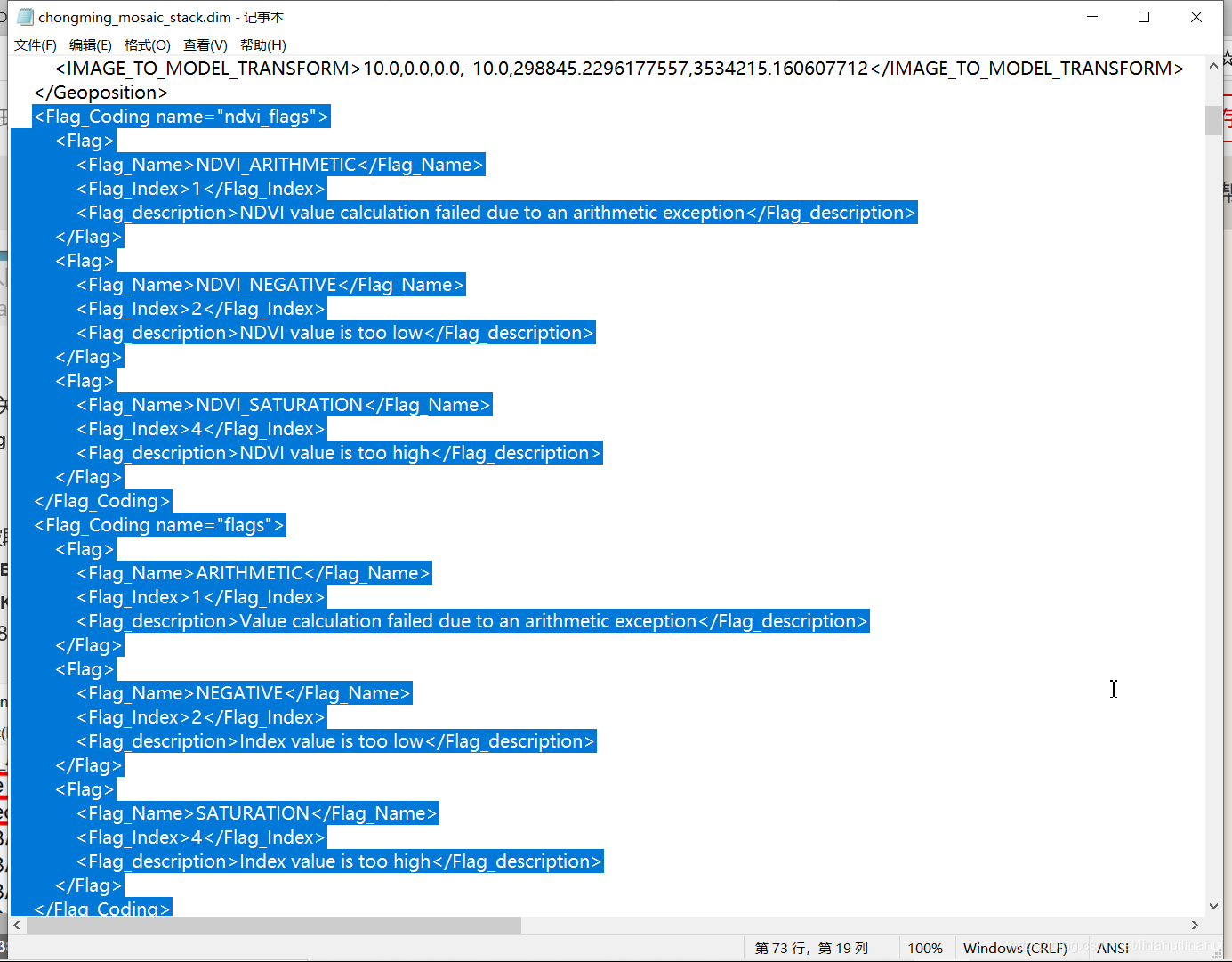
找到flags相关的节点内容(<Flag_Coding name=“ndvi_flags”>,<Flag_Coding name=“flags”>),删掉:
对于count波段的处理,见下图(先找到<Image_Interpretation>下的<Spectral_Band_Info>节点,然后处理报错节点<VALID_MASK_TERM >),需要处理B1, B2,B3,B4,B5, B6,B7,B8,B8A,B9, B11,B12共计12个波段:
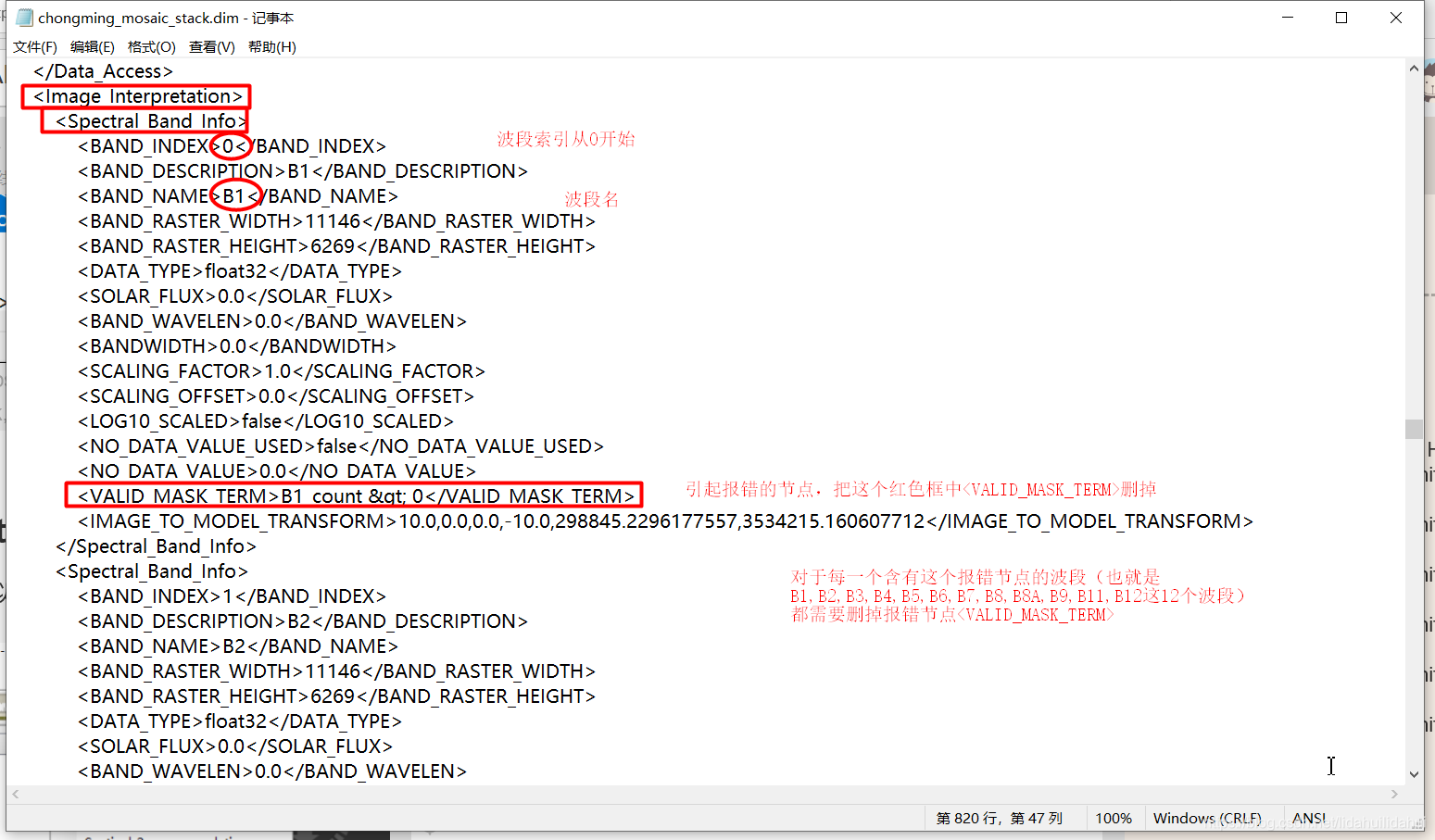
修改后,点击保存。回到SNAP,重新打开这个数据集,并打开B1波段,不再出现报错提示:
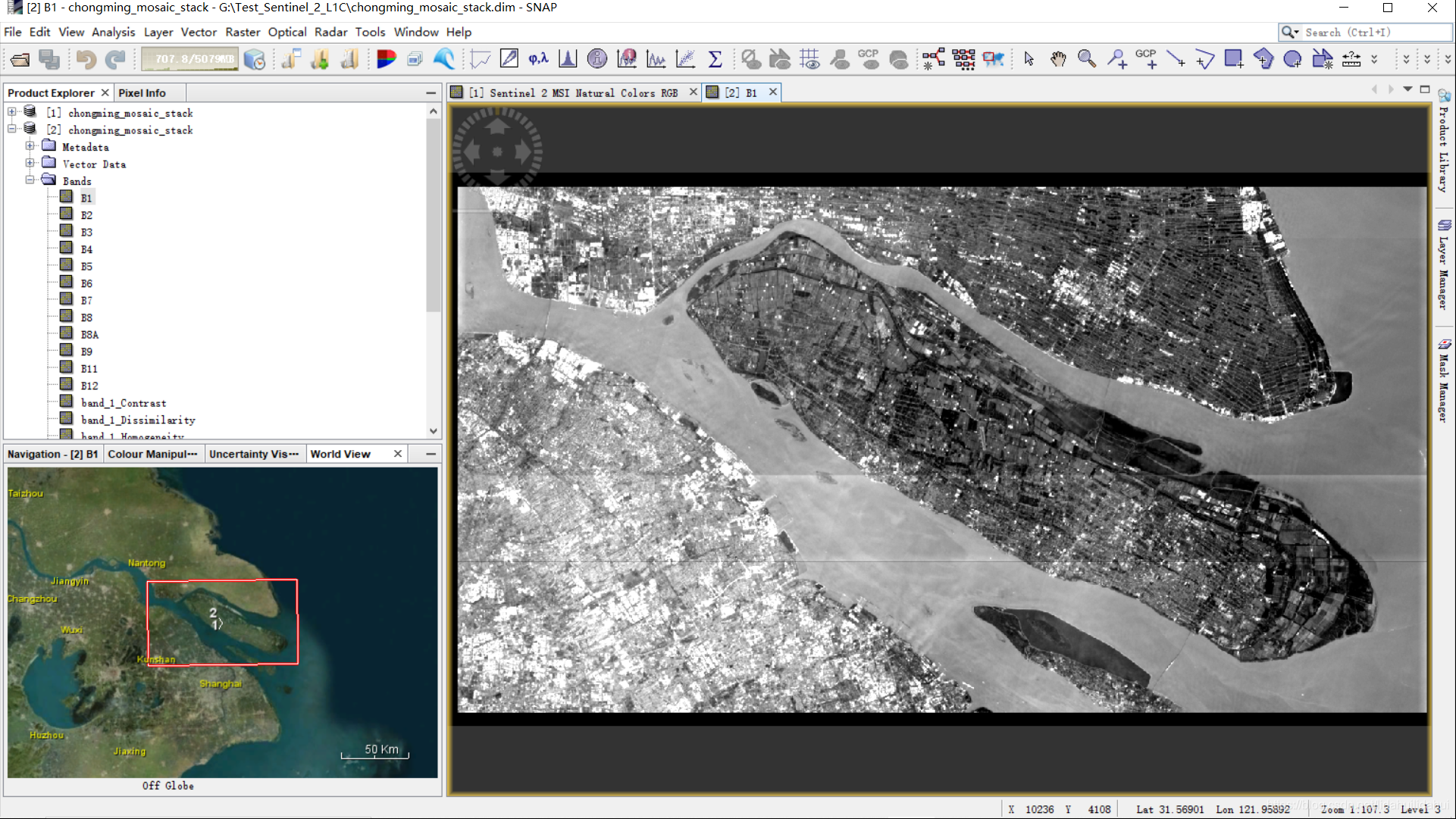
OK,pefect!
一个Sentinel-2监督分类影像特征工程(12个光谱特征,10个纹理特征,4个指数特征集,合计26个特征)构建完毕!接下来,我们利用SNAP的随机森林算法进行分类(见下一篇博客)。
最后,希望你能有所收获!
结语
这次我们利用目前免费的遥感影像质量最高的Sentinel-2影像构建了一个用于分类的特征工程数据集,涵盖了许多遥感影像的基本处理。下篇博客再利用其来进行监督分类。尽管SNAP软件在某些功能上还不够完善,利如这里的PCA变换,波段叠加不够直观。但是,SNAP允许你自由添加插件,只要你弄清其接口(gpt命令行工具,Python,Java,bat脚本,sh脚本等),那么你可以为之添加丰富的功能。并且SNAP在不断地迭代更新过程中,有理由相信其会变得越来越好,在其庞大的开源社区群体以及欧空局等官方机构的支持下。最后,如果你对SNAP处理遥感数据感兴趣的话,也可以加入,博主创建的欧空局SNAP处理交流群:665903216。祝好!
参考文献
[1] 张磊, 宫兆宁, 王启为, 金点点, 汪星. Sentinel-2影像多特征优选的黄河三角洲湿地信息提取. 遥感学报, 2019,23(2):313-326.
[2] 朱文泉,林文鹏. 遥感数字图像处理----原理与方法[M]. 北京:高等教育出版社,2016.
[3] Orfeo Toolbox(OTB) 7.0.0 doc文档:https://www.orfeo-toolbox.org/CookBook/Applications/app_DimensionalityReduction.html?highlight=pca
