(1+1)-ES与传统的进化策略的区别
与传统的进化策略相比区别较大,它没有初始化种群的一步,而是初始化一条父染色体,并且在整个寻优过程中,只有一条父染色体和一条由其变异生成的子染色体存在。
(1+1)-ES具体步骤
问题:求
在
上的最大值。
步骤:
1、随机初始化一条父染色体,称其为
,且其上只含有一个基因(表示
值)。
2、将
进行变异操作得到子染色体
。
3、求
和
的适应度(即
对应的
值)。
4、如果
的适应度大于
的,则用
代替
,否则保留
。
5、根据
更新变异强度。
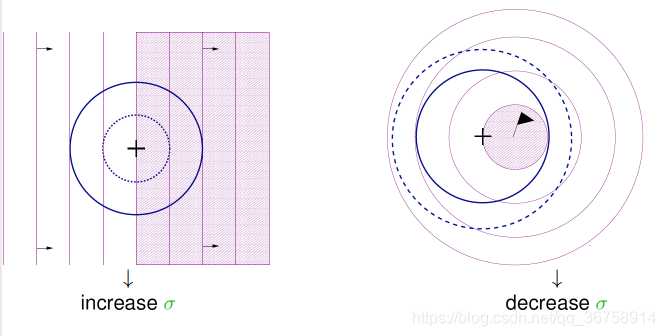
是在
年由
提出的,如下图所示:还没到收敛的时候(下面左图), 增大变异强度可以使其更快收敛; 如果已经快到收敛了(下面右图), 则需要减小变异强度防止在最优点处震荡。那么该如何判断是否快到收敛了呢?就是如果有
的变异比原始的
好的话,就是快收敛了(下面右图)。而在左图中, 有一半的可能性比原始
好,另一半则比原始
差,所以还没到收敛。
 用一个公式概括为:
用一个公式概括为:
其中
。
在
中,如果
优于
,则
,否则
。
(1+1)-ES的python程序
import numpy as np
import matplotlib.pyplot as plt
DNA_SIZE = 1 # DNA (real number)
DNA_BOUND = [0, 5] # solution upper and lower bounds
N_GENERATIONS = 200
MUT_STRENGTH = 5. # initial step size (dynamic mutation strength)
def F(x): return np.sin(10*x)*x + np.cos(2*x)*x # to find the maximum of this function
# find non-zero fitness for selection
def get_fitness(pred): return pred.flatten()
def make_kid(parent):
# no crossover, only mutation
k = parent + MUT_STRENGTH * np.random.randn(DNA_SIZE)
k = np.clip(k, *DNA_BOUND)
return k
def kill_bad(parent, kid):
global MUT_STRENGTH
fp = get_fitness(F(parent))[0]
fk = get_fitness(F(kid))[0]
p_target = 1/5 # 新产生的子染色体有1/5的概率要优于父染色体
if fp < fk: # kid better than parent
parent = kid
ps = 1. # kid win -> ps = 1 (successful offspring)
else:
ps = 0.
# adjust global mutation strength
MUT_STRENGTH *= np.exp(1/np.sqrt(DNA_SIZE+1) * (ps - p_target)/(1 - p_target))
return parent
parent = 5 * np.random.rand(DNA_SIZE) # parent DNA
for _ in range(N_GENERATIONS):
# ES part
kid = make_kid(parent)
py, ky = F(parent), F(kid) # for later plot
parent = kill_bad(parent, kid)
