
我们之前说到, 一般的遗传算法使用的 DNA 是二进制编码的, 爸妈的 DNA 通过交叉配对, 组成宝宝的 DNA, 宝宝也会通过一定的变异获得新的功能. 但一般的进化策略却有些不同。
进化策略
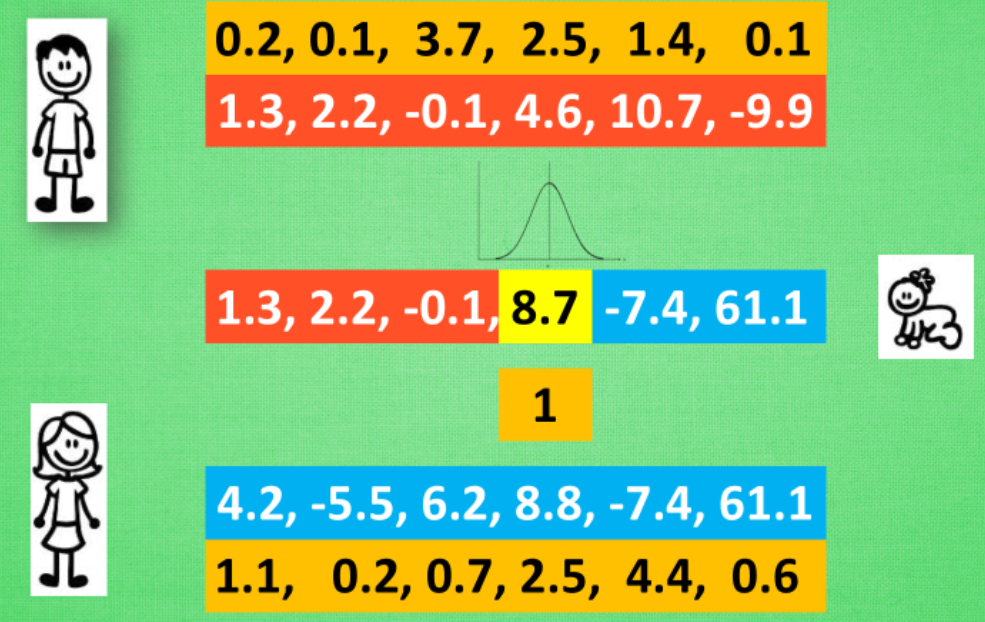
爸妈的 DNA 不用再是 01 的这种形式, 我们可以用实数来代替, 因为我们抛开了二进制的转换问题, 从而能解决实际生活中的很多由实数组成的真实问题. 比如我有一个关于 x 的公式, 而这个公式中其他参数, 我都能用 DNA 中的实数代替, 然后进化我的 DNA, 也就是优化这个公式啦. 这样用起来, 的确比遗传算法方便.
同样, 在制造宝宝的时候, 我们也能用到交叉配对, 一部分来自爸爸, 一部分来自妈妈. 可是我们应该如何变异呢? 遗传算法中简单的随机选择然后取逆做法貌似在这里行不通. 不过进化策略中的另外一个因素起了决定性的作用. 这就是变异强度. 简单来说, 我们将爸妈遗传下来的值看做是正态分布的平均值, 再在这个平均值上附加一个标准差, 我们就能用正态分布产生一个相近的数了. 比如在这个8.8位置上的变异强度为1, 我们就按照1的标准差和8.8的均值产生一个离8.8的比较近的数, 比如8.7. 然后对宝宝每一位上的值进行同样的操作. 就能产生稍微不同的宝宝 DNA 啦. 所以, 变异强度也可以被当成一组遗传信息从爸妈的 DNA 里遗传下来. 甚至遗传给宝宝的变异强度基因也能变异. 进化策略的玩法也能多种多样.
我们总结一下, 在进化策略中, 可以有两种遗传被继承给后代, 一种是记录所有位置的均值, 一种是记录这个均值的变异强度, 有了这套体系, 我们就能更加轻松自在的在实数区间上进行变异了. 这种思路甚至还能被用在神经网络的参数优化上, 因为这些参数本来就是一些实数.
和遗传算法的异同
遗传算法 (后面简称 GA) 和 ES 真差不多.不过我具体的列出来, 方便看清楚.
| 遗传算法(GA) |
进化策略(ES) |
| 选好父母进行繁殖 | 先繁殖, 选好的孩子 |
通常用二进制编码 DNA(例如:11010010) |
通常 DNA 就是实数, 比如 1.221 |
| 通过随机让 1 变成 0 这样变异 DNA | 通过正态分布(Normal distribution)变异 DNA |
具体来说, 传统的 GA 的 DNA 形式是这样:
DNA=11010010
而传统的 ES DNA 形式分两种, 它有两条 DNA. 一个 DNA 是控制数值的, 第二个 DNA 是控制这个数值的变异强度. 比如一个问题有4个变量. 那一个 DNA 中就有4个位置存放这4个变量的值 (这就是我们要得到的答案值). 第二个 DNA 中就存放4个变量的变动幅度值.
DNA1=1.23, -0.13, 2.35, 112.5 可以理解为4个正态分布的4个平均值.
DNA2=0.1, 2.44, 5.112, 2.144 可以理解为4个正态分布的4个标准差.
所以这两条 DNA 都需要被 crossover 和 mutate.
代码
import numpy as np
import matplotlib.pyplot as plt
import matplotlib
matplotlib.use('TkAgg')
DNA_SIZE = 1 # DNA (real number)
DNA_BOUND = [0, 5] # solution upper and lower bounds
N_GENERATIONS = 200
POP_SIZE = 100 # population size
N_KID = 50 # n kids per generation
# to find the maximum of this function
def F(x):
return np.sin(10*x)*x + np.cos(2*x)*x
# find non-zero fitness for selection
def get_fitness(pred):
return pred.flatten()
# 我们随机找到一对父母, 然后将父母的 DNA 和 mut_strength 基因都 crossover 给 kid.
# 然后再根据 mut_strength mutate 一下 kid 的 DNA. 也就是用正态分布抽一个 DNA sample. 而且 mut_strength 也能变异.
# 将变异强度变异以后, 他就能在快收敛的时候很自觉的逐渐减小变异强度, 方便收敛.
# 返回为 kids 的字典,其中包含了种群中新生成的 n_kid 个个体。
def make_kid(pop, n_kid):
# 1、生成n_kid个孩子的空的DNA和变异强度mut_strength
kids = {'DNA': np.empty((n_kid, DNA_SIZE))} # (50,1)
kids['mut_strength'] = np.empty_like(kids['DNA']) # (50,1)
# 更新生成的这50个kids['DNA']和['mut_strength']值
for kv, ks in zip(kids['DNA'], kids['mut_strength']):
# 2、交叉crossover
# 2.1、随机在种群中选择两个个体作为父母p1,p2
p1, p2 = np.random.choice(np.arange(POP_SIZE), size=2, replace=False)
# 2.2、随机选择交叉点crossover points(cp),这个交叉点是一个随机的布尔值
cp = np.random.randint(0, 2, DNA_SIZE, dtype=np.bool)
# 2.3、交叉点cp为True的时候,kv取p1的值,否则取p2的值
kv[cp] = pop['DNA'][p1, cp]
kv[~cp] = pop['DNA'][p2, ~cp]
# 交叉点cp为True的时候,ks取p1的值,否则取p2的值
ks[cp] = pop['mut_strength'][p1, cp]
ks[~cp] = pop['mut_strength'][p2, ~cp]
# 3、基于正态分布变异:mutate
# 将 ks 中所有的元素加上以 0.5 为均值、大小与 ks 相同的随机数数组,然后取结果与 0 的最大值,这个值将作为下一步对 kv 进行变异的系数。这里加上 0.5是为了使加上随机数后不会出现负数,在保证系数不为负数的前提下,引入随机变化。
ks[:] = np.maximum(ks + (np.random.rand(*ks.shape)-0.5), 0.)
# 将 kv 中的每个元素都加上以0为均值、ks 为标准差的正态分布随机数,完成基因变异的操作(变异过程中,每个位点的变异强度也在进行随机变化)。注意到这里使用的是 * 运算符,如果 kv 是一个 n 维数组,则这个运算将作用于所有维度上的元素,而且还需要 numpy 广播机制的支持。
kv += ks * np.random.randn(*kv.shape)
kv[:] = np.clip(kv, *DNA_BOUND) # clip the mutated value
return kids
## 根据适应度 fitness 选择适应度最靠前的一些人, 抛弃掉那些适应度不佳的人. 这个很简单.
def kill_bad(pop, kids):
# put pop and kids together
for key in ['DNA', 'mut_strength']:
pop[key] = np.vstack((pop[key], kids[key]))
fitness = get_fitness(F(pop['DNA'])) # calculate global fitness
idx = np.arange(pop['DNA'].shape[0])
good_idx = idx[fitness.argsort()][-POP_SIZE:] # selected by fitness ranking (not value)
for key in ['DNA', 'mut_strength']:
pop[key] = pop[key][good_idx]
return pop
# 我们用一个 python 字典来存这两种 DNA 的信息. 这里 DNA 存的是均值, mut_strength 存的是标准差.
# 因为最优点只有一个, 所以我们的 DNA 的长度就只有一个. 我们用一个 python 字典来存这两种 DNA 的信息. 这里 DNA 存的是均值, mut_strength 存的是标准差.
pop = dict(DNA = 5 * np.random.rand(1, DNA_SIZE).repeat(POP_SIZE, axis=0), # initialize the pop DNA values (100,1)
mut_strength = np.random.rand(POP_SIZE, DNA_SIZE)) # initialize the pop mutation strength values (100,1)
plt.ion()
x = np.linspace(*DNA_BOUND, 200)
plt.plot(x, F(x))
for _ in range(N_GENERATIONS):
if 'sca' in globals():
sca.remove()
sca = plt.scatter(pop['DNA'], F(pop['DNA']), s=200, lw=0, c='red', alpha=0.5)
plt.pause(0.05)
# ES part
# 生成新的种群
kids = make_kid(pop, N_KID)
# 保存优秀的个体
pop = kill_bad(pop, kids)
plt.ioff()
plt.show()3.1 进化策略 Evolution Strategy_哔哩哔哩_bilibili