目的:将所关心的对象按照一定的规则或者标准,分成不同的类别,以便有针对性的进行进一步有效处理。(利用数理统计方法对数据的变量或者观测进行分类)
1、概述
分类
- 模糊聚类:对象与类别的从属关系是有一定概率的
- 非模糊聚类:属于或者不属于,聚类的对象为离散的
1.1 聚类分析方法比较
- 层次法(CLUSTER)
- 凝聚式:先将每个观测都归为一类,每次将最相似的2个类合并成一个新类,直到所有观测都成为一个类或者到达设定条件停止(SAS中的层次法都是凝聚式)
- 分裂式:与凝聚式相反,先将所有观测归为一类,再按相似程度一分为二,直到每个观测自成一类,或者达到设定条件停止
- 划分法
- 先指定某几个类中心,通过计算每个样本到中心的距离将其归到最近的类,然后不断调整类中心直到收敛。

- 衡量聚类分析好坏的标准:
- 同类间,对象高度相似
- 不同类间,对象几乎没有相似性
1.2 如何度量相似性
定义一个距离函数:距离越小 越相似,定义距离的合理与否,从根本上决定了聚类分析的效果。
常见的距离函数:
- 街区距离
- 欧式距离
- 闵可夫斯基距离
- 汉明距离
SAS允许用户自定义距离函数,经距离函数计算的结果可以作为sas聚类分析过程的数据。

/*聚类分析 对10只股票的10到13年间的收益率进行聚类 1、首先计算每2只股票的距离*/ data work.stock; title "股票数据输入"; length stock $3; input stock $ div_2010 div_2011 div_2012 div_2013; datalines; s1 8.4 8.2 8.4 8.1 s2 7.9 8.9 10.4 8.9 s3 9.7 10.7 11.4 7.8 s4 6.5 7.2 7.3 7.7 s5 6.5 6.9 7.0 7.2 s6 5.9 6.4 6.9 7.4 s7 7.1 7.5 8.4 7.8 s8 6.7 6.9 7.0 7.0 s9 6.7 7.3 7.8 7.9 s10 5.6 6.1 7.2 7.0 ; proc distance data=work.stock method=DCORR /*指定使用欧式距离*/ out=work.distdcorr; var interval(div_2010 div_2011 div_2012 div_2013); id stock; title "股票距离计算"; run; proc print data = work.distdcorr; title '打印work.distdcorr'; run;
2 层次法与划分法
2.1 使用过程FASTCLUS (划分法)实现K均值聚类法
划分法中最常用的是K均值聚类法。K均值聚类法的一个重要特点是算法收敛的时间和待分析数据的观测数据成正比。K均值聚类法可以用来处理规模较大的数据。
K均值聚类法的步骤:
- 选定K个观测作为K类的种子
- 读入所有观测,计算每个观测与K个种子间的距离,并将观测暂时归类到其距离最近的种子所在的类
- 根据最新类的中的观测,重新计算类的的中心
- 重复2-3步,直到收敛。至此所有K类的种子最终确定
- 再次读入所有观测,将每个观测归类到与其距离最近的种子所在的类,分类结束。
这里的K值是预估的,预估的方法有:
- 根据背景知识进行判断
- 根据分类目标判断,比如希望客户被分成3-4类
- 作图法:根据图像来判断
- 启发式:先给出一个较大的K值,在聚类得到一个建议类数,然后根据这个建议类数,进行聚类分析直到K值稳定在某一个常数
种子的选择从根本上决定类分类的好坏
SAS 通过proc fastclus 来实现K均值聚类,默认使用欧式距离来计算观测之间的距离。
FASTCLUS具有以下特点:
- 适用于分析较大的数据集,100条以上
- 方差较大的变量对分析结果的作用也比较大。如果数据集变量方差之间的差异较大,可以考虑先对数据集进行标准


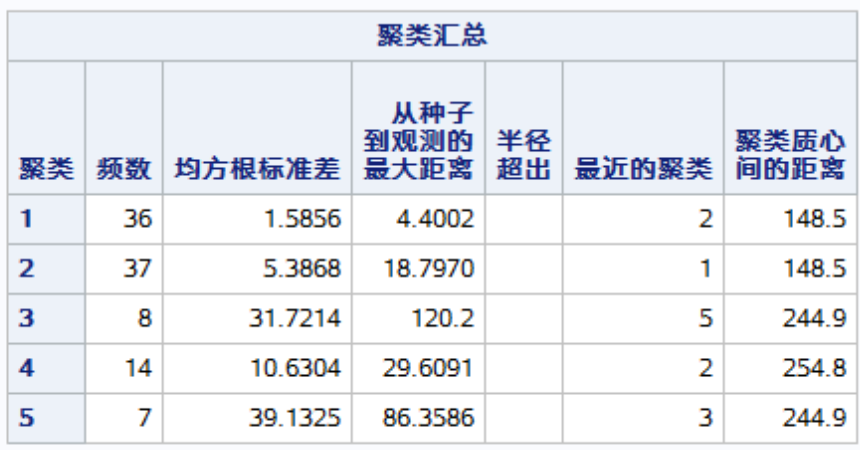
/*划分法与层次法*/ data sasuser.food_cal; set tmp1.food_cal; run; proc fastclus data=sasuser.food_cal maxc=5 /*运行生成分类数据的最大值,默认100*/ maxiter=10 /*重新计算聚类中心的最大迭代次数*/ out=work.clus; var kcal fat protein;/*三个变量进行聚类*/ title "使用fastclus 进行k均值聚类法分析"; run;
运行结果如下:
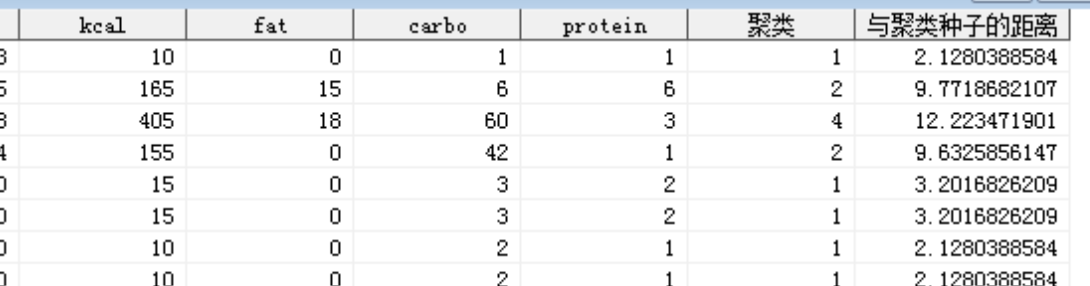
最终输出数据集work.clus中可以看到每一条观测所属的具体类:
2.2使用过程CLUSTER实现层次法
SAS提供了11种层次法,可以通过指定proc cluster中的选项来实现,形式如下:

proc cluster中,使用不同的方法起聚类结果也会不一样,总结如下:
1、在蒙特卡罗随机模拟方法中,分类效果比较好的是“可变法”,分类效果比较差的是“MCQUITTY”和“最短距离法”
2、对离群点比较敏感的是“离差平方和”和“最长距离法”
3、对离群点比较不敏感的是“类平均法”和“重心法”
4、除了“最大似然法”之外,其他方法均支持距离数据集作为过程步的输入
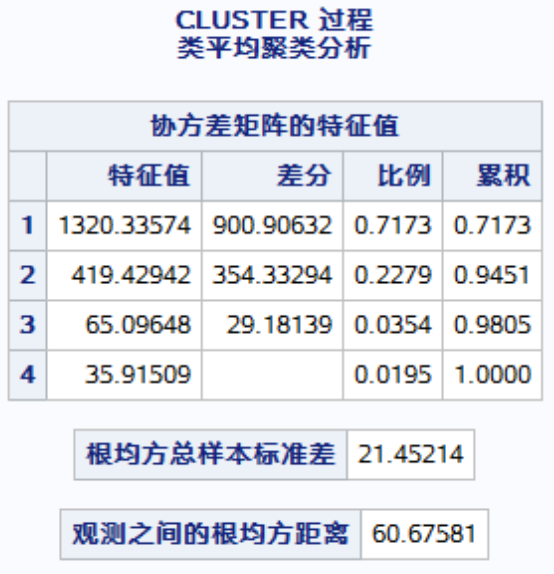
/*使用过程cluster实现层次法*/ data sasuser.nutrition; set tmp1.nutrition; run; proc cluster data=sasuser.nutrition outtree=work.tree /*使用类平均 层次法,输出ccc的值,输出伪f统计量和伪T统计量*/ method=ave ccc pseudo; var Magnesium_mg percent_water Protein_g Saturate_Fat_g; id food; title "使用类平均 层次法进行对营养成分进行聚类分析"; run;
下面分析下运行的结果:
cluster输出了类平均聚类分析图
还有聚类的历史:
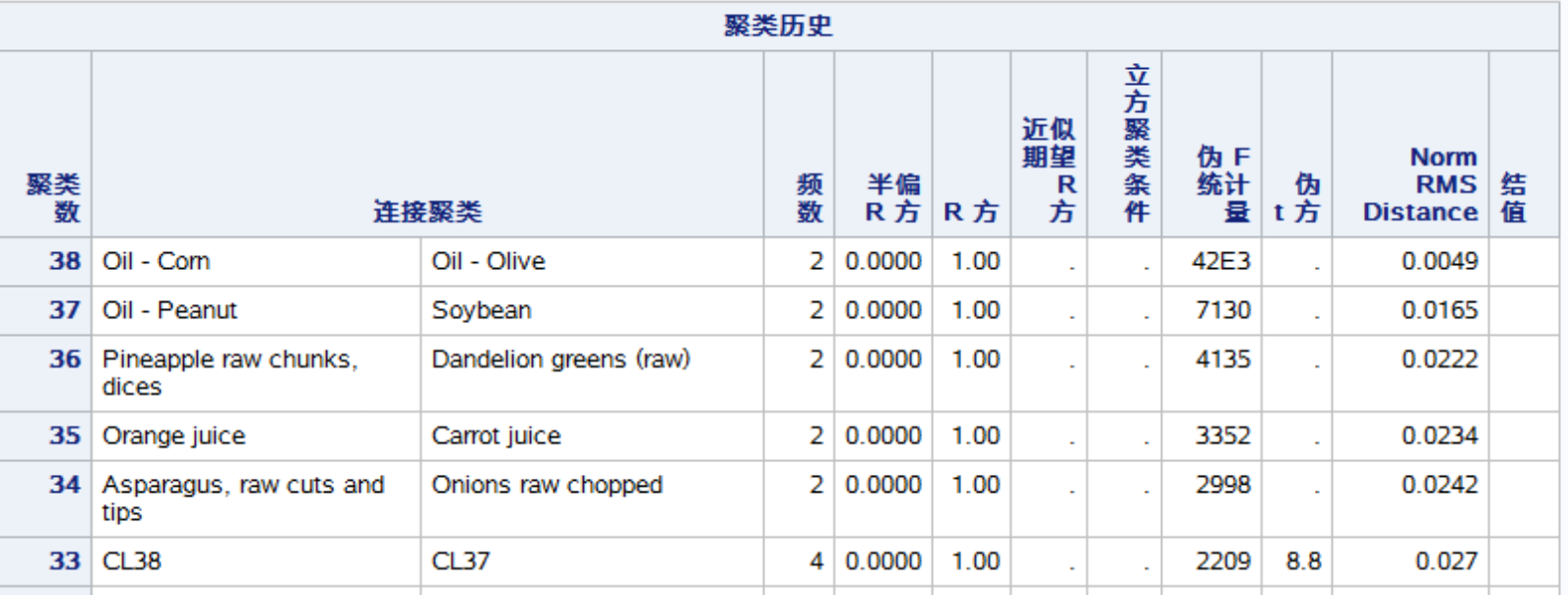
其中:第一列表示当前的聚类数,第二三列表示当前合并的聚类,第四列表示当前类包含的观测数。第5-6列表示半偏R方和R方,R方越大表示类之间分的越开,聚类效果越好;若半偏R方较大,说明本次并类的效果不好,应该考虑聚类在上一步停止。第8列现实的是CCC(立方聚类条件)的值,ccc的峰值表示建议聚类数;第9列显示的是伪F统计量,表示类与类之间的分离程度。越大表示分类效果越好。outtree=work.tree 生成类数据集用来生成树形图。
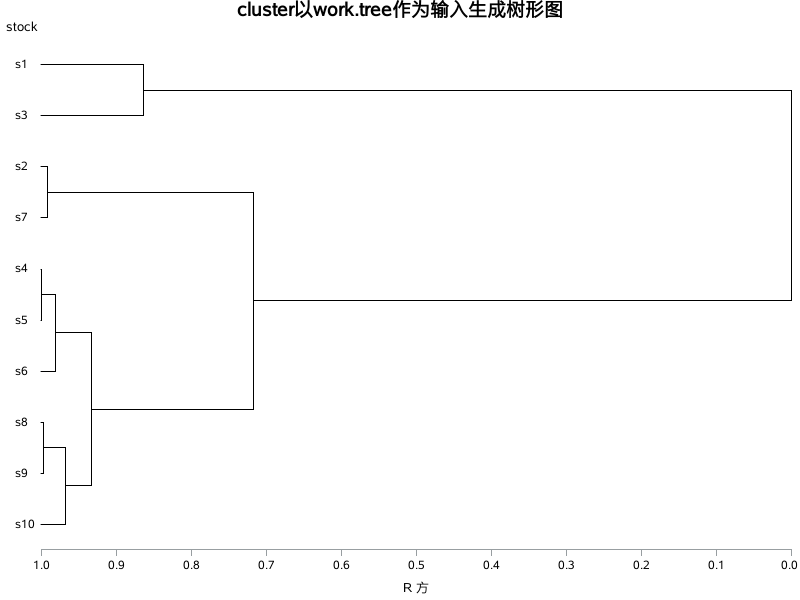
/*cluster以work.tree作为输入,生成树形图*/ proc cluster data=work.distdcorr method=ward outtree=work.tree; id stock; run; axis1 order=(0 to 1 by 0.1); proc tree data=work.tree haxis=axis1 horizontal; height _rsq_; id stock; title "cluster以work.tree作为输入生成树形图"; run;
axis1 order=(0 to 1 by 0.1) 定义了一条坐标轴的属性,haxis=axis1使用该坐标轴,horizontal指定了输出水平树形图。
height _rsq_ 指定了输入数据集中的变量_rsq_作为横轴,id为变量stock;