基本原理
基于可逆终止的,荧光标记dNTP,做边合成边测序
样本准备 Sample Prep
通过不同实验方法得到的样品,需要先提取样本基因组中的DNA,用超声波将其随机打断。
然后使用酶将两端补平,使用 Klenow 酶在3‘ 端加一个 A 碱基(用于连接接头序列)。
为了后续扩增,测序分析,需要为这些DNA片段添加特定的接头序列(已知的,用于测序识别),大概有三种:
- sequencing binding site(绿)
- index(红,黄)
- 流动池引物互补的序列(蓝,紫)

添加完接头序列后的DNA片段集合叫DNA文库 (DNA library),这样就完成了样品准备工作。
成簇 Cluster Generation
成簇是DNA片段被扩增的过程,该过程在流动池 (Flowcell) 中完成。它是一片带有8条通道(lanes)的玻璃载玻,每个通道内表面附有两种DNA引物。

首先,引物会与样品中的DNA片段的接头序列互补配对,固定在通道表面

通过聚合酶生成杂交片段的互补片段,然后加入NaOH碱溶液后,双链分子变性,原始模板链(左边的链)被流动池中的液体洗去

加入中性液体用于中和碱溶液,剩下的单链拷贝链另一端的接头就会与通道表面的引物结合,形成单链桥。

同样的,在聚合酶参与下,生成互补链,最终形成双链桥

通过变性,DNA分子线性化,变为两个单链拷贝
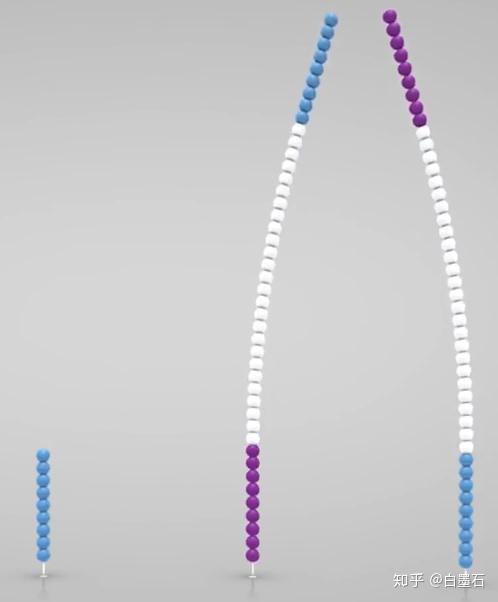
它们又分别与自己配对的引物结合

重复这个循环,同时形成数百万的簇。在这个过程中,所有的DNA片段都会被克隆扩增。
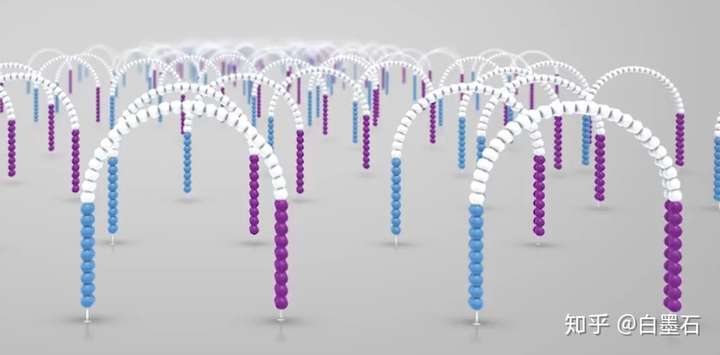
桥式扩增后,反向链会被切断洗去,仅留下正向链。为防止特异性结合重新形成单链桥,3‘端被封锁
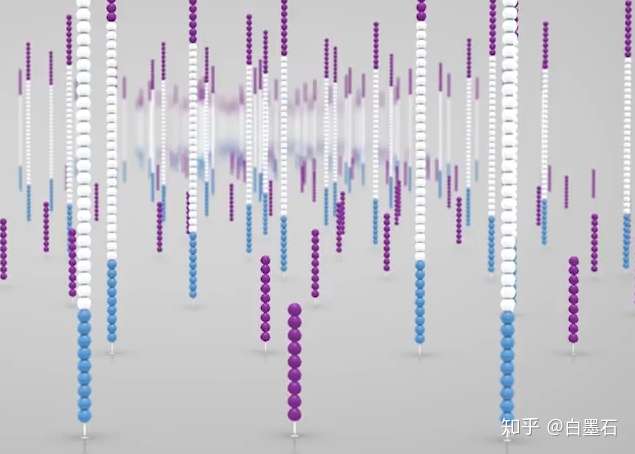
测序 Sequencing
首先,在Flowcell中加入荧光标记的dNTP和酶,由引物起始开始合成子链。
但是dNTP存在 3’端叠氮基会阻碍子链延伸,这使得每个循环只能测得一个碱基。
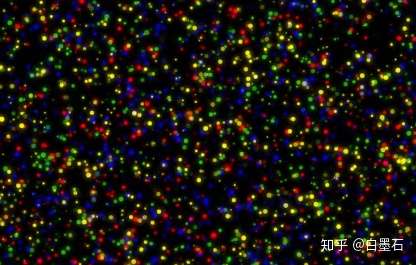
合成完一个碱基后, Flowcell 通入液体洗掉多余的dNTP和酶,使用显微镜的激光扫描特征荧光信号。

荧光发射波长与信号强度一起决定了碱基的读出,所有的DNA片段的一个碱基会被同时读取。在大规模并行的过程中,机器读取的图像类似下面这样
加入化学试剂将叠氮基团与荧光基团切除,然后 Flowcell 再通入荧光标记的dNTP和酶,由引物起始开始合成一个碱基。不断重复这个过程,完成第一次读取。
由于测序仪每次测序时的通量比较大,所以每次测得的序列可能不止一个样本。
为了去区分每个样本及正负链,科学家构建DNA文库时,在接头序列加入了的不同 index(或 barcode)来区分来源。
首先,在完成第一次读取后,复制出的链会被洗去

index 片段引物被引入并与模板杂交,完成序列读取后被洗去。这样读取到的序列与开始时已知的index比对后就可以给测得的序列贴上标签,方便后续分析。

Paired-end测序已经是现在的主流,它提高了测序长度的同时,又可以为结构变异分析提供新方法。要完成双末端测序,首先要将模板链3’去保护,模板折叠,index片段引入

在聚合酶参与下形成双链桥

然后变性,恢复为单链。注意,这次是将正向链切除并洗去,只留下反向链

反向链以测序引物为起始,与正向链类似,经过多个循环后完成读取。
数据分析 Data Analysis
测序完成后会产生数百万个 reads,基于在样品准备时构建的 index 分类来自不同样本的序列。对于每个样品来说,具有相似延伸的碱基被聚在一起。正向和反向read配对生成连续序列。这些序列通过与参考基因组匹配后,实现完整序列的构建。