通过深度学习增强药药反应和药食反应的预测效果
前言
还是不要不务正业了,重新回到DDI的怀抱之中。(笑)
这篇是PNAS(美国科学院)的论文,还是有点分量的……虽然我觉得有点水
本篇文章主要讲的是基于深度学习的药物副作用与药物宇食物的作用的预测。
背景
药与药(drug-drug interaction,DDI),药与食物(drug-food constituent interaction,DFI)之间会产生极其大的反应,其中有很多恶性的反应(adverse drug events, ADE),也有良性的反应。我们药减少恶性的反应并且增加良性的反应。
作者主要描述了基于深度学习的DDI和DFI的预测,模型名为DeepDDI。
方法与实验
作者使用了深度学习的方法进行DDI的预测,将DDI作为86类分类器(具体哪八十六类,可以参考deep-ddi所有数据文件的dataset-S01)。
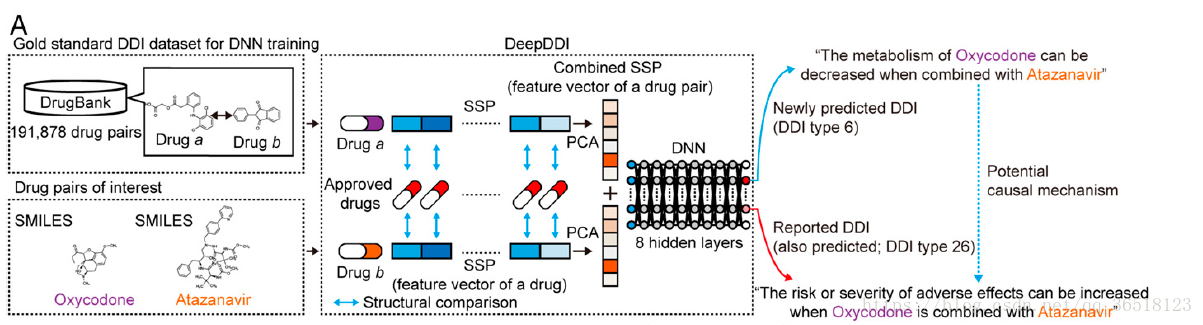
上图是模型主要架构
可以看见,将药物的SMILES(simplified molecular-input line-entry system,简化分子线性输入规范,用于描述一个化学物质的结构)输入,并且可以生成SSP(structural similarity profile,药物的特征向量,可以描述给定药物的独特结构特征)

ps:SSP我google+百度了半天没查到,可能SSP是他们原创的方法
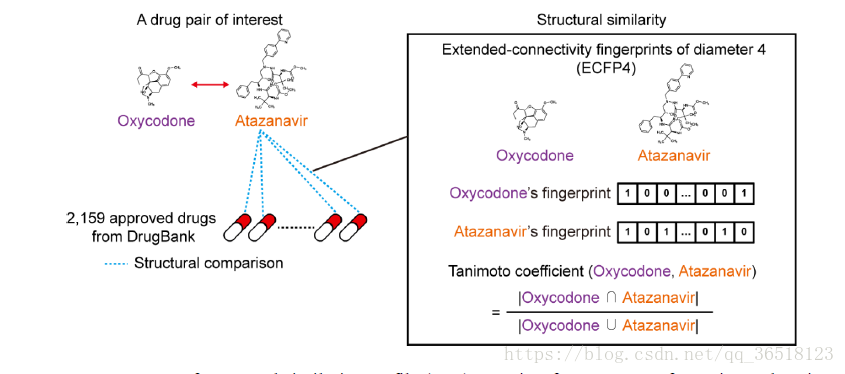
SSP可以对某个单独的药物描述结构性特征。计算方法是与所有的2159个DrugBank中的药物的ECFP4(illustration of ECFP4)经行比对。通过计算对每个药物的ECFP4的Tanimoto系数 来算出SSP。
ECFP4使用Python库RDKit计算的。
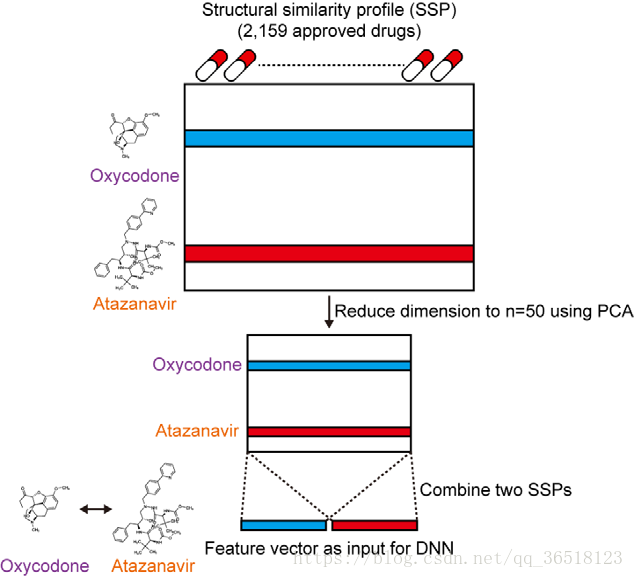
计算出SSP之后,再使用PCA将SSP向量降维到50维。最后将两个50维的PCA输入到DNN中进行多分类。
ps: 作者再这里说PCA降维到10、20、30、40、50维都试验过,最终确定了50维是最好的
DNN我不用多说,注意输入为100维,代表了两种药物PCA降维后的维数和。输出有86种,代表了86类的多分类器。设置了一个阈值,超过这个阈值者就算作有这一类 ,设为1;否则设为0。最优的阈值维0.47
隐藏层的激活函数是ReLU,输出层的激活函数是sigmoid
用了Batch Normalization
Loss function是交叉熵
优化方法是Adam算法
代码层面用了Keras + Tensorflow后台
注意使用了GTX1080GPU运算(其实没啥……就是想到我那破1060就想哭)
训练还是老套路60%,20%,20%的分配,跑100次
DeepDDI最终可以生成人类可读的语句。
比如
羟考酮(oxycodone,阿片类止痛药)与阿扎那韦(atazanavir,抗转录药物)的反应会被描述成
“与阿扎那韦共同作用时,羟考酮的代谢效应会减少”
“当阿扎那韦与羟考酮共同作用时,不利反应的严重性或概率会增加”
优点
较之间的运算方法,DeepDDI有很多优点,比如:
- 其他的很多计算方法并没有很好的描述药与药之间的反应,而DeepDDI可以输出人类可读的语句。
- 其他的很多计算方法需要极其详细的药物信息就比如说药物的靶,有反应的药物,副作用。而DeepDDI是基于SMILES的方法,需要的信息比较单一
- DeepDDI的精度极高,大约92%
- SSP比两种最好的向量生成方法(Molecular Autoencoder, Mol2vec)都更加精确
缺点
- 神经网络的可解释性不强
相关的工作
之前也有基于Deep Learning的DDI的工作。
Drug Drug interaction extraction via convolutional nerual network
Drug-drug Interaction Extraction via Recurrent Neural Network with Multiple Attention Layers
Dependency-based long short term memory network for drug-drug interaction extraction
与本篇不同的是,这些工作很多事基于复杂网络研究的DDI,而本文可以说是基于相识度(化学成分结构)的DDI。
结果
使用的数据是gold standard DDI dataset from DrugBank(deep-ddi所有数据文件的dataset-S02),包含了192284种DDI反应
虽然这个并不是输出一个one-hot向量,但是其实只有406(0.2%)的药物与药物对被检测有两种及以上的反应,而191472(99.8%)的药物与药物对之间只有一种反应。(PS:可不可以最后输出的时候用Softmax而不是Sigmoid,因为只有0.2%的多种反应)
应用场景
原文给出了起码三个应用场景
- 在gold standard DDI dataset中我们发现只有14种DDI类别是明确的有害的类别,其他的DDI情况不明或者是依赖药物施用环境。因此如果我们使用了某种药物(比如用环磷酰胺去治疗癌症)我们可以通过替换他的对应的拥有同样药理效果的药物(贝利司他,氨羟二磷酸二钠,苏灵大)去减少副作用(心脏中毒的概率)。
- 我们可以通过DDI的类型去判断药物是否有“恶性副作用”。如果DNN输出的类型小于阈值0.47,则就视为没有
- 我们可以用于判断药物与食物成分的共同作用(DFI)。一些特定的食物会减少一些药物的效果。就比如说,治疗高血压的三十个药物的体内浓度(这里翻译不好……意会,意会)可以被潜在的食物成分,比如左旋谷氨酸、左谷酰胺、 亚精胺,减少。多价正离子,比如
、
,可以造成药物的治疗失败。
讨论
- 三及多个药物的DDI并没有研究——造成无法研究的原因是数据库的缺乏(如果有这样的数据库的话我认为用RNN/LSTM/GRU可以解)
- DeepDDI的可解释性有待研究(Deep learning的原罪……)
- 很多其他的数据,比如转录组数据可以被使用
我的想法
- PCA有没有更好的解决方案?能不能用其他的方法(比如构造自编码器之类的(瞎编))代替PCA
- DNN的改善方法?可不可以想其他的网络而不是MLP?
- 讨论中的一些问题还有可进步的地方
- 代码我暂时没看,RDKit很难配,可能根据代码出一篇博客
- 看不懂的化学生物问题不要紧,我也不懂(逃ε=ε=ε=┏(゜ロ゜;)┛)
- 这篇的一个特点就是它是基于化学结构与成分的Deep learning based DDI,而不是基于复杂网络的DDI