我正在做一个关于SVM的小项目,在我执行验证SVM训练后的模型的时候,得到的report分数总是很高,无论是召回率(查全率)、精准度、还是f1-score都很高:
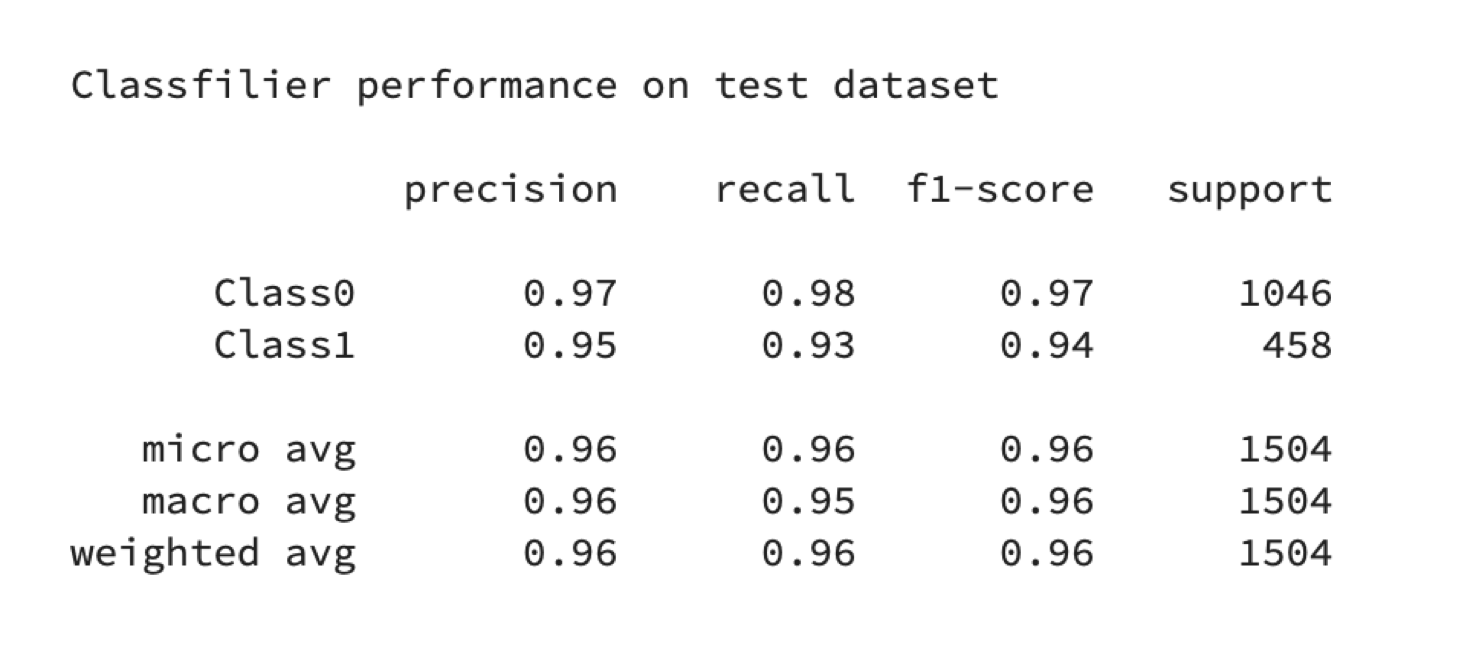
图1 分类器分数report
但是,对于训练的效果就非常差,差到连包含训练集的测试集都无法正确分类,如下图所示,左边是原图像,右边是分类图像,(我标注的标签样本是黄色区域与褐色区域),其中SVC的默认参数为rbf、C=1.0、gamma=“auto_deprecated”,LinearSVC的默认参数为:C=1.0、class_weight=none、dual=true、loss=“squard_hinge”:
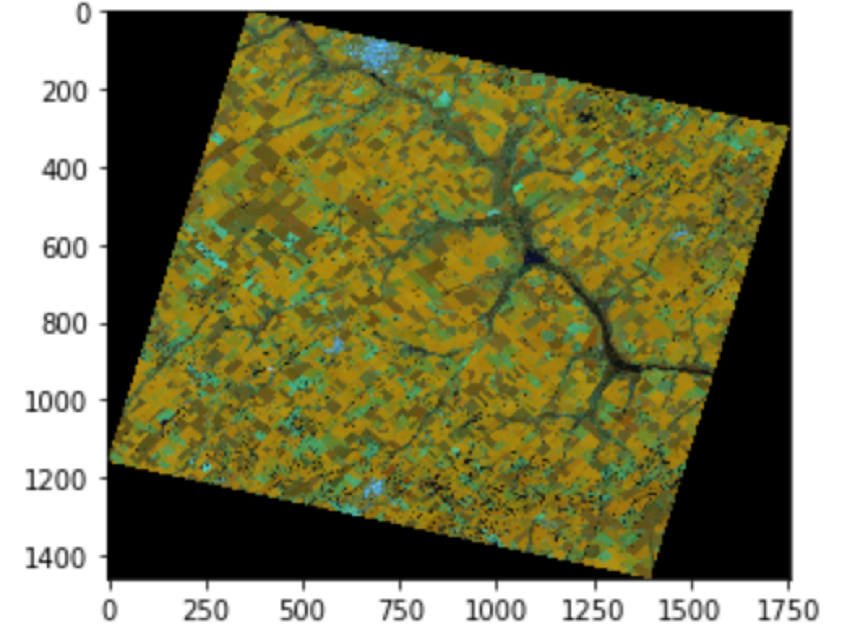
a.原图
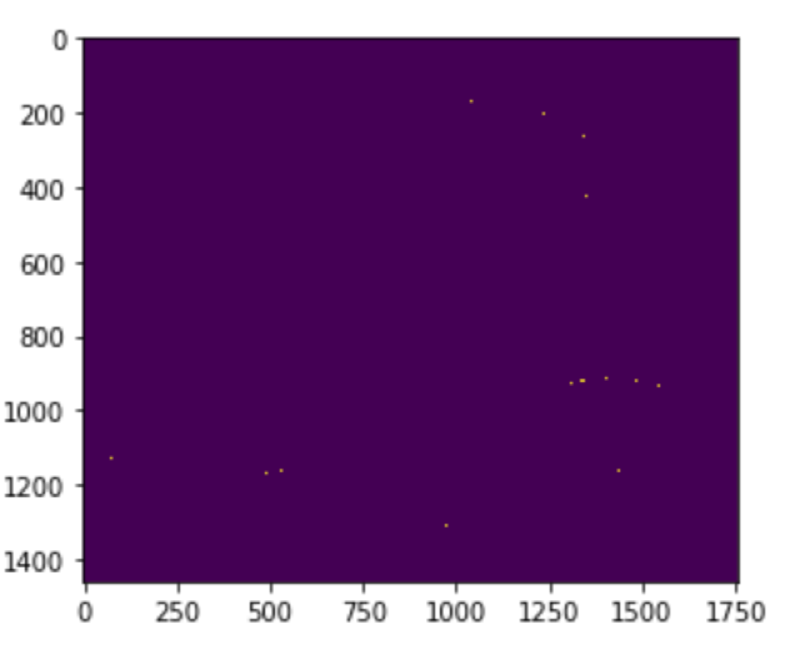
b.SVC(default parameter)
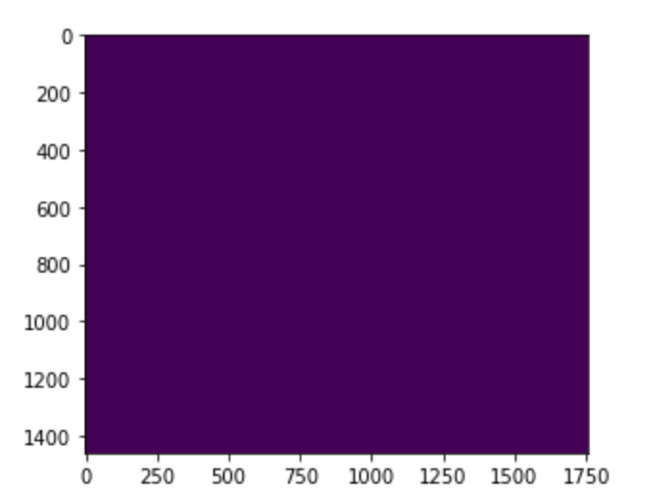
c.LinearSVC(default parameter)
图2. 默认分类效果
由上文可以发现,分类器分类的效果很不好,为了进一步验证这个问题的原因,接下来我分别对LinearSVC和SVC进行参数调整:
1、LinearSVC参数调整
C:使用损失函数是用来对样本的分类偏差进行描述,例如:
由上文可以发现,分类器分类的效果很不好,为了进一步验证这个问题的原因,接下来我分别对LinearSVC和SVC进行参数调整:
1、LinearSVC参数调整
C:使用损失函数是用来对样本的分类偏差进行描述,例如:
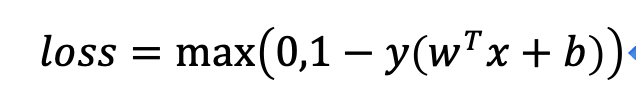
引入松弛变量后,优化问题可以写为:
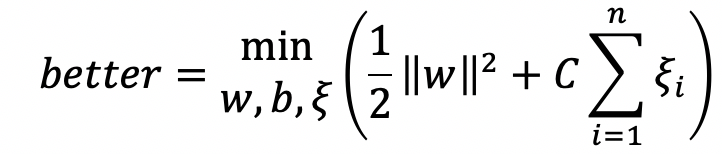
约束条件为:
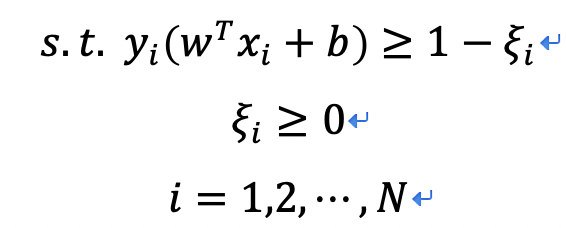

对于不同的C值对于本实例中的分类影响为,如图3所示:
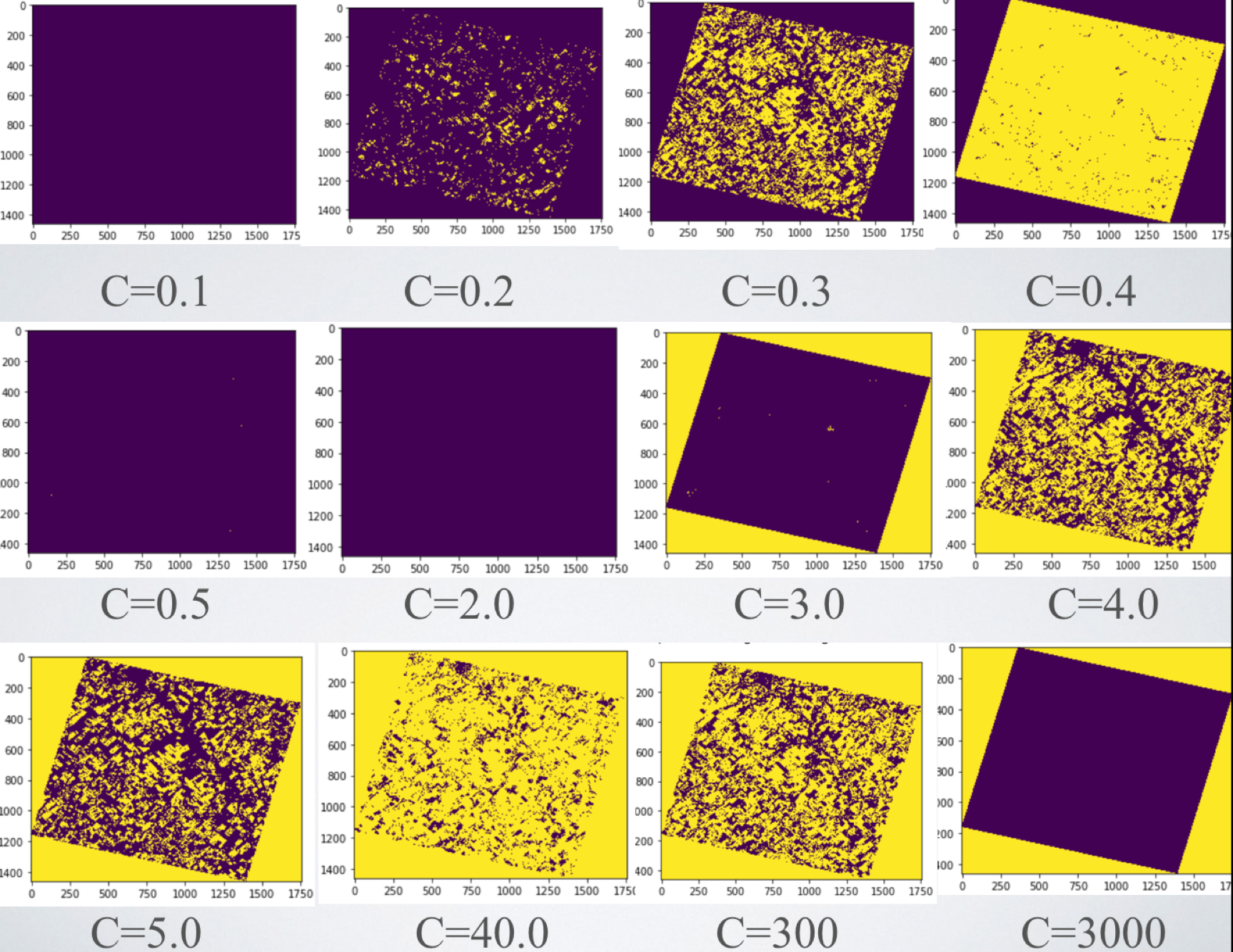
图3. LinearSVC在不同惩罚系数C下的表现
由此可以看出SVC在惩罚系数为0.3、4.0、300时能够较准确根据颜色对图像进行分类。但是作者发现,惩罚系数相同,重新训练时,会有不同的效果展示,才疏学浅,尚未能解释,如果有知道为什么的大神,敬请指点迷津。
2、SVC参数调整
SVC模型中有两个非常重要的参数,即C与gamma,其中C是惩罚系数,这里的惩罚系数同LinearSVC中的惩罚系数意义相同,表示对误差的宽容度,C越高,说明越是不能容忍误差的出现,C越小,表示越容易欠拟合,泛化能力变差。而另一个非常重要的参数gamma,是在选择RBF函数作为核函数后,才出现的,这个也是LinearSVC中所不包含的。高斯函数RBF中的gamma值有一个自带的默认值:gamma='auto_deprecated';表示的是数据映射到新的特征空间后的分布,gamma值越大,支持向量就越少,gamma值越小,支持向量就越多。支持向量的个数影响训练与预测的速度。
RBF公式里面的sigma和gamma的关系为:
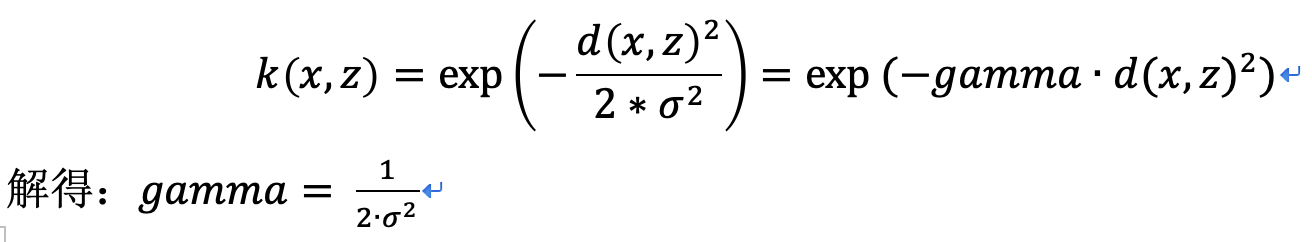
gamma的物理意义是关于RBF的幅宽,它可以影响每个支持向量对应的高斯的作用范围,进而影响分类器的泛化能力。在本例中我更改了多组c的值、gamma的值效果均是差之千里:
C=2、gamma=’auto’
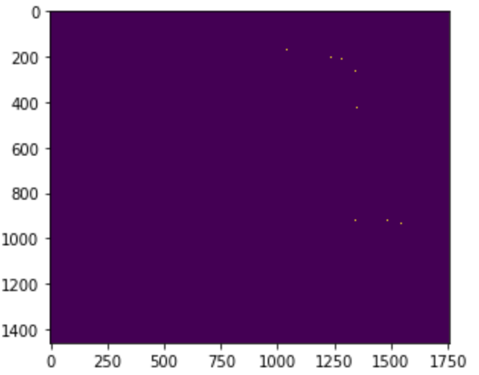
C=default、gamma=1
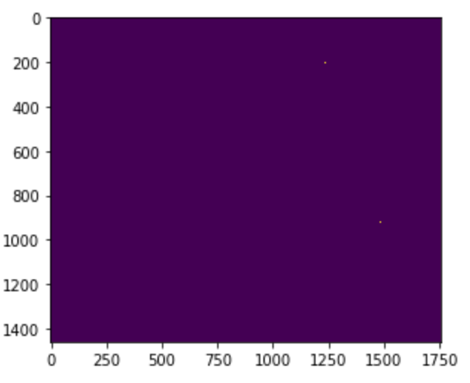
C=default、gamma=10
C=default、gamma=100
图4 SVC参数修改后分类效果
如此效果真的是让人头疼,不过有一点可以确定的是在样本海量的情况下,LinearSVC表现的要更好一些,算法执行时间很快,模型也较小,同样的样本下SVC用到的时间大概是LinearSVC的60倍左右。但是我们不能就此而已,我们要为问题寻找思路,这便转换成了一个寻优问题。
对于同时受到两个参数影响的调参问题,我们可以使用一些寻优算法,常用的寻优算法可分为两类:一类是在参数定义域空间内进行网格式搜索,例如专门针对SVC参数的GridSearch算法、交叉验证算法,这类算法虽然稳定性高、参数估计准确,但是算法时间复杂度较高,计算量较大;另一类是采用启发式优化,例如gaSVMForClass遗传算法参数寻优、psoSVMForClass粒子群优化算法参数寻优等,这类算法能够更快的寻找到最优参数组合。
本文先从GridSearch入手,毕竟它是sklearn模块中的子模块,导入方法也较简单:直接from sklearn.model_selection import GridSearchCV就ok。
GridSearch网格搜索是一种调参手段,它的基本原理是暴力求解,即尝试每一种可能性,将表现最好的认为是结果。使用的方法是穷举搜索,在所有的候选参数中,通过循环遍历,尝试每一种可能性。对于要调整两个参数c和g的高斯核函数来说,加入c的可能值有5种,g的可能值有6种,那么要遍历所有的c与g的组合参数值,就有5*6种可能,也就是可以列一个表格包含5列6行的表格,然后依次对表格内容进行遍历便可以实现遍历所有的30种可能了。
例如:遍历gamma在[0.001, 0.01, 0.1, 10, 100, 1000]范围,C在[0.001, 0.01, 0.1, 10, 100, 1000]范围内的最优值,便可以执行以下程序:
best_score = 0 for gamma in [0.001, 0.01, 0.1, 10, 100, 1000]: for C in [0.001, 0.01, 0.1, 10, 100, 1000]: svm = SVC(gamma=gamma, C=C) svm.fit(X_train, y_train) score = svm.score(X_test, y_test) if score > best_score: best_score = score best_parameters = {'gamma':gamma, 'C':C} print("best score:{:.2f}".format(best_score)) print("best parameters:{}".format(best_parameters))
最终结果便是:
best score:0.95
best parameters:{'gamma': 0.01, 'C': 10}
由此可以看出,其实这个GridSearch方法就是一个代替我们去进行尝试的machine,它无法自己寻求合理的c与gamma的取值范围,且比较耗时,你设置的C值和gamma值越多,寻优时间就会翻倍增加。另外我将这对最优组合放在实际预测中查看效果,效果也是不甚理想啊:
图5 GridSearch寻到的最优C&gamma组合预测的效果
效果不理想的原因与我设置的C值与gamma值不全肯定有很大的关系,所以再利用我们这种方法就不怎么可行了,于是我们继续更改:
第一种笨方法:
还是利用GridSearch,只不过在C、gamma赋值处将固定值替换为初值加步长的形式,用到了range()函数。
第二种方法是利用我前文所讲到的离子群优化算法寻优:
离子群优化算法(Particle Swarm Optimization Algorithm, PSO)是一种基于群体协作的随机搜索算法,其原理是源于对于鸟类捕食行为的研究,通过群体中的个体之间的协作核信息共享来寻找最优解,PSO算法的优势在于简单的算法实现和简洁的参数设置,目前较广泛的应用于函数优化、图像处理等方面,但是其缺点也是不可忽视的:提前收敛、容易陷入局部最优、维数灾难等缺点。
PSO算法的基本流程为:
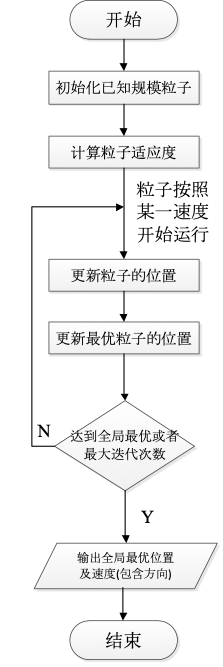
Step1:初始化一群粒子(假设群体规模为已知),初始化随机位置及速度;
Step2:评价每个粒子的适应度(计算其与最优位置的距离);
Step3:对每个微粒,将其适应值与其经过的最好位置pbest作比较,如果较好的话,则更新较好位置为最好位置;
Step4:加入权重因子,继续迭代更改粒子的速度及位置;
Step5:重复step2和step3直至达到结束条件(全局最优/迭代次数上限),否则继续执行2,3。
(未完待续,详情见下篇)