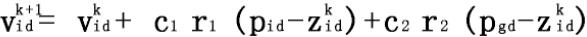概述:
正如其他的那些进化算法一样,粒子群算法的灵感同样来自于大自然。它由Eberhart和Kennedy共同提出的,其基本思想来自于他们早期对许多鸟类的群体行为进行建模仿真研究结果的启发。
假设在一块广袤无垠的栖息地上有一群自由自在的鸟儿和一堆丰盛的食物,可是鸟儿却不知道食物在哪,现在我们的目标是让所有的鸟儿找到这堆美食,那该怎么办呢?Kennedy认为鸟之间存在着相互交换信息,于是他们在仿真中添加了一些内容:每个个体(鸟儿)能够通过一定规则估计自身位子的适应值,并能记住自己当前所找到的最好位置,记作:“局部最优pBest”。此外还应记住整个群里中所有鸟儿找到的最优位置,记作:“全局最优gBest”。这两个最优变量使得鸟在某种程度上朝这些方向靠近。
基本数学概念:
设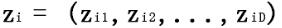
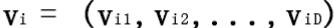
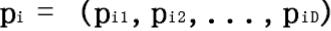
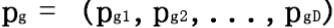
在每次迭代中,粒子根据以下公式进行更新速度和位置:
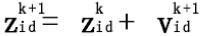
其中i = 1,2,3…m;d = 1,2,3…D;k是迭代次数;r1和r2为[ 0 ,1]之间的随机数,为了保持群体的多样性;c1和c2为学习因子,也称加速因子,其使粒子具有自我总结和向群体中优秀个体学习的能力,从而向自己的历史最优点以及群体内最优点靠近,这两个参数对粒子群算法的收敛起的作用不是很大,但适当调整这两个参数可以减少局部最优的困扰。
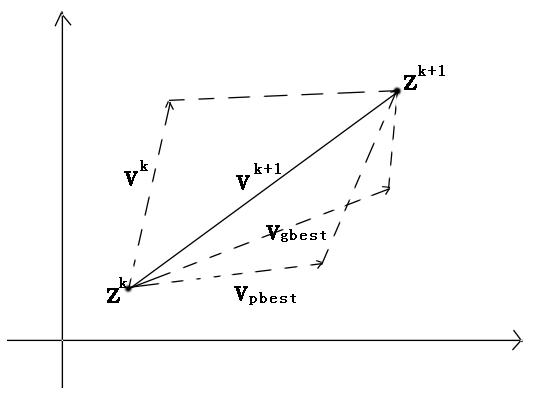
粒子群算法的速度公式右边包含了3个部分:第一部分是粒子之前的速度矢量,第二部分是粒子向自身最优位置靠近的速度矢量,第三部分为粒子向群体最优位置靠近的速度矢量。如果没了后面两部分,粒子将会保持相同的速度矢量朝一个方向飞行,这样粒子有极大的可能找不到最优解,如果没了第一部分时粒子的飞行速度将仅仅由它们当前位置和历史最优位子决定,则速度自身是无记忆的,且整个群体没有向空间拓展的功能,除非全局最优解在初始群体的空间内,否则无法搜索到全局最优。
算法流程:
1)开始
2)设定最大迭代次数(这里迭代次数人为设定,也可以自行根据条件来跳出循环)
3)设定群体个数(大群体搜索快计算慢,小群体搜索慢计算快)
4)初始化每个粒子的初始位子z
5)初始化每个粒子的初始速度v
6)计算每个粒子的初始位置的适应值fun(z),通过比较粒子间的适应值来选择初始群体最优位置gbest
7)将每个粒子各自的初始位置赋值给它们的pbest
8)k=0
9)k=k+1
10)根据速度更新公式更新每个粒子的新速度v
11)根据位置更新公式更新每个粒子的新位置z
12)每个粒子通过对比新旧位置更新pbest
13)更新粒子群体的最优位置gbest
14)从(9)开始迭代,直到达到迭代次数后跳出循环
算法效果展示:
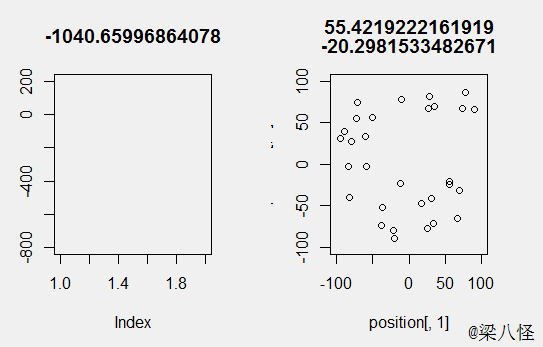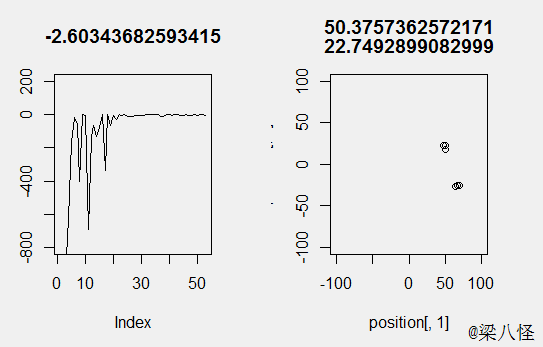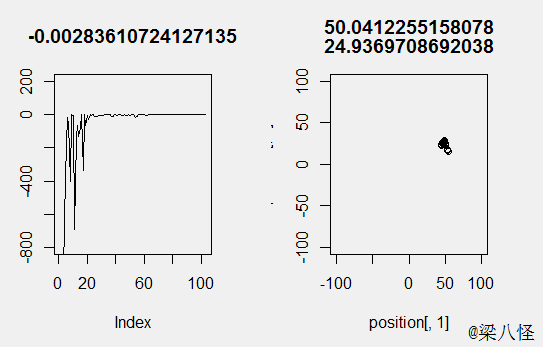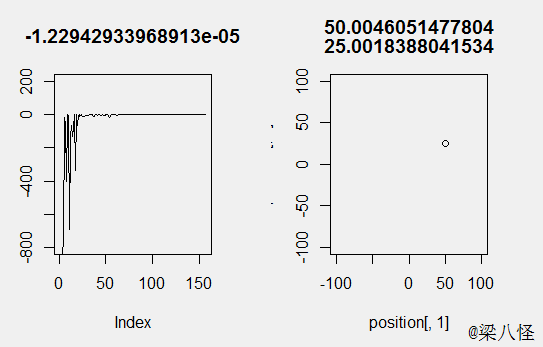
输入函数f(x) = -1/2*((x1-50)(x1-50)+(x2-25)(x2-25)) , 我们很容易知道其最值为0,分别为x1=50和x2=25的时候:
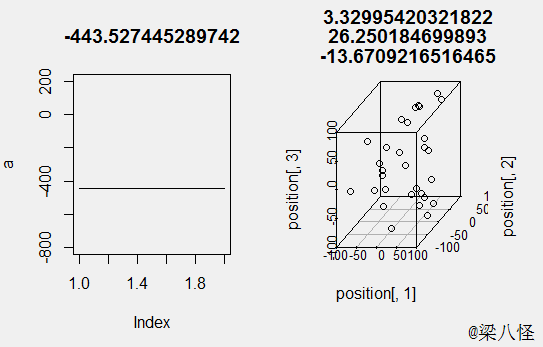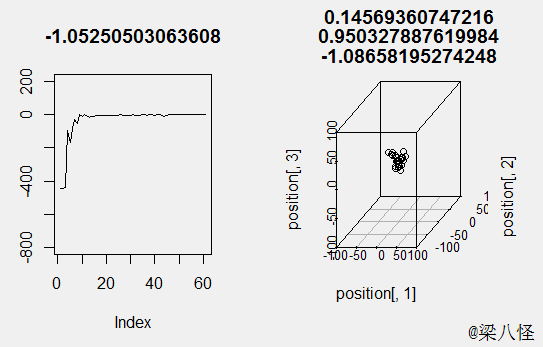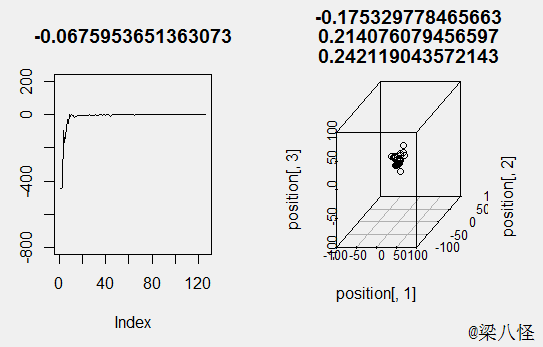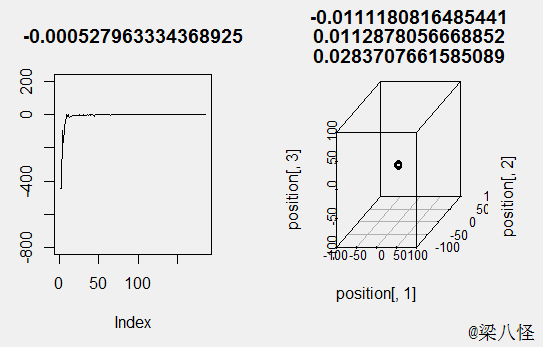
输入函数f(x) = -1/2*(x1*x1+x2*x2+x3*x3) , 我们很容易知道其最值为0,分别为x1=0,x2=0,x3=0的时候:
R代码
PSO<-function(circle = 1000,biomass = 1000,c1,c2,position_range,vector_range,FUN,purpose = 'max'){
dimension<-nrow(position_range);#计算维度
position <- matrix(nrow = biomass,ncol = dimension)#初始化位置
for (i in 1:biomass) {
position[i,]<-apply(position_range, 1, FUN = function(x){
return( runif(n = 1,min = x[1],max= x[2]))
})
}
vector <- matrix(nrow = biomass,ncol = dimension)##初始化速度
for (i in 1:biomass) {
vector[i,]<-apply(vector_range, 1, FUN = function(x){
return( runif(n = 1,min = x[1],max= x[2]))
})
}
pbest<-position;#初始化每个粒子最优点
fun<-match.fun(FUN)
adopter<-apply(position, 1, FUN = fun)#计算全局最优点
gbest<-matrix(data = rep(x = position[which(adopter == match.fun(purpose)(adopter,na.rm = T))[1],],biomass),nrow = biomass,byrow = T)
for (i in 1:circle) {
vector<-vector+c1*runif(n = 1,min = 0,max = 1)*(pbest-position)+c2*runif(n = 1,min = 0,max = 1)*(gbest-position)#更新速度
temp<-position+vector#重新计算位置
for (i in 1:dimension) {#控制粒子的位置和速度在域内
temp[which(temp[,i]<position_range[i,1]),]<-position_range[i,1]
temp[which(temp[,i]>position_range[i,2]),]<-position_range[i,2]
vector[which(vector[,i]<vector_range[i,1]),]<-vector_range[i,1]
vector[which(vector[,i]>vector_range[i,2]),]<-vector_range[i,2]
}
if (purpose == 'max')#更新每个粒子的最优点
c<-which(apply(temp, 1, FUN =fun)>apply(position, 1, FUN =fun))
else if (purpose == 'min')
c<-which(apply(temp, 1, FUN =fun)<apply(position, 1, FUN =fun))
pbest[c,]<-temp[c,]
position<-temp#更新位置
adopter<-apply(position, 1, FUN = fun)#更新全局最优点
gbest<-matrix(data = rep(x = position[which(adopter == match.fun(purpose)(adopter,na.rm = T))[1],],biomass),nrow = biomass,byrow = T)
}
return(list(best = gbest[1,],value = fun(gbest[1,])))
}