KEGG数据库在线使用
KEGG简介
KEGG是一个整合了基因组、化学和系统功能信息的数据库。把从已经完整测序的基因组中得到的基因目录与更高级别的细胞、物种和生态系统水平的系统功能关联起来是KEGG数据库的特色之一。与其他数据库相比,KEGG 的一个显著特点就是具有强大的图形功能,它利用图形而不是繁缛的文字来介绍众多的代谢途径以及各途径之间的关系,这样可以使研究者能够对其关注的代谢途径有直观全面的了解。
ko:表示通路,这个通路是不分物种的,相当于所有物种某一通路的并集。
KO(KEGG Orthology):是KEGG中一个“专有名词”,表征一个基因。KO作为ko通路中的基本单位,它是蛋白质(酶)的一个分类体系。通常序列高度相似且在同一通路中具有相似功能的蛋白质被归为一组,即一个KO。
打开KEGG数据库
输入网址 https://www.kegg.jp/kegg/kegg2.html:

KEGG - Table of Contents功能简介:
- 以分类列表的形式介绍了KEGG所包含各个内容模块,其中蓝色的字体可以直接点击进入以获取更为详细的各模块相关内容介绍;
- 拥有全局性检索框,在检索框内输入关键词,即可查询KEGG中与关键词相关的Pathway(通路)、Module (模块)、Orthology(直系同源)和Genome(基因组)、Genes(基因)和Enzyme(酶)等诸多信息;如有特殊需求,也可以进行个性化筛选,比如仅搜索与关键词相关的Module信息。
KEGG相关数据库关系如何?
打开另一网址 https://www.genome.jp/linkdb/,将看见这个界面:

LinkDB: database of link information功能简介:
LinkDB解析了KEGG数据库内部数据的流通以及和其它数据库的联系。若是需要了解具体某个数据库(如 :Pathway)的来源,直接点击上图下方的Pathway即可高亮显示该数据库数据来源相关的各类数据库名称。此外,还可以直接下载数据库与数据库间的编号对应关系!
如何利用KEGG数据库完成数据挖掘?
问题:做类似下面文献中的两张图?


思考:从上图来看的话,首要任务就是收集氮代谢(Nitrogen metabolism)相关的数据信息啊,那怎么搜呢?
步骤如下(查找方式多种,此处以上面介绍的“KEGG最优打开方式”为例进行演示):










- 最优方式打开KEGG数据库,Search栏直接输入“Nitrogen”再回车,将会检索到KEGG中与氮代谢相关的各类信息,其中KEGG PATHWAY下的map00910就是我们要查找的关键信息:
- 页面跳转到https://www.kegg.jp/dbget-bin/www_bfind_sub?mode=bfind&max_hit=1000&dbkey=kegg&keywords=Nitrogen,如下所示:
- 点击上一步中的“map00910”,将出现如下图所展示的氮代谢相关内容的精细描述,有7个相关Module,2个疾病相关内容,还有我们需要get的氮相关基因KEGG ORTHOLOGY(KO)等信息。
- 点击上一步中的KO pathway编号“ko00910”,将出现一个与上一步相似的页面,但是其中已经包含了60个参与氮代谢的KO号,如下图所示:
- 最后,从自己的KEGG注释结果中挑出相关的KO或Gene和Module等信息作为输入数据,就可以去分析作图了(比如上面的Heatmap、重构代谢通路图)。
其他KEGG数据库在线工具如何使用?
打开KEGG数据库时,可以发现KEGG数据库还提供多种其它在线分析工具:

先给大家介绍上图中2种比较炫酷的工具,其它工具可根据个人需求自行学习!
KEGG Mapper - Search & Color Pathway工具
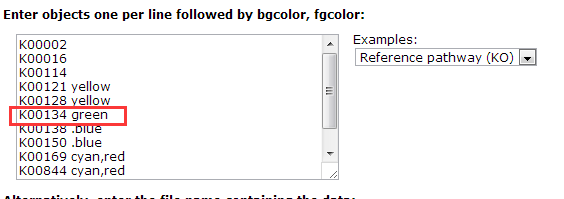
利用Search & Color Pathway在线工具可DIY通路图中的基因(KO)或其它信息(如文字)的背景填充色,具体操作界面、输入参数设置和步骤可参考下图:

修改:上图中的Examples所处的状态应为:

这里,输入的KO编号如下:
K00002
K00016
K00114
K00121 yellow
K00128 yellow
K00134 green
K00138 .blue
K00150 .blue
K00169 cyan,red
K00844 cyan,red
点击“Exec”之后,结果如下:

KEGG会根据输入的KO编号从Reference Pathway库中找出与之相关的代谢通路,并将其列出。这里,我们点击“ko00010”,将得到如下代谢通路图:

可以看出,我们设置的颜色在代谢通路图中已经显现出来了

BlastKOALA工具
一种在线KEGG注释方法,具体操作界面、输入参数设置和步骤可参考下图:

基因注释
1.提交序列文件,为Fasta格式的蛋白质序列文件(The maximum number of sequences allowed ranges from 5000 to 10000 depending on the KEGG GENES dataset selected);
2.Enter taxonomy group of your genome(选择物种类型:区分原核与真核);
3.Enter KEGG GENES database file to be search(默认即可)
4.Enter your email address(填写你的邮件地址,提交任务后,需要从邮箱确认,结果也将会发送到邮箱);
5.提交任务,并从个人邮箱确认(数据只会保留一个星期)
输入个人邮箱地址(确认任务提交是否成功),点击“Request for email confirmation”按钮后,呈现的结果如下(网上找的图):

okay,KEGG的内容实在是太多了,短时间是没法讲完的,所以,有机会以后再继续分享吧!