转载自https://mp.weixin.qq.com/s/pqbMXMkuqEXbLf31PTxGZQ
KEGG简介
KEGG 数据库于 1995 年由 Kanehisa Laboratories 推出 0.1 版,目前发展为一个综合性数据库,其中最核心的为 KEGG PATHWAY 和 KEGG ORTHOLOGY 数据库。在 KEGG ORTHOLOGY 数据库中,将行使相同功能的基因聚在一起,称为 Ortholog Groups (KO entries),每个 KO 包含多个基因信息,并在一至多个 pathway 中发挥作用。而在 KEGG PATHWAY 数据库中,将生物代谢通路划分为 6 类,分别为:细胞过程(Cellular Processes)、环境信息处理(Environmental Information Processing)、遗传信息处理(Genetic Information Processing)、人类疾病(Human Diseases)、新陈代谢(Metabolism)、生物体系统(Organismal Systems),其中每类又被系统分类为二、三、四层。第二层目前包括有 43 种子 pathway;第三层即为其代谢通路图;第四层为每个代谢通路图的具体注释信息。
KEGG简单探索
首先打开KEGG数据库,你会进入下面这个界面,我们直奔主题,点击KEGG PATHWAY。 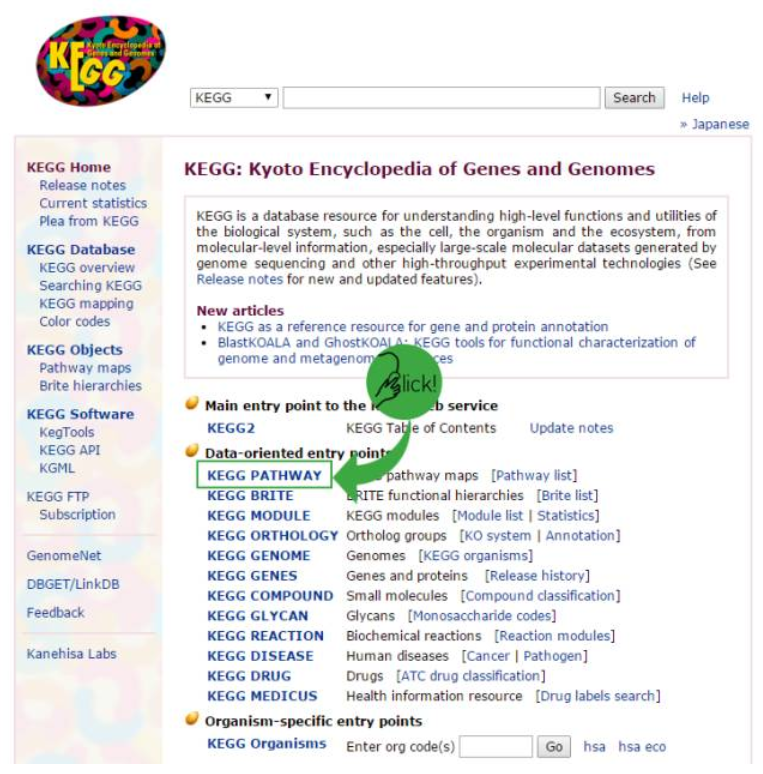
点击进去之后,你看到的界面是下面这样的。
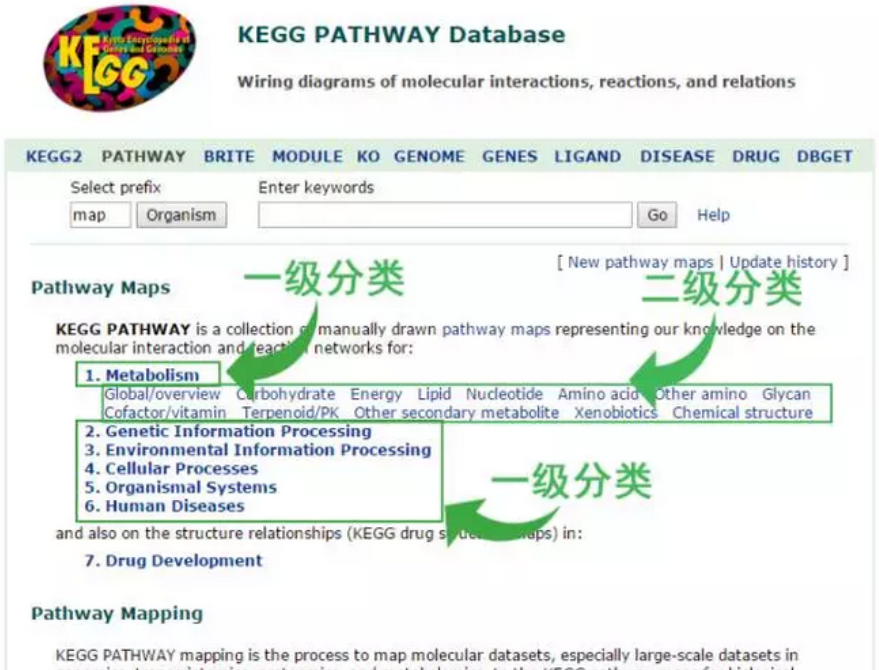
点击cellular process,页面迅速的跳到这部分的二级pathway了,这时你可以看到二级通路下面,更细化的通路。
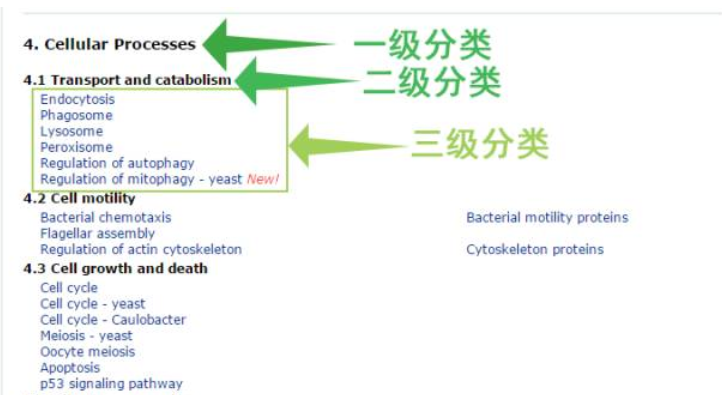
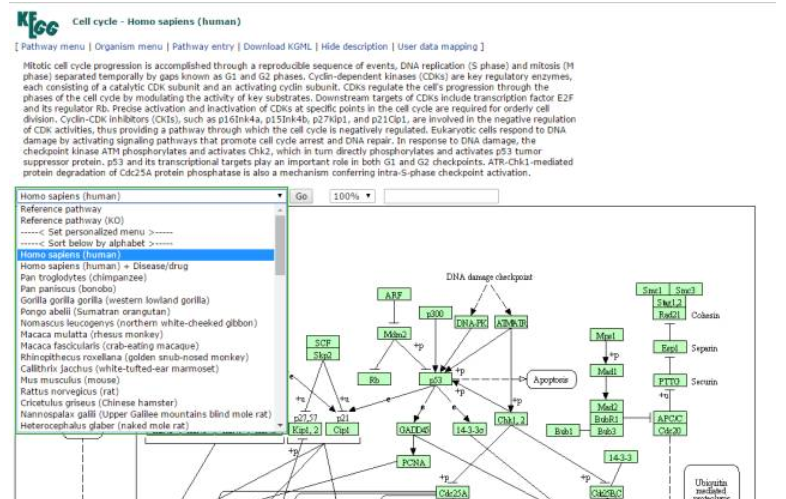
再点击三级分类的通路,就可以找到我们想要的pathway map了。比如点击三级分类pathway map 的“cell cycle”,然后就进入了下面这样的界面。
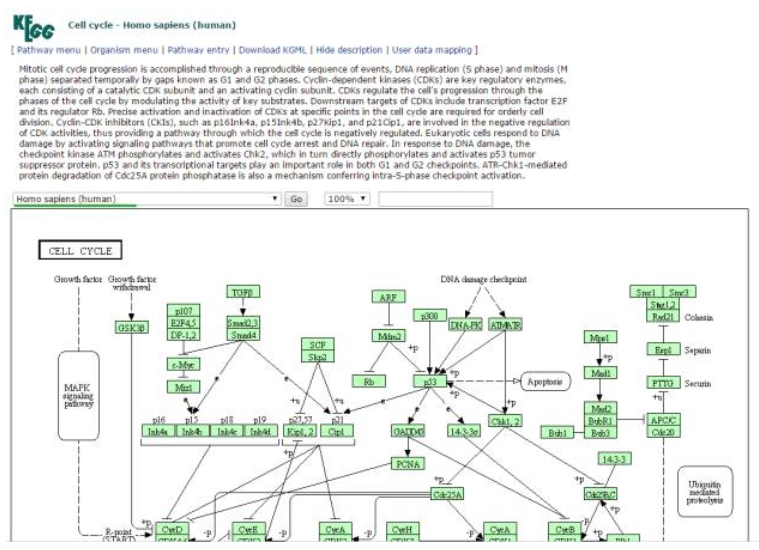
注意哈,一般默认是人的pathway,如果你像查看其他物种上的pathway,你需要点击下拉三角形,选择你所关心的物种。如下面图中所示哈。
KEEGG代谢通路图解读
KEGG pathway中有着大量的通路图,以PI3K-Akt signaling pathway(ko04151)为例,里面包含了大量的蛋白等化合物,以及它们之间相互作用的关系。 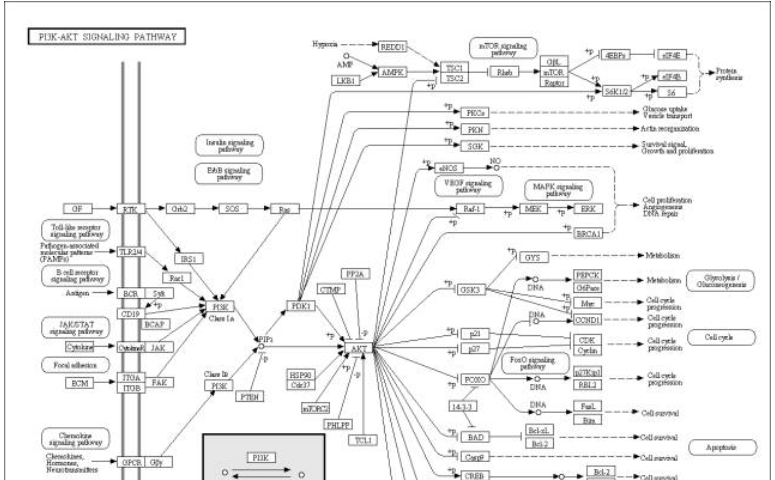


正确认识代谢通路富集分析
我一直建议不要仅仅基于Pathway富集分析的结果解读数据,人为的解读和挑选是必不可少的。因为生物数据的解读,在现阶段更多是生物学问题,而不是数学问题。原因大体如下:
(1)基因调控是个系统,不要仅仅看成1个孤立的pathway。
在今年参加的第二届植物抗逆会议上,1个教授说了一句话,我认为很有道理。“在植物体内其实根本就不存在pathway,什么脱落酸通路,水杨酸通路,其实这些调控因子都是相互联通,相互影响的,是个整体。只是我们人类为了研究方便,人为将这些系统拆分各个子集。 ” 所以,如果你真的将pathway看成1个个破碎的途径,以为某种处理只会影响某个pathway,富集分析必须在数学上或统计学上得到1个指向性很强的结论,那是不大可能的。
具体说了,说基因调控是个系统,可以从两个层面进行解读:
-
a)1个基因的改变可以造成整个系统的改变;
举几个例子:把1个生命活动必须的蛋白敲除后,整个细胞会发生紊乱。而植物抗病应激,也往往是1个受体蛋白识别了病原的外源蛋白,然后导致整个细胞系统的变化。 -
b)1个基因往往有多个功能,但执行具体的功能往往是不同蛋白复合物共同作用的结果。
例如,基因X理论上在不同情况下,有可能参与A、B、C通路。在某个生物处理下,或许基因X 只在A通路里起作用。但如果进行基因注释的话,X同样也会被注释到B、C。所以,富集分析的结果总是会涉及特别多的通路。例如,研究人的项目,无论什么研究背景,常常会富集到帕金森综合症通路。不是你的材料真的得了帕金森综合症,只是那些与你实验处理相关的基因,在一定条件下也可以参与到帕金森综合症的过程,所以被注释到了这个通路里。
小结:所以,我们也看到了。无论什么实验处理,总有可能导致整个系统的变化。同时,基因的通路注释也有欺骗性。那么,从这一堆冗余信息中,想得到与我们研究相关的结论,离不开人为的筛选也解读。从那个复杂的整体中,筛选出核心的局部片段,这是个技术活。“这样的话是否存在一个问题就是在结果的解释上比较主观,也会因自身背景知识的不足而漏掉一些新颖的结果”。那当然,同样的结果1个外行可能什么都没有看见。但1个资深的学者可能会把握到很精彩的内容。好像任何领域都是如此,除了提高内功好像没有其他捷径。
(2)pathway富集分析的统计假设,并非在任何情况下都适用
pathway富集分析,在生物学上的假设是:1个pathway上游基因的改变,会导致下游相关基因改变,从而改变通路中大量基因的表达,达到统计学上富集的效果。但很多pathway中,基因A、B、C并不是相互调控的关系,而是共同参与某个过程的不同部分。
例如,代谢物X的合成修饰。基因A、B、C分步骤参与合成的3个步骤。基因A给X前体加了羟基,然后传递到下游;基因B又给X前体加了苯环,再传递到下游;基因C又给X的前体加了个乙酰基,完成X的合成。那么,基因A、B、C是参与了的相同的通路。如果基因A发生表达量变化,会直接调控影响B、C的表达量变化吗? 看来很有可能不会,所以从RNA-seq差异分析的富集分析结果中,这个通路是不显著的。那么基因A的表达变化是否有生物学意义? 当然有,因为代谢物X的合成的确受影响了。
类似的例子,理论上DNA差异甲基化的结果,就不能看pathway富集分析的结果。1个pathway 1个基因的DNA甲基化变化,就足以改变这个通路的基因表达,而不需要整个通路的甲基化都发生变化。DNA甲基化、组蛋白CHIP-seq的结果,其实只看功能注释、或通路注释就足够了,不需要考虑富集。
所以,我们还是要观察、理解某个核心pathway中基因的相互作用,才能判断其中的基因变化是否有生物学意义, 而不仅仅看富集分析的p value或Q value。
(3)目前的pathway是不完整的。
目前KEGG等数据库,收录的是已有的研究结果。但这些pathway的信息,远没有到达完善的水准。大部分通路只是了解1个大概的调控途径,而中间有什么转录因子参与、是否还有其他代谢物的生成,都是不知道的。这些通路的完整性,也会影响pathway富集分析结果。例如,基因A发生变化了,看起来下游基因没有变化。也许是还有其他的调控在起作用,只是这些调控作用现在还不知道而已。
总结:pathway 和 GO富集分析结果的解读,应该从生物学意义的角度出发,P value 和 Q value只是个参考而已,那些不显著的通路也值得解读(从功能注释的角度解读,而不是从富集分析的角度解读)。只要结果可以解释,有意义,不用太迷信P value。
代谢通路可视化
以下三个图是主要使用 pathview, KEGGREST和KEGGgraph这三个R包绘制的。我是根据例子教程绘制的,具体原理我还没有弄明白。

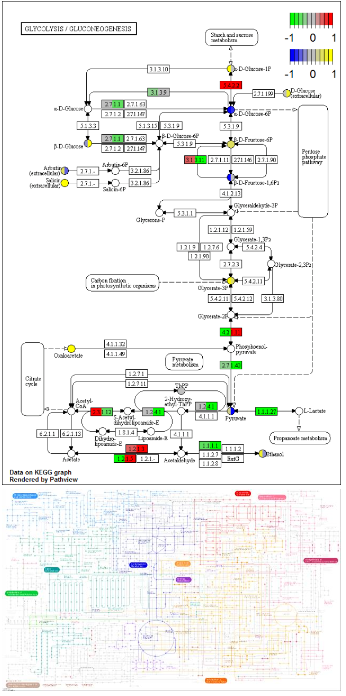
原图在KEGG的链接是http://www.genome.jp/kegg-bin/show_pathway?map01100
还有其他一些软件:
clusterProfiler:https://www.bioconductor.org/packages/devel/bioc/vignettes/clusterProfiler/inst/doc/clusterProfiler.html
Reactome: http://www.reactome.org/
ReactomePA:https://www.bioconductor.org/packages/release/bioc/vignettes/ReactomePA/inst/doc/ReactomePA.html
参考资料
http://www.omicshare.com/forum/forum.php?mod=viewthread&tid=235&highlight=KEGG
http://www.omicshare.com/forum/forum.php?mod=viewthread&tid=330&highlight=KEGG
http://www.omicshare.com/forum/forum.php?mod=viewthread&tid=270&highlight=KEGG
http://www.omicshare.com/forum/thread-832-1-12.html(GO、PATHWAY富集分析中是否一定需要选择显著富集的通路)
https://github.com/dgrapov/TeachingDemos/blob/master/Demos/Pathway%20Analysis/KEGG%20Pathway%20Enrichment.md