GEO生信数据挖掘(十)肺结核数据-差异分析-WGCNA分析(900行代码整理注释更新版本)
通过 前面十篇文章的学习,我们应该已经可以获取到一个”心仪的基因列表“了,相较于原始基因数量,这个列表的数量已经有了明显的缩小,为了进一步确定Hub gene 需要借助两个工具。
使用STRING在线数据库进行PPI分析。
使用Cytoscape本地客户端进行蛋白互作关系图绘制。
STRING在线数据库进行PPI分析
STRING在线数据库(STRING: functional protein association networks:https://cn.string-db.org/)
准备好自己感兴趣的gene list 整个这列复制一下,STRING也支持导入文件。

STRING 操作第一步界面
看图有操作说明


也可以选择自动(auto-select),数量多的话,点击SEARCH后,第二步也会提示,选择物种。
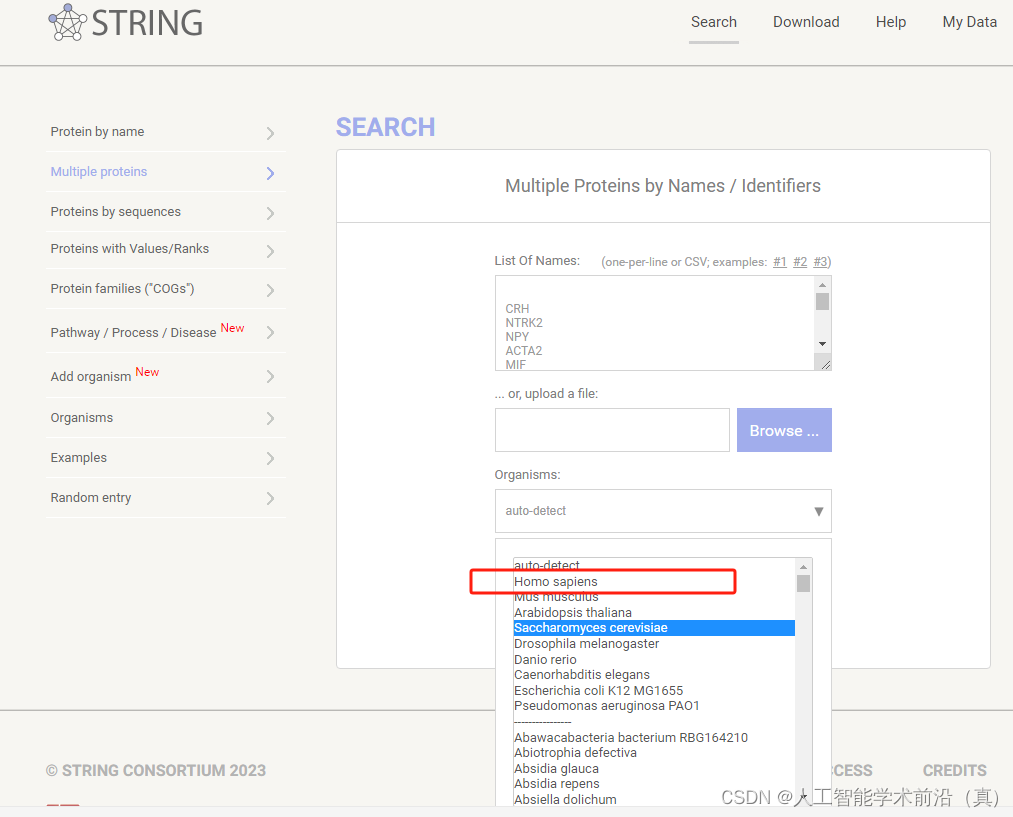
第二步的界面
可以看看基因,没问题,点击CONTINUE

第三步界面 出现结果

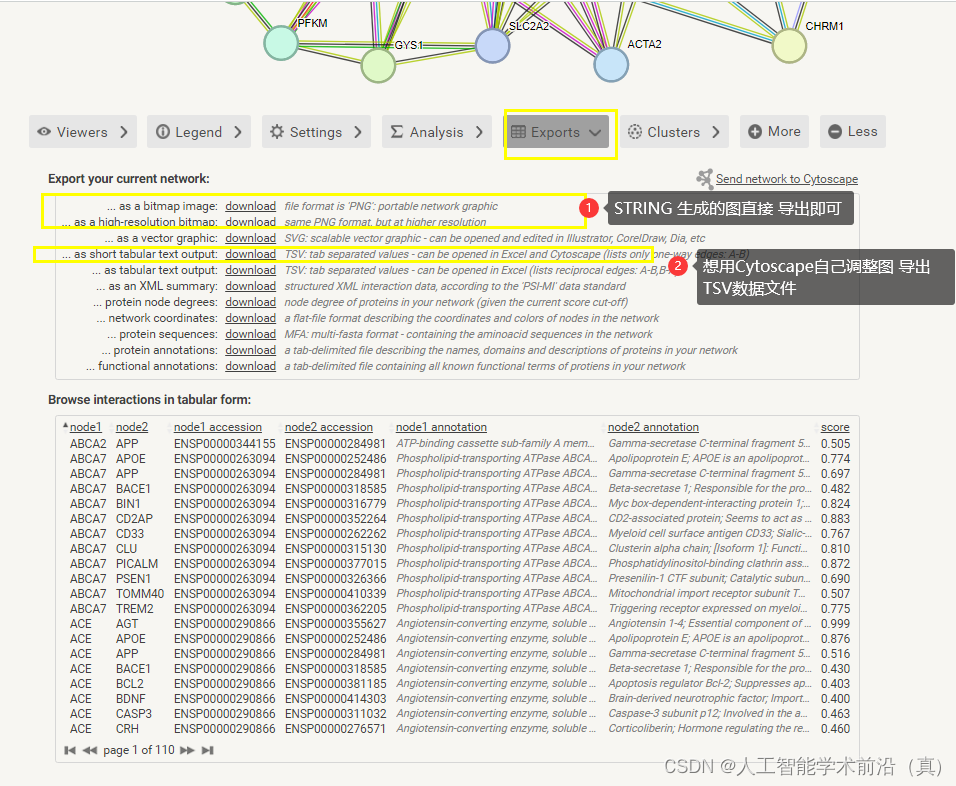
一般会用到 Settings 和Exports
Setting设置看图,调整完毕后,点击UPDATE更新结果。

结果导出
用Cytoscape自己调整图 导出TSV数据文件
使用Cytoscape本地客户端进行蛋白互作关系图绘制
TSV文件已经从STRING数据库中下载保存完毕。

安装Cytoscape 需要配置java环境
不同版本对应的java版本有要去(本文使用Cytoscape3.10,jdk-17_windows-x64_bin,总计369M的压缩包,链接: https://pan.baidu.com/s/19gjKs9w6TM2ylXxupA5ykw?pwd=4p7y 提取码: 4p7y 复制这段内容后打开百度网盘手机App,操作更方便哦)
如果觉得百度盘慢,评论区留下邮箱,私发。
打开Cytoscape的界面
导入文件

设置列

初步图形生成
先删除孤立节点(可以配合Ctrl 键进行操作)

首先进行数据网络分析
选中数据,Tool,analyze Network,
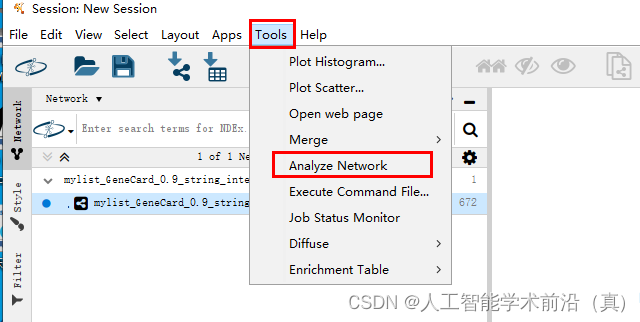
可以再次观察,下方的数据区域(Node Table),增加很多信息,我们最关心的是degree列,degree数据列值越大,那么说明,对应基因越重要,有更多的基因连接到了目标基因。
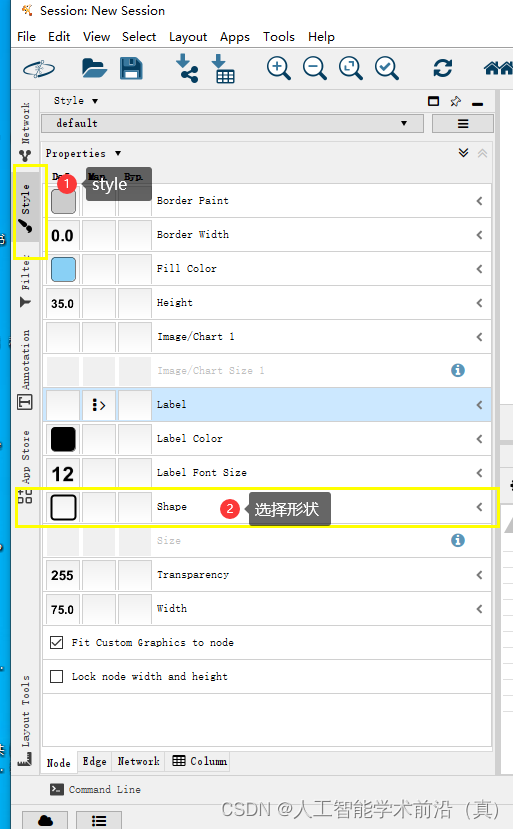
基因样式设置
style -> shape -> 圆形
设置圆形的样式,并勾选,锁定节点的长宽

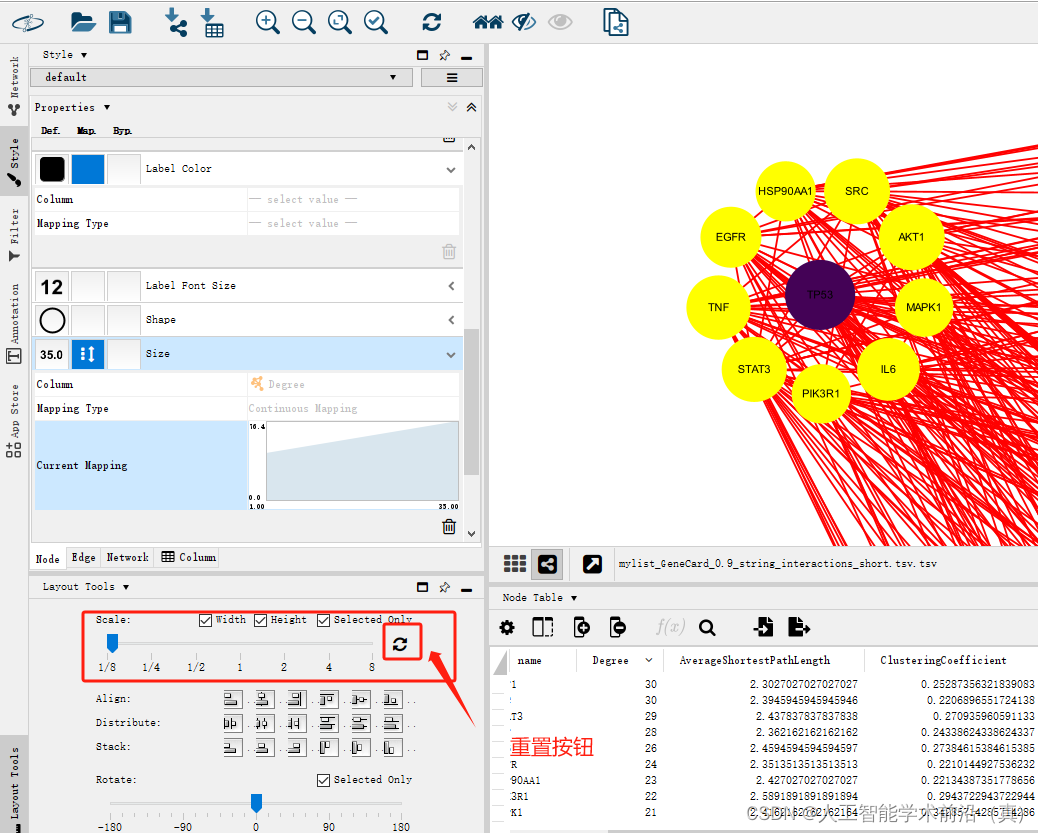
节点的大小设置
让节点的大小和degree 关联起来。

观察TP53基因,大小明显改变,且是最大的。
节点的形状太小,把size 的下限提高。

节点的颜色设置
让节点的颜色和degree 关联起来。
设置Fill Color
degre越大则颜色越深,反之,则越浅。(设置方法,同理)

基因选择
可以鼠标选择图中的节点,配合快捷键
还可以在Node Table 列表中进行选择基因名称,然后右击鼠标,功能菜单中选择图中的节点。

基因节点的排列样式
目前,节点已经设置完毕,边可以根据需要继续设置,方法类似。
现在图形的样式排布比较随意,可以将节点按照环形,或者方形进行排列。
Layout ->Grid layout
可以选择部分节点,也可以直接对所有节点进行操作。
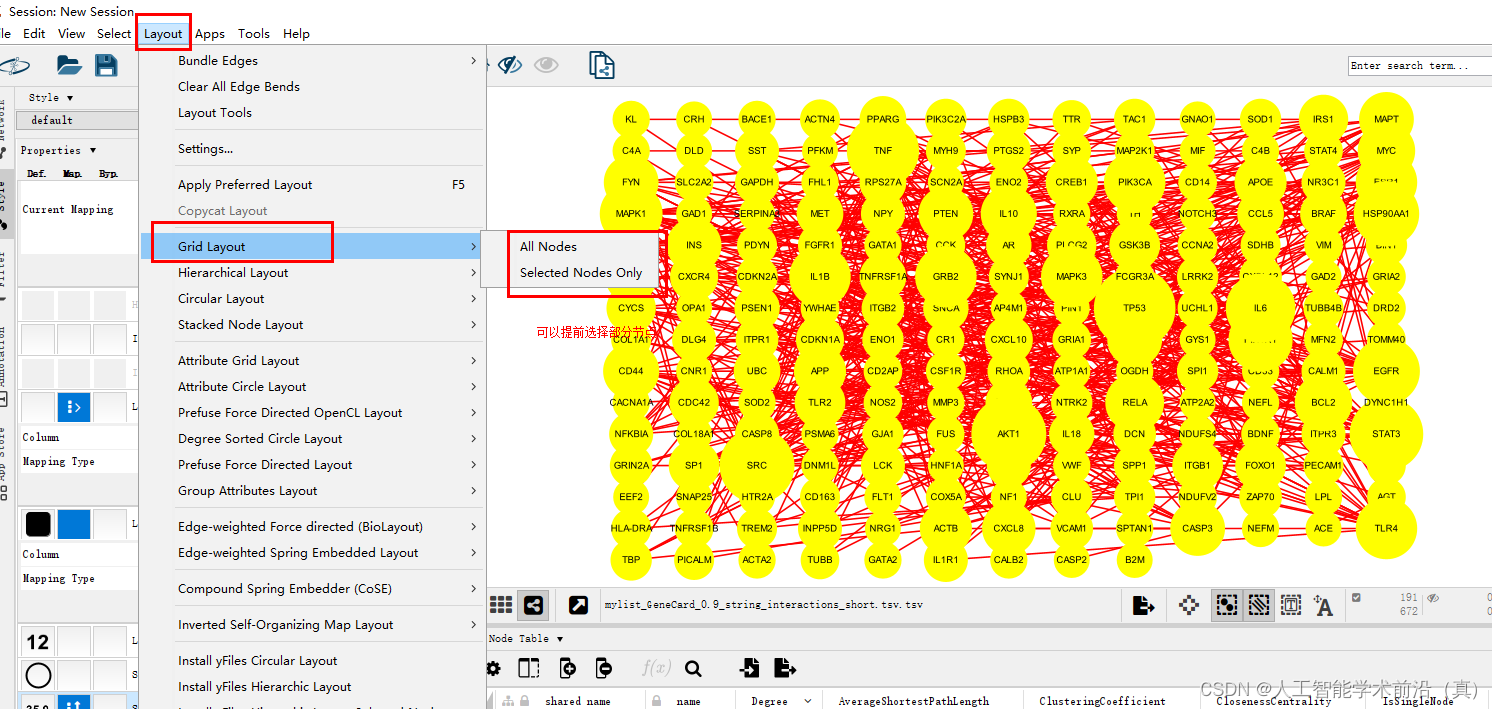
设置环形结构
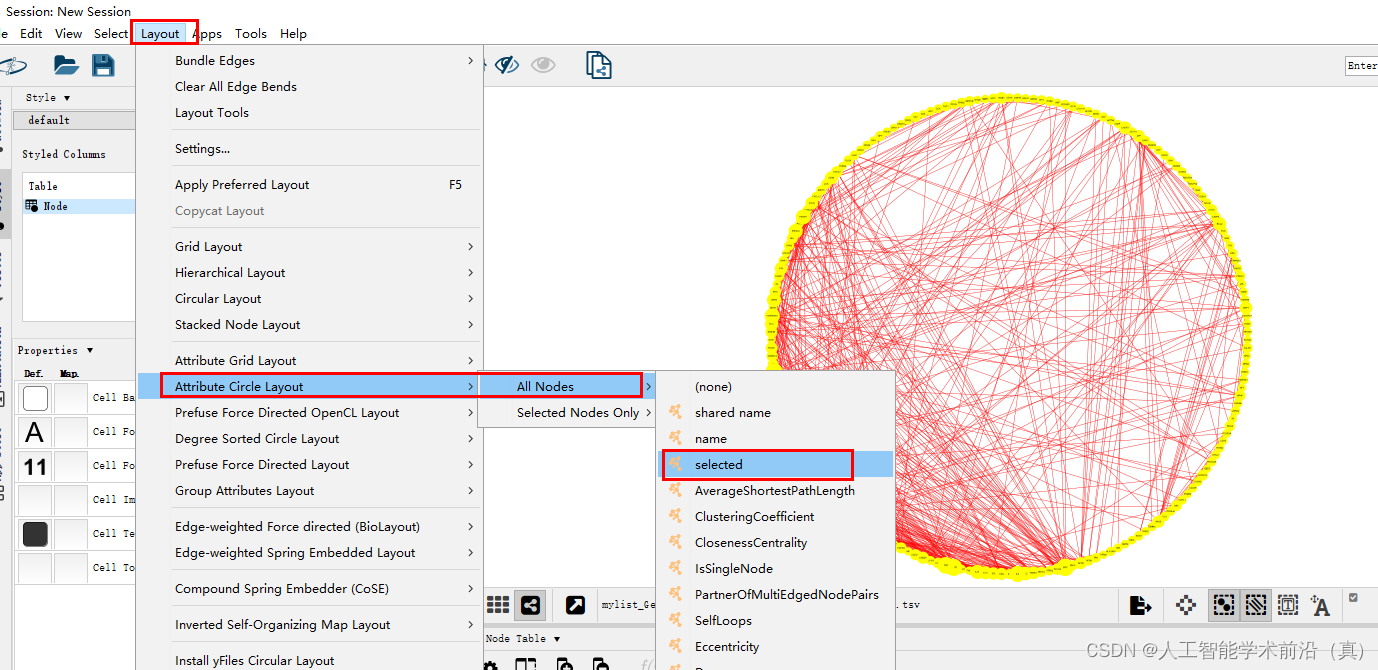
复杂的环形结构排列
上面形状有了,但是由于本例中节点的数量的太多,还可以继续美化。


选择degree前10个基因先绘制一个圆形,再绘制其他基因。可以迭代多个圆形。
选择了三组degree 分段后的基因,生成了三个圆形,下面进行排列

使用 Layout Tool 进行图形排列
打开layout Tool
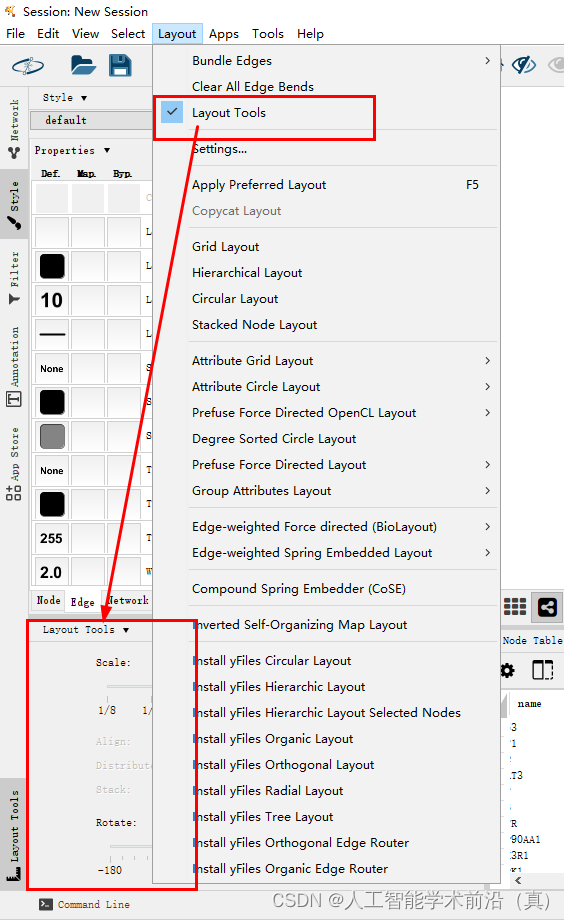
调整scale 放大缩小,可以调整圆环的距离。

如果缩小至最测了,仍然需要缩小,可以点击重置比例。比例又会回到1。
最终调整完毕

导出文件
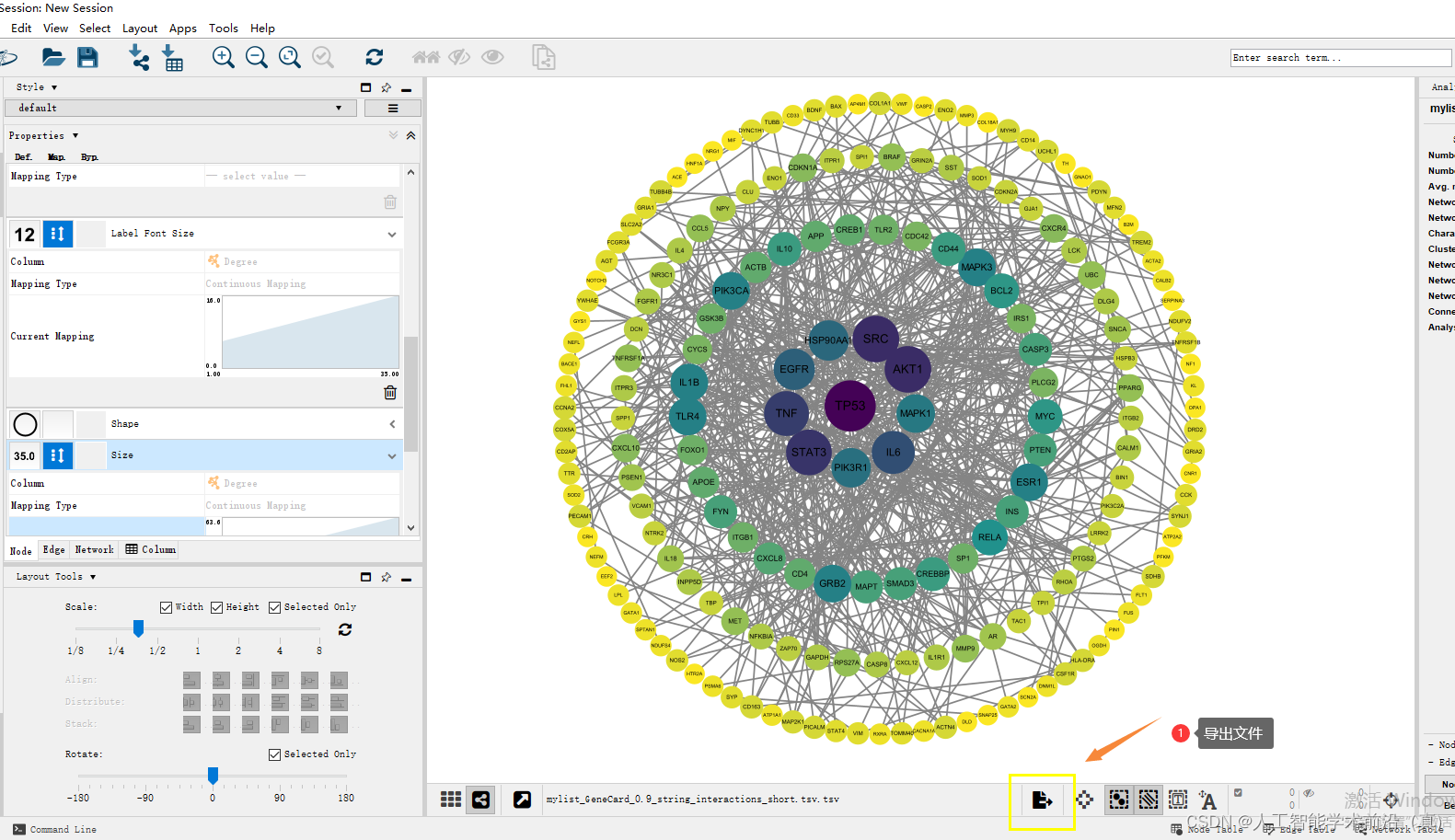
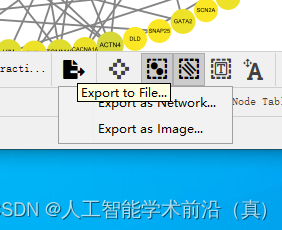
可以导出session项目文件,后续可以继续编辑,可以导出图片,再论文中使用。
以上绘图共做就完成了,SCI 指日可待。