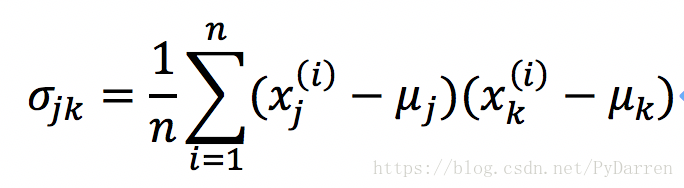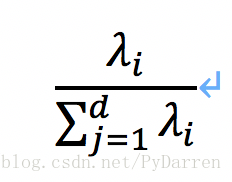如果一个模型在训练集上的表现要好于交叉验证集和测试集的化,这意味着模型是过拟合的。过拟合是指模型对训练集数据的特定观察值拟合的非常近,但训练集和测试集的真实数据分布并不一定是一致的,即模型存在较高的方差。产生过拟合的原因是建立在训练集上的模型过于复杂,而常用的降低泛化误差的方案有:
- 收集更多的训练数据;
- 通过正则化引入罚项(在逻辑回归中已论述);
- 选择一个参数较少的简单的模型;
- 降低数据的维度;
通过特征选择进行降维的方法对未经正则化处理的模型特别有效。降维技术主要分为两类:特征选择和特征提取。通过特征选择,可以选出原始特征的一个子集。对于特征提取,是通过对现有的特征信息进行推演,构造出一个新的特征子空间。
一、序列特征选择算法
序列特征选择算法是一种贪婪搜索算法,用于将原始的d维特征空间压缩到一个k维特征子空间,其中k<d。使用该算法出于对考虑是:能够剔除不相关特征或噪声,自动选出与问题最相关的特征子集,从而提高计算效率或是降低模型的泛化误差。这在模型不支持正则化时尤为有效。
一个经典的算法是序列向后选择算法(Sequential Backward Selection, SBS),其目的是在分类性能衰减最小的约束下,降低原始特征空间上的数据维度,以提高计算效率。某些情况下SBS可以在模型面临过拟合问题时提高模型的预测精度。
SBS算法理念:依次从特征集合中删除某些特征,直到新的特征子空间包含指定数量的特征。为了确定每一步需要删除的特征,我们定义一个需要最小化的标准衡量函数J,该函数的计算准则是:比较分类器的性能在删除某个特征前后的差异。因此每一步中待删除的特征就是那些能够使得标准衡量函数值尽可能大的特征,即使得模型损失性能最小的特征。
由于sklearn中没有现成的SBS算法,下面是手撸的版本:
1. from sklearn.base import clone
2. from itertools import combinations
3. import numpy as np
4. from sklearn.cross_validation import train_test_split
5. from sklearn.metrics import accuracy_score #衡量分类器的模型和评估器在特征空间上的性能
6.
7.
8. class SBS(object):
9.
10. def __init__(self, estimator, k_features, scoring=accuracy_score, test_size=0.25, random_state=1):
11. self.scoring = scoring
12. self.estimator = clone(estimator) # 模型深拷贝,不附加任何数据
13. self.k_features = k_features # 指定返回的特征数量
14. self.test_size = test_size
15. self.random_state = random_state # 设定随机数种子,数字对应的系统时间生成的随机数是相同的
16.
17. def fit(self, X, y):
18. X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=self.test_size,
19. random_state=self.random_state)
20.
21. dim = X_train.shape[1] # 矩阵的行维度
22. self.indices_ = tuple(range(dim)) #最终特征子集的列标
23. self.subsets_ = [self.indices_]
24. score = self._calc_score(X_train, y_train, X_test, y_test, self.indices_)
25.
26. self.scores_ = [score]
27.
28. while dim > self.k_features:
29. scores = []
30. subsets = []
31.
32. for p in combinations(self.indices_, r=dim - 1): #combinations用来循环创建特征子集进行评估删减
33. score = self._calc_score(X_train, y_train, X_test, y_test, p)
34. scores.append(score)
35. subsets.append(p)
36.
37. best = np.argmax(scores)
38. self.indices_ = subsets[best]
39. self.subsets_.append(self.indices_)
40. dim -= 1
41.
42. self.scores_.append(scores[best])
43. self.k_score_ = self.scores_[-1] #得出k个特征下的最优分数
44.
45. return self
46.
47. def transform(self, X):
48. return X[:, self.indices_] #返回由选定的特征列构成的新数组
49.
50. def _calc_score(self, X_train, y_train, X_test, y_test, indices):
51. self.estimator.fit(X_train[:, indices], y_train)
52. y_pred = self.estimator.predict(X_test[:, indices])
53. score = self.scoring(y_test, y_pred)
54. return score
将SBS算法应用于KNN分类器:
1. #将SBS应用于sklearn中KNN分类器
2. from sklearn.neighbors import KNeighborsClassifier
3. knn = KNeighborsClassifier(n_neighbors=2)
4. sbs = SBS(knn, k_features=1)
5. sbs.fit(X_train_std,y_train)
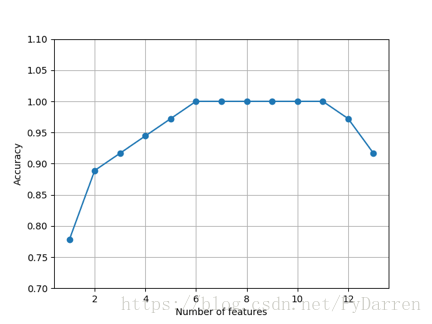
6. k_feat = [len(k) for k in sbs.subsets_]
7. plt.plot(k_feat, sbs.scores_, marker='o')
8. plt.ylim([0.7,1.1])
9. plt.ylabel('Accuracy')
10. plt.xlabel("Number of features")
11. plt.grid()
12. plt.savefig("/Users/chenda/Desktop/1.png")
13. plt.show()
可以看出,当特征数量为6-10时,算法准确率达到100%,下面查看一下哪6个特征在验证集有如此好的表现。
1. k6 = list(sbs.subsets_[7])
2. print(df_wine.columns[1:][k6])
3.
4. Index(['Alcohol', 'Malic acid', 'Ash', 'Proanthocyanins', 'Color intensity',
5. 'Proline'],
6. dtype='object')
比较KNN分类器在原始数据集和新的6个特征集上的表现:
1. knn.fit(X_train_std ,y_train)
2. print("Old training accuracy: {}".format(knn.score(X_train_std,y_train)))
3. print("Old test accuracy: {}".format(knn.score(X_test_std,y_test)))
4.
5. knn.fit(X_train_std[:,k6],y_train)
6. print("New training accuracy: {}".format(knn.score(X_train_std[:,k6],y_train)))
7. print("New test accuracy: {}".format(knn.score(X_test_std[:,k6],y_test)))
8.
9. Old training accuracy: 0.9859154929577465
10. Old test accuracy: 0.9166666666666666
11. New training accuracy: 0.971830985915493
12. New test accuracy: 0.9722222222222222
可以看出过拟合现象明显缓解。
二、通过随机森林判定特征的重要性(待更)
三、主成分分析
与特征选择类似,可以通过特征抽取来减少数据集中特征的数量。不过,当使用序列后向选择等特征选择算法时,能够保持数据等原始特征,而特征抽取算法则会将数据转换或者映射到一个新的特征空间,从而降低“维度灾难”,尤其在模型不适合正则化处理时。
主成分分析(principal component analysis,PCA)是一种广泛应用于不同领域的无监督线性数据转换技术,这意味着我们可以忽略类标信息。其目标是在高维度数据中找到最大方差的方向,并将数据映射到一个维度不大于原始数据的子空间上。第一主成分的方差应该是最大的,各主成分之间相互正交,后续各主成分也具备尽可能大的方差。由于主成分的方向对数据值的范围高度敏感,故需要对特征进行标准化处理,以让各特征具有相同的重要性。
- 算法流程:
- 标准化原始数据(很重要,否则速度会非常慢);
- 构造样本的协方差矩阵;
- 计算协方差矩阵的特征值和相应的特征向量;
- 选择与前k个最大特征值对应的特征向量;
- 通过k个特征向量构建映射矩阵;
- 通过映射矩阵W将d维的输入数据集X转换到新的k维特征子空间。
按流程手撸版本:
<一> 导入数据
1. #导入数据并预处理
2. import pandas as pd
3. df_wine = pd.read_csv("http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data", header=None)
4. df_wine.columns = [
5. 'Class label','Alcohol',
6. 'Malic acid','Ash',
7. 'Alcalinity of ash','Magnesium',
8. 'Total phenols','Flavanoids',
9. 'Nonflavanoid phenols',
10. 'Proanthocyanins',
11. 'Color intensity','Hue',
12. 'OD280/OD315 of diluted wines',
13. 'Proline',
14. ]
15.
16. from sklearn.cross_validation import train_test_split
17. X,y = df_wine.iloc[:,1:].values, df_wine.iloc[:,0].values
18. X_train,X_test,y_train,y_test = train_test_split(X, y, test_size=0.2, random_state=0)
19.
20. from sklearn.preprocessing import StandardScaler
21. std = StandardScaler()
22. X_train_std = std.fit_transform(X_train)
23. X_test_std = std.fit_transform(X_test)
<二> 构造协方差矩阵
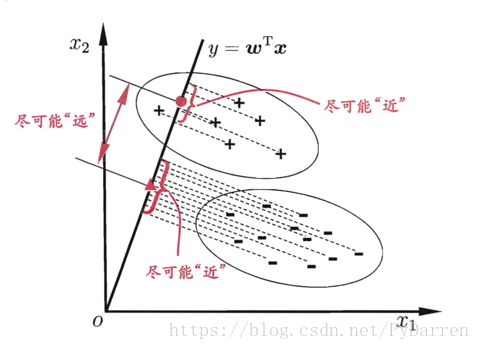
两个特征xj和xk之间的协方差为:
其中μ为均值,在标准化之后为零。两个特征之间的协方差如果为正,说明它们会同时增减,为负的话则两个特征朝着相反的方向变动。
协方差矩阵的特征向量代表主成分(最大方差方向),而对应的特征值大小就决定了特征向量的重要性。
1. #构造协方差矩阵
2. import numpy as np
3. cov_mat = np.cov(X_train_std.T) #得到协方差矩阵
4. eigen_vals, eigen_vecs = np.linalg.eig(cov_mat) #得到特征值和对应的特征向量
5. print("\nEigenValues \n%s" % eigen_vals)
6. print(eigen_vecs)
特征值λi的方差贡献率是指,特征值λi与所有特征值之和的比值:
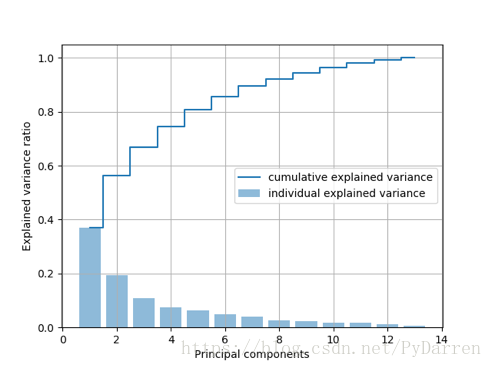
1. tot = sum(eigen_vals)
2. var_exp = [(i/tot) for i in
3. sorted(eigen_vals, reverse=True)]
4. cum_var_exp = np.cumsum(var_exp) #计算累计方差
5.
6. import matplotlib.pyplot as plt
7. plt.bar(range(1,14), var_exp, alpha=0.5, align='center',
8. label = 'individual explained variance')
9. plt.step(range(1,14), cum_var_exp, where='mid',
10. label='cumulative explained variance')
11. plt.xlabel("Principal components")
12. plt.ylabel("Explained variance ratio")
13. plt.grid()
14. plt.legend(loc='best')
15. plt.savefig("/Users/chenda/Desktop/2.png")
16. plt.show()
<三> 特征转换
将数据集中的信息转换到新的主成分轴上。对特征值进行降序排列,通过挑选出的特征值对应的特征向量构造出映射矩阵,然后使用映射矩阵将数据集转换到低维的子空间。
1. # ###特征转换
2. eigen_pairs = [(np.abs(eigen_vals[i]),eigen_vecs[:,i])
3. for i in range(len(eigen_vals))] #获取特征对
4. eigen_pairs.sort(reverse=True) #降序排列
5.
6. w = np.hstack((eigen_pairs[0][1][:, np.newaxis],
7. eigen_pairs[1][1][:, np.newaxis])) #选择前两个特征向量得到13*2的映射矩阵
8. print("Matrix W: \n", w)
9.
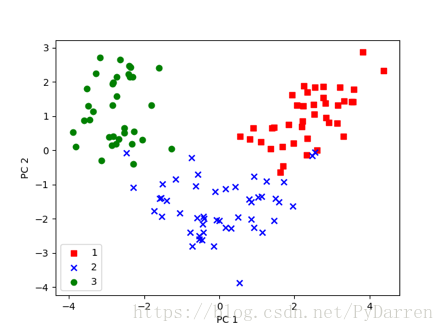
10. X_train_pca = X_train_std.dot(w) #通过计算点积,可以将整个124*3维的训练集转换到包含两个主成分的子空间上
11.
12. #进行可视化展示
13. colors = ['r','b','g']
14. makers = ['s','x','o']
15. for l ,c,m in zip(np.unique(y_train),colors,makers):
16. plt.scatter(X_train_pca[y_train==l,0],
17. X_train_pca[y_train==l,1],
18. c=c, label=l, marker=m)
19. plt.xlabel("PC 1")
20. plt.ylabel("PC 2")
21. # plt.grid()
22. plt.legend(loc="best")
23. plt.savefig("/Users/chenda/Desktop/3.png")
24. plt.show()
使用sklearn进行主成分分析:
1. #使用sklearn进行PCA
2. from matplotlib.colors import ListedColormap
3.
4. def plot_decision_regions(X, y, classifier, resolution=0.02):
5. '''''
6. 决策区域可视化函数
7. :param X: 特征集
8. :param y: 类标
9. :param classifier: 分类器
10. :param resolution:
11. '''
12.
13. #设置对应的标示和颜色
14. markers = ('s','x','o','^','v')
15. colors = ('red','blue','lightgreen','gray','cyan')
16. cmap = ListedColormap(colors[:len(np.unique(y))]) #生成颜色示例图
17.
18. #绘制决策区域
19. x1_min, x1_max = X[:,0].min() - 1, X[:,0].max() + 1
20. x2_min, x2_max = X[:1].min() - 1, X[:,1].max() + 1
21. xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
22. np.arange(x2_min, x2_max, resolution)) #将最大、最小值向量生成2维数组
23. Z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T) #得到预测的类标,ravel将多维数组降低到一维
24. Z = Z.reshape(xx1.shape) #将Z转换成相同维度
25. plt.contourf(xx1, xx2, Z, alpha=0.4, cmap=cmap) #对不同类绘制不同颜色对区域
26. plt.xlim(xx1.min(), xx1.max())
27. plt.ylim(xx2.min(), xx2.max())
28.
29. for idx, cl in enumerate(np.unique(y)):
30. plt.scatter(x=X[y == cl, 0], y=X[y == cl, 1],
31. alpha=0.8, c=cmap(idx),
32. marker=markers[idx], label=cl)
33.
34.
35.
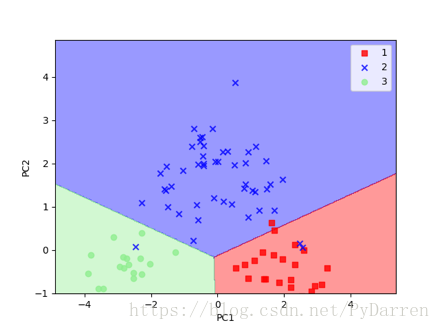
36. import numpy as np
37. import matplotlib.pyplot as plt
38. from sklearn.linear_model import LogisticRegression
39. from sklearn.decomposition import PCA
40. pca = PCA(n_components=2)
41. lr = LogisticRegression()
42. X_train_pca = pca.fit_transform(X_train_std)
43. X_test_pca = pca.fit_transform(X_test_std)
44. lr.fit(X_train_pca, y_train)
45. plot_decision_regions(X_train_pca, y_train, classifier=lr)
46. plt.xlabel("PC1")
47. plt.ylabel("PC2")
48. plt.legend(loc="best")
49. plt.savefig('/Users/chenda/Desktop/4.png')
50. plt.show()
可以看出与之前手撸的版本图像沿着y轴反转了,原因是特征向量的符号互为相反数,有需要的话在数据上乘以-1即可实现图像的镜像。
四、线性判别分析
线型判别分析(Linear Discriminant Analysis, LDA)是一种可以作为特征提取的技术,可以提高数据分析过程中的计算效率,同时对不适用于正则化的模型,它可以降低因维度灾难导致的过拟合。PCA试图在数据集中找到方差最大的正交的主成分量的轴,而LDA的目标是发现可以最优化分类的特征子空间。PCA是无监督算法,LDA是监督算法,LDA是一种更优越的用于分类的特征提取技术。
LDA的思想非常朴素:给定训练样集,设法将样本投射到一条直线上,使得同类尽可能近,异类尽可能远;在对新样本进行分类时,投射到同一直线上,再根据投影点的位置来确定新样本的类别。
关于LDA的假定:
- 数据呈正态分布;
- 各类别数据具有相同的协方差矩阵;
- 样本间相互独立。
- 即使一个或多个假定不成立,LDA仍旧可以很好地完成降维工作。
算法步骤:
- 对d维数据进行标准化处理;
- 对于每一个类别,计算d维的均值向量;
- 构造类间的散布矩阵SB以及类内的散布矩阵SW;
- 计算矩阵SW^(-1)SB的特征值及对应的特征向量;
- 选取前k个特征值和特征向量,构造一个d×k维的转换矩阵W,其中特征向量以列的形式排列;
- 使用转换矩阵W将样本映射到新的特征子空间上。
1. # coding: utf-8
2.
3. # In[1]:
4.
5.
6. #Created by: Chen Da
7. #Created on: 2018/8/4
8.
9. import numpy as np
10. import pandas as pd
11.
12. #导入数据
13. df_wine = pd.read_csv("https://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data",header=None)
14.
15. #划分训练集和测试机并进行标准化
16. from sklearn.cross_validation import train_test_split
17. from sklearn.preprocessing import StandardScaler
18.
19. X,y = df_wine.iloc[:,1:].values, df_wine.iloc[:,0].values
20. X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0)
21.
22. std = StandardScaler()
23. X_train_std = std.fit_transform(X_train)
24. X_test_std = std.fit_transform(X_test)
25.
26.
27. # In[3]:
28.
29.
30. from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
31.
32.
33. # In[4]:
34.
35.
36. lda = LDA(n_components=2)
37.
38.
39. # In[5]:
40.
41. X_train_lda = lda.fit_transform(X_train_std, y_train)
42.
43.
44. # In[6]:
45.
46.
47. from sklearn.linear_model import LogisticRegression
48.
49.
50. # In[7]:
51.
52.
53. lr = LogisticRegression()
54.
55.
56. # In[8]:
57.
58.
59. lr.fit(X_train_lda, y_train)
60.
61.
62. # In[9]:
63.
64.
65. from matplotlib.colors import ListedColormap
66. import matplotlib.pyplot as plt
67.
68. def plot_decision_regions(X,y,classifier,resolution=0.02):
69. '''''
70. 决策区域可视化展示
71. :param X:
72. :param y:
73. :param classifier:
74. :param resolution:
75. :return:
76. '''
77.
78. #setup marker genertor and color map
79. markers = ('s','x','o','^','v')
80. colors = ('red','blue','lightgreen','gray','cyan')
81. cmap = ListedColormap(colors[:len(np.unique(y))])
82.
83. # #plot the decision surface
84. # x1_min,x1_max = X[:,0].min() - 1, X[:,0].max() + 1
85. # x2_min,x2_max = X[:,1].min() - 1, X[:,1].max() + 1
86. # xx1,xx2 = np.meshgrid(np.arange(x1_min,x1_max,resolution),
87. # np.arange(x2_min,x2_max,resolution))
88. # Z = classifier.predict(np.array([xx1.ravel(),xx1.ravel()]).T)
89. # Z = Z.reshape(xx1.shape)
90. # plt.contourf(xx1,xx2,Z,alpha=0.4,cmap=cmap)
91. # plt.xlim(xx1.min(),xx1.max())
92. # plt.ylim(xx2.min(),xx2.max())
93.
94. #plot class sample
95. for idx,cl in enumerate(np.unique(y)):
96. plt.scatter(x=X[y == cl,0], y=X[y == cl,1],
97. alpha=0.8, c=cmap(idx), marker=markers[idx], label=cl)
98.
99.
100. # In[11]:
101.
102.
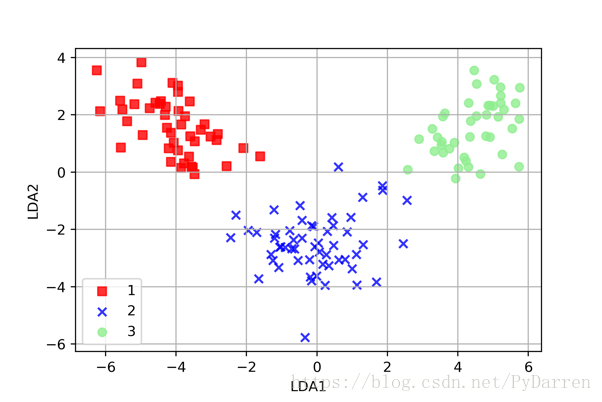
103. plot_decision_regions(X_train_lda, y_train, classifier=lr)
104. plt.xlabel('LDA1')
105. plt.ylabel('LDA2')
106. plt.grid()
107. plt.legend(loc='best')
108. plt.show()
五、核主成分分析
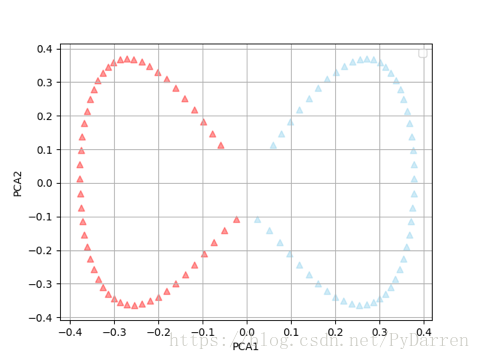
针对现实世界的非对称问题,采用核PCA可以通过非线性映射将数据转换到一个高维空间,然后在此高维空间中使用标准PCA将其映射到另外一个低维空间中,并通过线性分类器对其进行划分。其高昂的计算成本通过核技巧进行解决。
1. import matplotlib.pyplot as plt
2. from sklearn.datasets import make_moons
3. from sklearn.decomposition import KernelPCA
4.
5. X, y = make_moons(n_samples=100,random_state=123)
6.
7. scikit_pca = KernelPCA(n_components=2, kernel='rbf', gamma=13)
8. X_kenel_pca = scikit_pca.fit_transform(X)
9.
10. plt.scatter(X_kenel_pca[y == 0, 0], X_kenel_pca[y == 0, 1],
11. c='red', alpha=0.4, marker='^')
12. plt.scatter(X_kenel_pca[y == 1, 0], X_kenel_pca[y == 1, 1],
13. c='skyblue', alpha=0.4, marker='^')
14. plt.xlabel('PCA1')
15. plt.ylabel('PCA2')
16. plt.legend(loc='best')
17. plt.grid()
18. plt.show()