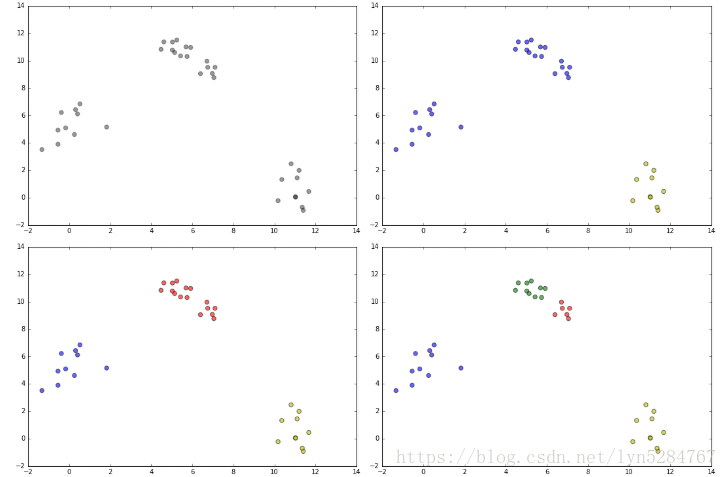
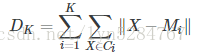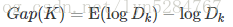一,介绍
采用K均值算法进行聚类,首先需要做的是确定K的个数,一般来讲,有以下几种方法:
1、按需选择
简单地说就是按照建模的需求和目的来选择聚类的个数。比如说,一个游戏公司想把所有玩家做聚类分析,分成顶级、高级、中级、菜鸟四类,那么K=4;如果房地产公司想把当地的商品房分成高中低三档,那么K=3。按需选择虽然合理,但是未必能保证在做K-Means时能够得到清晰的分界线。
2、观察法
就是用肉眼看,看这些点大概聚成几堆。这个方法虽然简单,但是同时也模棱两可。
左上角是原始点。右上角分成了两类。左下角是三类,左下角是四类。至于K到底是选3还是选4,可能每个人都有不同的选择。
观察法的另一个缺陷就是:原始数据维数要低,一般是两维(平面散点)或者三维(立体散点),否则人类肉眼则无法观察。对于高维数据,我们通常利用PCA降维,然后再进行肉眼观察。
3、手肘法
手肘法本质上也是一种间接的观察法。我们将得到K个聚类的中心点Mi, i=1,2,⋯,K。以及每个原始点所对应的聚类Ci,i=1,2,⋯,K。我们通常采用所有样本点到它所在的聚类的中心点的距离的和作为模型的度量,记为DK。
这里距离可以采用欧式距离。
对于不同的K,最后我们会得到不同的中心点和聚类,所有会有不同的度量。
我们把上面的例子用不同的K去计算,会得到不同的结果。把K作为横坐标,DK作为纵坐标,我们可以得到下面的折线。
很显然K越大,距离和越小。但是我们注意到K=3是一个拐点,就像是我们的肘部一样,K=1到3下降很快,K=3之后趋于平稳。手肘法认为这个拐点就是最佳的K。
手肘法是一个经验方法,而且肉眼观察也因人而异,特别是遇到模棱两可的时候。相比于直接观察法,手肘法的一个优点是,适用于高维的样本数据。有时候人们也会把手肘法用于不同的度量上,如组内方差组间方差比。
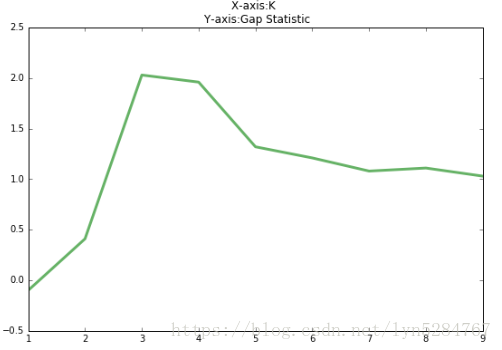
4、Gap Statistic方法
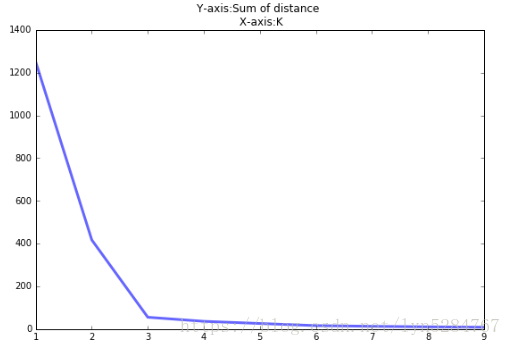
这里我们要继续使用上面的DK。Gap Statistic的定义为:
这里E(logDk)指的是logDk的期望。这个数值通常通过蒙特卡洛模拟产生,我们在样本里所在的矩形区域中(高维的话就是立方体区域)按照均匀分布随机地产生和原始样本数一样多的随机样本,并对这个随机样本做聚类,从而得到一个DK。如此往复多次,通常20次,我们可以得到20个logDK。对这20个数值求平均值,就得到了E(logDK)的近似值。最终可以计算Gap Statisitc。而Gap statistic取得最大值所对应的K就是最佳的K。
用上图的例子,我们计算了K=1,2,..9对应的Gap Statisitc.
确定好K的个数后,算法开始,算法流程如下:
1,确定K个聚类的中心;
2,计算每个点与各个中心的距离,选择距离最近的中心成为其分类,公式如下:
3,更新聚类的中心;
4,重复上述步骤直到结束
二,代码
数据如下:
0.697 0.46 0.774 0.376 0.634 0.264 0.608 0.318 0.556 0.215 0.403 0.237 0.481 0.149 0.437 0.211 0.666 0.091 0.243 0.267 0.245 0.057 0.343 0.099 0.639 0.161 0.657 0.198 0.36 0.37 0.593 0.042 0.719 0.103 0.359 0.188 0.339 0.241 0.282 0.257 0.748 0.232 0.714 0.346 0.483 0.312 0.478 0.437 0.525 0.369 0.751 0.489 0.532 0.472 0.473 0.376 0.725 0.445 0.446 0.459
python代码:
import matplotlib.pyplot as plt
from numpy import *
import random
def loadDataSet(filename):
fr = open(filename)
numberOfLines = len(fr.readlines())
returnMat = zeros((numberOfLines, 2))
classLabelVector = ['密度','含糖率']
fr = open(filename)
index = 0
for line in fr.readlines():
line = line.strip().split('\t')
returnMat[index, :] = line[0:2]
index += 1
return returnMat, classLabelVector
# 欧几里得距离
def edistance(v1, v2):
result=0.0
for i in range(len(v1)):
result +=(v1[i]-v2[i])**2
return sqrt(result)
# 特征值归一化
def autoNorm(dataSet):
minVals = dataSet.min(0) # 获取特征值最小值
maxVals = dataSet.max(0) # 获取特征值最大值
ranges = maxVals - minVals
m = dataSet.shape[0]
normDataSet = dataSet - tile(minVals, (m,1))
normDataSet = normDataSet/tile(ranges, (m,1)) #归一化
return normDataSet, ranges, minVals
def kcluster(rows, distance=edistance, k=3):
normDataSet, ranges, minVals = autoNorm(rows) # 归一化数据到0-1之间
count = normDataSet.shape[0] # 数据总数
randinfo = random.sample(range(0, count), k)
clusters = [normDataSet[randinfo[i]] for i in range(len(randinfo))] # 随机选取k个值作为聚类中心
lastmatches = None
for t in range(100):
bestmatches = [[] for i in range(k)]
# 寻找最近中心
for j in range(count):
row = normDataSet[j]
bestmatch = 0
for i in range(k):
d = distance(row,clusters[i])
if d < distance(row,clusters[bestmatch]): bestmatch = i
bestmatches[bestmatch].append(j)
# 如果没有变化则认为最佳,退出循环
if bestmatches == lastmatches: break
lastmatches = bestmatches
# 移动聚类的中心
for i in range(k):
avgs = [0.0] * len(normDataSet[0])
if len(bestmatches[i]) > 0:
for rowid in bestmatches[i]:
for m in range(len(normDataSet[rowid])):
avgs[m] += normDataSet[rowid][m]
for j in range(len(avgs)):
avgs[j] /= len(bestmatches[i])
clusters[i] = avgs
return bestmatches
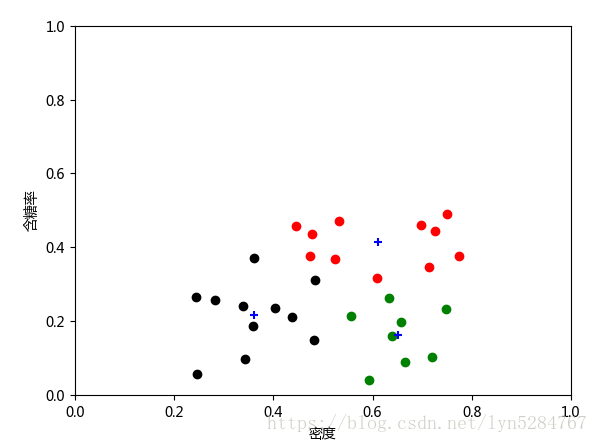
def plot(dataMat, labelMat,bestmatches):
xcord = [];ycord = []
sumx1=0.0;sumy1=0.0;sumx2=0.0;sumy2=0.0;sumx3=0.0;sumy3=0.0
midx = [];midy=[]
for i in range(len(dataMat)):
xcord.append(float(dataMat[i][0]));ycord.append(float(dataMat[i][1]))
for i in range(len(bestmatches)):
for j in bestmatches[i]:
if(i==0):
plt.scatter(xcord[j], ycord[j], color='red')
sumx1+=xcord[j]
sumy1+=ycord[j]
if(i == 1):
plt.scatter(xcord[j], ycord[j], color='green')
sumx2 += xcord[j]
sumy2 += ycord[j]
if (i == 2):
plt.scatter(xcord[j], ycord[j], color='black')
sumx3 += xcord[j]
sumy3 += ycord[j]
midx.append(sumx1/len(bestmatches[0]))
midx.append(sumx2 / len(bestmatches[1]))
midx.append(sumx3 / len(bestmatches[2]))
midy.append(sumy1 / len(bestmatches[0]))
midy.append(sumy2 / len(bestmatches[1]))
midy.append(sumy3 / len(bestmatches[2]))
plt.scatter(midx,midy, marker = '+',color='blue')
plt.xlabel(labelMat[0]);plt.ylabel(labelMat[1])
plt.xlim((0, 1))
plt.ylim((0, 1))
plt.show()
if __name__=='__main__':
dataMat, labelMat = loadDataSet('watermelon4.0.txt')
bestmatches = kcluster(dataMat)
plot(dataMat, labelMat,bestmatches)
结果: