论文下载链接为:
https://link.springer.com/search?query=Cell+tracking+via+Structured+Prediction+and+Learning
如果有下载不了的同学找小鱼,小鱼可以分享文档给你。
这篇论文的特点在于:
特点1、增加了一个分割层来产生分割掩码,得到更精确的cell proposal。
小鱼看了很久才明白分割掩码到底是什么,小鱼认为分割掩码和类别预测很重要,是为每个类别分别预测一个二元掩码,对物体进行像素级别的分割提取,对目标的空间布局有一个更加精确的提取,对每个RoI都生成了一个二元掩码。如果还有不懂的可以参照这篇博客: Mask R-CNN论文解读
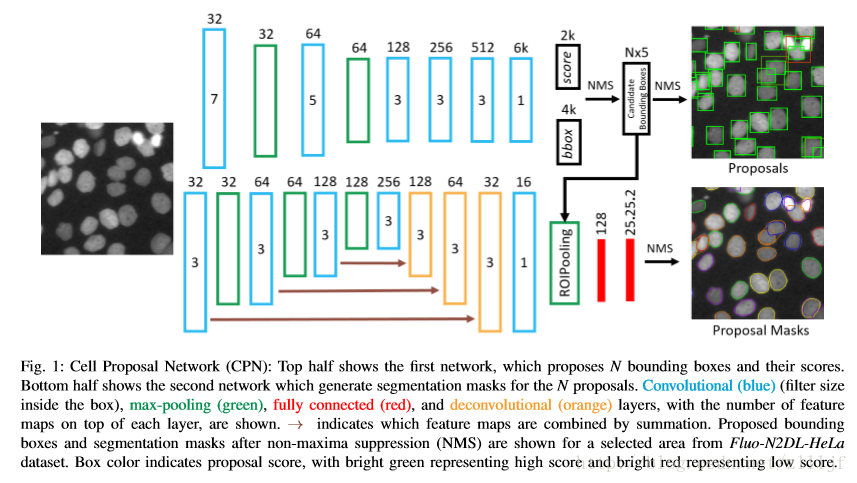
这个网络结果包括两部分:
1)Bounding box network
所有的卷积步幅为1,所有的最大池化为3*3步幅为2,图中上半部分过程为:
input – conv1(7*7 ) – max-pooling(3*3) – conv2(5*5 ) – max-pooling – conv3(3*3) – conv4(3*3) – conv5(3*3) – conv(6K)
这个网络的扩展为将原来RPN的全连接层改为全卷积层,其中包括2K score和4K bbox,最后用非极大值抑制NMS,设置IoU=0.8得到候选边界框。
2)Segmentation network
该网络包括两部分:
a、所有的卷积层都用3*3的步幅为1的卷积核,所有的最大池化用3*3步幅为1,图中下半部分过程为:
input - conv1 - max-pooling - conv2 - maxpooling - conv3 - maxpooling - conv4 - deconv - deconv - deconv - conv5 - pooling - fc - fc
该过程中的扩展为:五层卷积,conv1、conv2、conv3、conv4提取不同深度的特征图,得低细及高粗信息。接着conv2、conv3、conv4反卷积再相加得到和输入相同分辨率的特征图;conv5可在所有的候选边界框得到共享,同时conv5后的pooling相当于resize conv5的特征图,为每个候选框提取固定大小的特征图来预测分割掩码。mask是通过crop GT分割图来获取的。
特点2:用有向无环图来表示细胞跟踪。
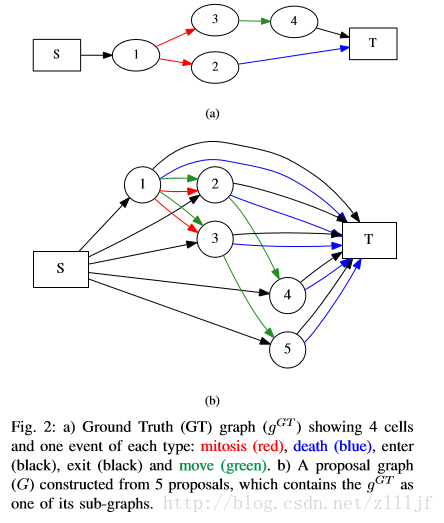
上部分表示一个GT图,包括了细胞运动的所有过程:出现,有丝分裂,移动,消失,死亡。
下部分图主要是用来表示选择子图的过程,所以细胞跟踪问题转换为选择最大可能性子图的问题,选择公式为:

因为图形模型中的提议节点:G,有与它们相关的代价,这取决于该建议节点为细胞的可能性。所以为每个边界都设置了特征集表示方式:
a、对于移动边界:
训练一个随机森林分类器,在测试集序列中用于对移动边界设置权重值,对移动边界进行分类的特征集包括:

b、对于有丝分裂边界:
训练一个随机的森林分类器,用于对有丝分裂边缘进行打分的特征集包括:

c、对于细胞死亡事件的序列,可以通过使用前面列出的形状和外观特征,或者其他特定的任务特定的特征来训练分类器,预测细胞死亡的可能性。
d、对于进入和退出边界,我们对每个序列使用一个恒定的常数概率表示。
特点3:对跟踪进行优化。
这部分看的不是很懂,就是用比较了优化细胞跟踪方法ILP和KSP,因为这两个方法小鱼不是很懂,所以这里不做太多解释,有问题可以相互交流。
最后,就是结果的展示,不难懂,但是很有价值看,比如一些评价指标,对比实验怎么做等,还是很有启发价值的。
这里小鱼稍微讲一下本论文的数据集:两个荧光显微镜数据(Fluo-N2DH-GOWT1和Fluo-N2DH-Hela)和两个相称显微镜数据(PhC-C2DH-U373和PhC-C2DL-PSC)这些数据可以在论文中提供的连接进行下载,如果有需要的可以去下载。
论文笔记之《Cell Tracking via Proposal Generation and Selection》
猜你喜欢
转载自blog.csdn.net/zllljf/article/details/78827844
今日推荐
周排行