香港疫情数据回归预测
在这篇博客中,我们将探讨如何使用回归预测在疫情中的确诊人数、痊愈人数和死亡人数。我们将通过特征工程、模型评估和选择等步骤完成这个任务。以下是详细的分析和讨论。
准备工作
导入相关模块
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.ensemble import RandomForestRegressor
from sklearn.linear_model import Ridge, LinearRegression
from sklearn.metrics import mean_squared_error
from sklearn.model_selection import KFold, cross_val_score
from sklearn.preprocessing import PolynomialFeatures
from sklearn.tree import DecisionTreeRegressor
数据读取与时间粒度处理
数据集包含了关于疫情的信息,如确诊人数、痊愈人数、死亡人数、更新时间等。我们选择了省份编码为810000(香港)的数据,然后对数据进行了预处理。
df = pd.read_csv('DXYArea.csv')
df = df.loc[df['province_zipCode'] == 810000]
将时间列转换为时间戳,并将时间戳转化为与最小值的差值,选取分钟和天作为特征,提取为updateTimeM和updataTimeD这两列。
df['updateTime'] = pd.to_datetime(df['updateTime'])
df['updateTimeM'] = df['updateTime'].apply(lambda x: (x - df['updateTime'].min()).total_seconds() / 60).astype(int)
df['updataTimeD'] = df['updateTime'].apply(lambda x: (x - df['updateTime'].min()).total_seconds() / 86400).astype(int)
确诊人数预测
特征观察
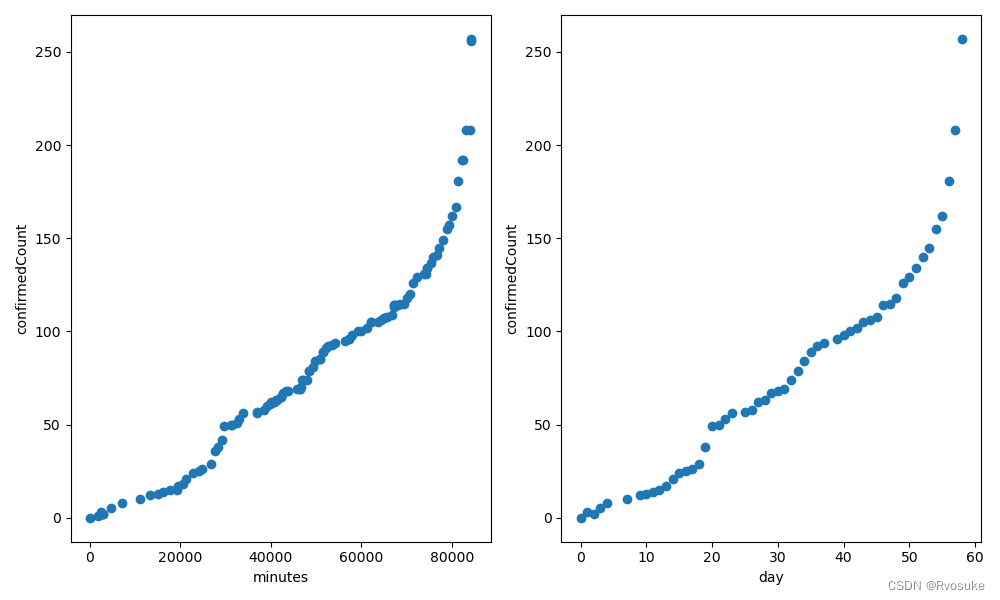
利用matplotlib库来分别绘制分钟和确诊人数、天和确诊人数之间的散点图。
plt.figure(figsize=(10, 6))
plt.subplot(1, 2, 1)
plt.scatter(df['updateTimeM'], df['province_confirmedCount'])
plt.xlabel('minutes')
plt.ylabel('confirmedCount')
根据 updataTimeD分组,并选取每组 province_confirmedCount’的最大值
df_max = df.groupby('updataTimeD').agg({
'province_confirmedCount': 'max'}).reset_index()
X_data_day = df_max['updataTimeD'].values.reshape(-1, 1)
Y_data_day = df_max['province_confirmedCount'].values.reshape(-1, 1)
plt.subplot(1, 2, 2)
plt.scatter(X_data_day, Y_data_day)
plt.xlabel('day')
plt.ylabel('confirmedCount')
plt.tight_layout()
plt.show()
模型拟合与选择
在进行数据可视化之后,我们发现选取天还是分钟作为特征,图形差异不大,为了获取能从更多的样例中训练,我们选取分钟作为特征进行模型训练。
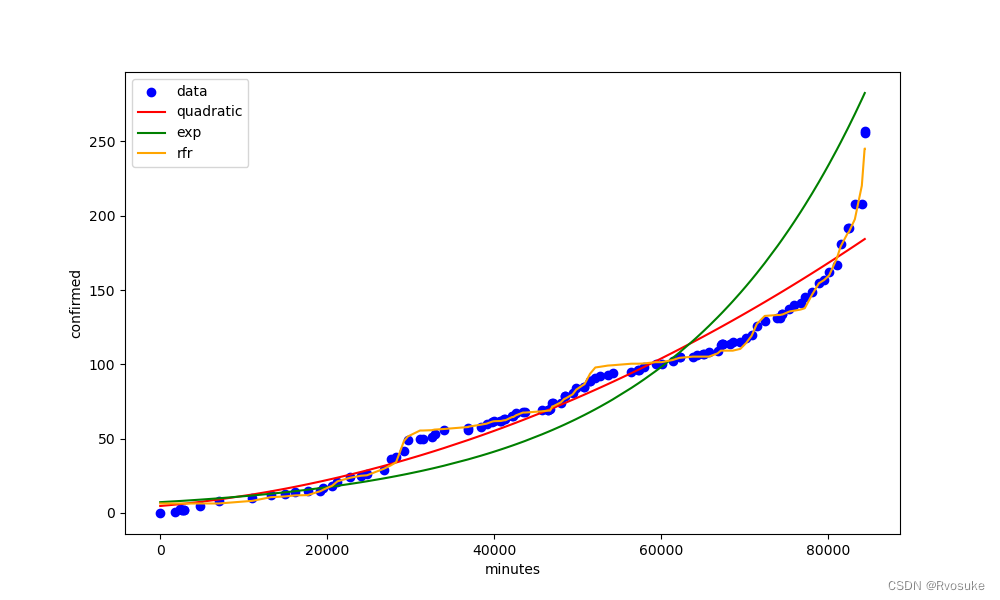
在图形观察后,我们考虑可以使用多项式回归,指数形式的线性回归,随机森林回归等模型进行训练,然后使用交叉验证的方法来评估模型的效果,
并绘制出拟合曲线,其中多项式回归中我们设置为2次。
选取随机森林回归的原因是,样本量比较大,若采用决策树回归,容易过拟合,而随机森林回归可以有效的减少过拟合的情况。
X_data = df['updateTimeM'].values.reshape(-1, 1) # 选取分钟作为特征
Y_data = df['province_confirmedCount'].values.reshape(-1, 1)
plt.figure(figsize=(10, 6))
plt.scatter(X_data, Y_data, color='blue', label='data')
# 多项式模型建立与拟合
pr = LinearRegression()
quadratic = PolynomialFeatures(degree=2)
X_quad = quadratic.fit_transform(X_data)
pr.fit(X_quad, Y_data)
Y_pred_quad = pr.predict(X_quad)
plt.plot(X_data, Y_pred_quad, color='red', label='quadratic')
# 指数形式的线性回归模型建立与拟合
elr = LinearRegression()
elr.fit(X_data, np.log1p(Y_data))
Y_pred_exp = np.exp(elr.predict(X_data))
plt.plot(X_data, Y_pred_exp, color='green', label='exp')
# 随机森林回归模型建立与拟合
rfr = RandomForestRegressor(n_estimators=100, max_depth=3)
rfr.fit(X_data, Y_data)
Y_pred_rfr = rfr.predict(X_data)
plt.plot(X_data, Y_pred_rfr, color='orange', label='rfr')
# 设置图例和标签
plt.legend(loc='upper left')
plt.xlabel('minutes')
plt.ylabel('confirmed')
plt.show()
交叉验证
mse1 = cross_val_score(rfr, X_data, Y_data, scoring='neg_mean_squared_error', cv=5)
mse2 = cross_val_score(pr, X_data, Y_data, scoring='neg_mean_squared_error', cv=5)
mse3 = cross_val_score(elr, X_data, Y_data, scoring='neg_mean_squared_error', cv=5)
print('Minute 随机森林 RMSE mean: ', np.sqrt(-np.mean(mse1)))
print('Minute 多项式 RMSE mean: ', np.sqrt(-np.mean(mse2)))
print('Minute 指数 RMSE mean: ', np.sqrt(-np.mean(mse3)))
mse1 = cross_val_score(rfr, X_data_day, Y_data_day, scoring='neg_mean_squared_error', cv=5)
mse2 = cross_val_score(pr, X_data_day, Y_data_day, scoring='neg_mean_squared_error', cv=5)
mse3 = cross_val_score(elr, X_data_day, Y_data_day, scoring='neg_mean_squared_error', cv=5)
print('Day 随机森林 RMSE mean: ', np.sqrt(-np.mean(mse1)))
print('Day 多项式 RMSE mean: ', np.sqrt(-np.mean(mse2)))
print('Day 指数 RMSE mean: ', np.sqrt(-np.mean(mse3)))
得到结果为:
Minute 随机森林 RMSE mean: 36.46252356222592
Minute 多项式 RMSE mean: 25.074007378463858
Minute 指数 RMSE mean: 25.074007378463858
Day 随机森林 RMSE mean: 32.69292646894495
Day 多项式 RMSE mean: 24.603137953525472
Day 指数 RMSE mean: 24.603137953525472
痊愈病例预测
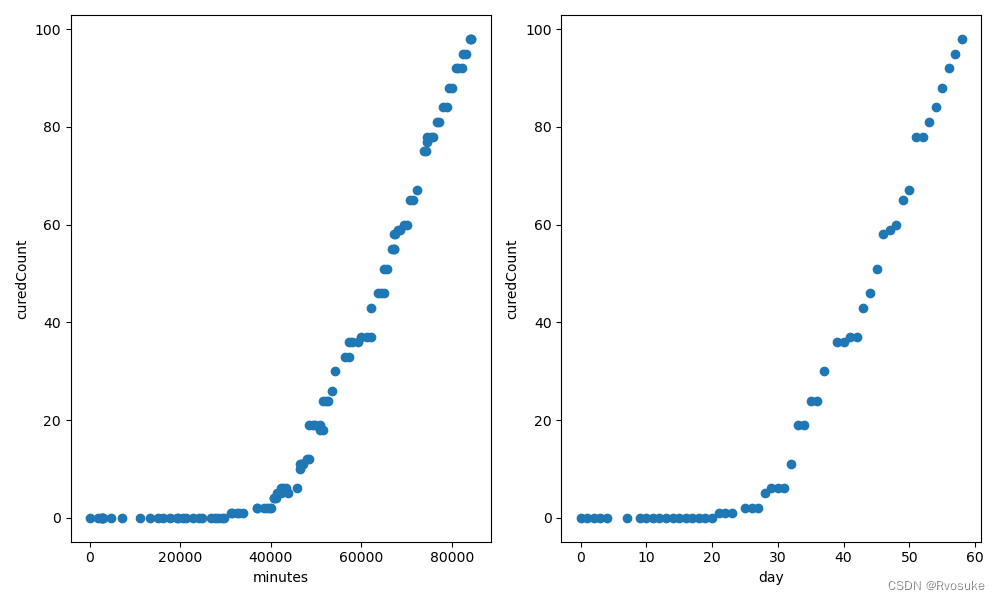
数据可视化
# 选取最后一列作为特征,‘province_curedCount’作为标签
# 分析分钟与标签的关系
plt.figure(figsize=(10, 6))
plt.subplot(1, 2, 1)
plt.scatter(df['updateTimeM'], df['province_curedCount'])
plt.xlabel('minutes')
plt.ylabel('curedCount')
# 分析天与标签的关系
# 根据 'updataTimeD' 分组,并选取每组 'province_curedCount' 的最大值
df_max = df.groupby('updataTimeD').agg({
'province_curedCount': 'max'}).reset_index()
X_data_day = df_max['updataTimeD'].values.reshape(-1, 1) # 与上次的分组不同
Y_data_day = df_max['province_curedCount'].values.reshape(-1, 1)
plt.subplot(1, 2, 2)
plt.scatter(X_data_day, Y_data_day)
plt.xlabel('day')
plt.ylabel('curedCount')
# 绘制图形
plt.tight_layout()
plt.show()
现在这幅图像很像是指数型的曲线,在第30天迎来了爆发,而爆发之后的曲线却是以平稳的速度上升,近似于直线。
所以我们需要考虑比较复杂的模型来进行拟合,比如多项式回归、指数回归、随机森林回归等。
而我们选用复杂的模型时,并不能预先判断刻度是分钟还是天好,所以我们以下的代码先利用分钟作为特征评估几种模型,选出最好的。
模型拟合
# 选取分钟作为特征,‘province_curedCount’作为标签
X_data = df['updateTimeM'].values.reshape(-1, 1)
Y_data = df['province_curedCount'].values.reshape(-1, 1)
# 选择多项式回归模型
poly = PolynomialFeatures(degree=2)
X_data_poly = poly.fit_transform(X_data)
pr = LinearRegression()
pr.fit(X_data_poly, Y_data)
plt.figure(figsize=(10, 6))
plt.scatter(X_data, Y_data, color='blue', label='data')
X_data_poly = poly.fit_transform(X_data)
Y_pred_poly = pr.predict(X_data_poly)
plt.plot(X_data, Y_pred_poly, color='green', label='polynomial linear regression')
# 选择指数回归模型
elr = LinearRegression()
elr.fit(X_data, np.log1p(Y_data))
Y_pred_exp = np.exp(elr.predict(X_data))
plt.plot(X_data, Y_pred_exp, color='red', label='exponential linear regression')
# 随机森林回归
rfr = RandomForestRegressor()
rfr.fit(X_data, Y_data)
Y_pred_rfr = rfr.predict(X_data)
plt.plot(X_data, Y_pred_rfr, color='yellow', label='random forest regression')
# 绘制图形
plt.xlabel('minutes')
plt.ylabel('curedCount')
plt.legend()
plt.show()
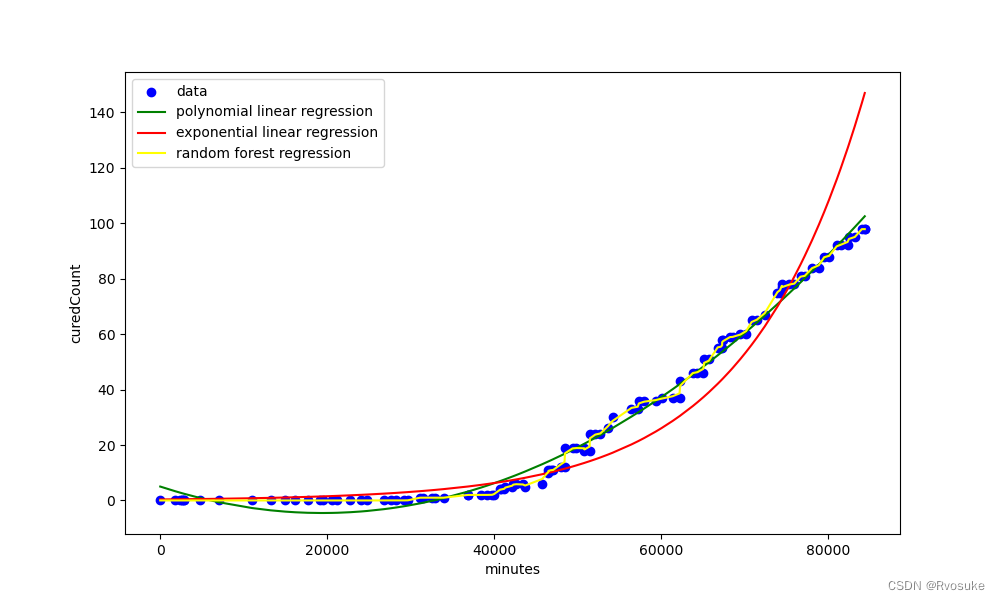
可以看出绘制出来的三条曲线还是有一定的差别的,其中指数回归在前期的拟合效果是最好的,但是在后期的拟合效果却不如多项式回归。
多项式回归在前期的拟合效果却不如指数回归,但是在后期的拟合效果却是最好的,而随机森林回归看起来像是过拟合。
通过直观地观察,如果利用交叉验证计算MSE,我们可以直接略去指数回归,它的后期偏差太大了。
我们决定保留随机森林回归与多项式回归的模型,再通过交叉验证分别评估特征为天与特征为分钟情况下模型的性能。
交叉验证评估粒度选择
# 选择随机森林回归模型计算MSE
mse1 = cross_val_score(rfr, X_data, Y_data, scoring='neg_mean_squared_error', cv=5)
mse2 = cross_val_score(pr, X_data, Y_data, scoring='neg_mean_squared_error', cv=5)
print('Minute 随机森林 MSE mean: ', -np.mean(mse1))
print('Minute 多项式 MSE mean: ', -np.mean(mse2))
mse1 = cross_val_score(rfr, X_data_day, Y_data_day, scoring='neg_mean_squared_error', cv=5)
mse2 = cross_val_score(pr, X_data_day, Y_data_day, scoring='neg_mean_squared_error', cv=5)
print('Day 随机森林 RMSE mean: ', -np.mean(mse1))
print('Day 多项式 RMSE mean: ', -np.mean(mse2))
输出结果:
Minute 随机森林 MSE mean: 168.10217015810272`
Minute 多项式 MSE mean: 926.8555128029426
Day 随机森林 RMSE mean: 175.18099999999998
Day 多项式 RMSE mean: 676.4682914327942
通过比较可以发现,当特征为分钟时,随机森林回归的MSE最小,所以我们选择随机森林回归模型,特征为分钟。
死亡人数预测
数据可视化
# 选取最后一列作为特征,‘province_deadCount’作为标签
X_data = df['updateTimeM'].values.reshape(-1, 1)
Y_data = df['province_deadCount'].values.reshape(-1, 1)
# 数据可视化
plt.figure(figsize=(10, 6))
plt.subplot(1, 2, 1)
plt.scatter(X_data, Y_data)
plt.xlabel('minutes')
plt.ylabel('deadCount')
df_max = df.groupby('updataTimeD').agg({
'province_deadCount': 'max'}).reset_index()
X_data_day = df_max['updataTimeD'].values.reshape(-1, 1)
Y_data_day = df_max['province_deadCount'].values.reshape(-1, 1)
plt.subplot(1, 2, 2)
plt.scatter(X_data_day, Y_data_day)
plt.xlabel('day')
plt.ylabel('deadCount')
plt.tight_layout()
plt.show()
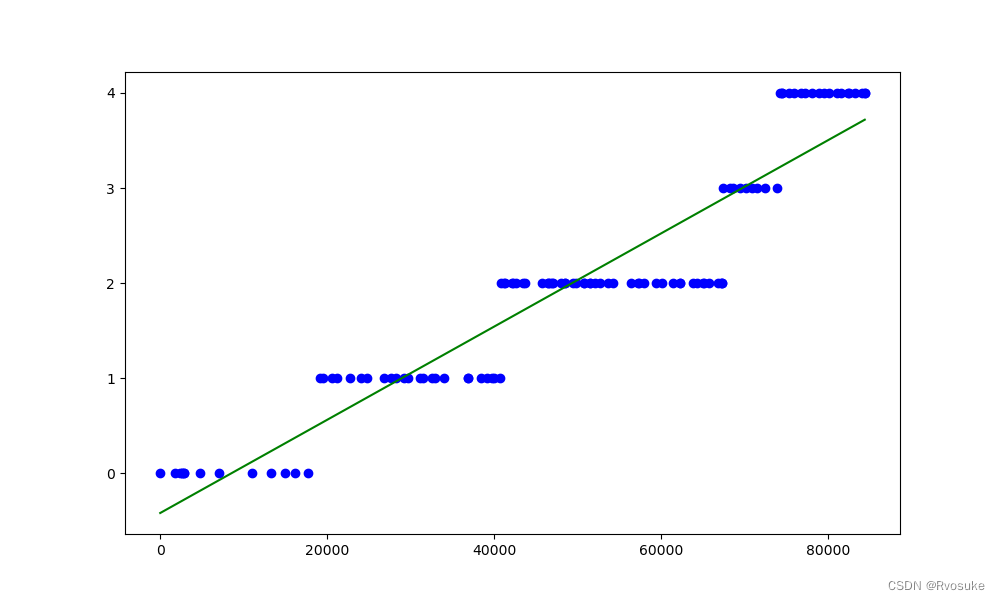
这个关系有点特殊,我暂时还没想到很好的解决办法,只能使用岭回归来调参拟合
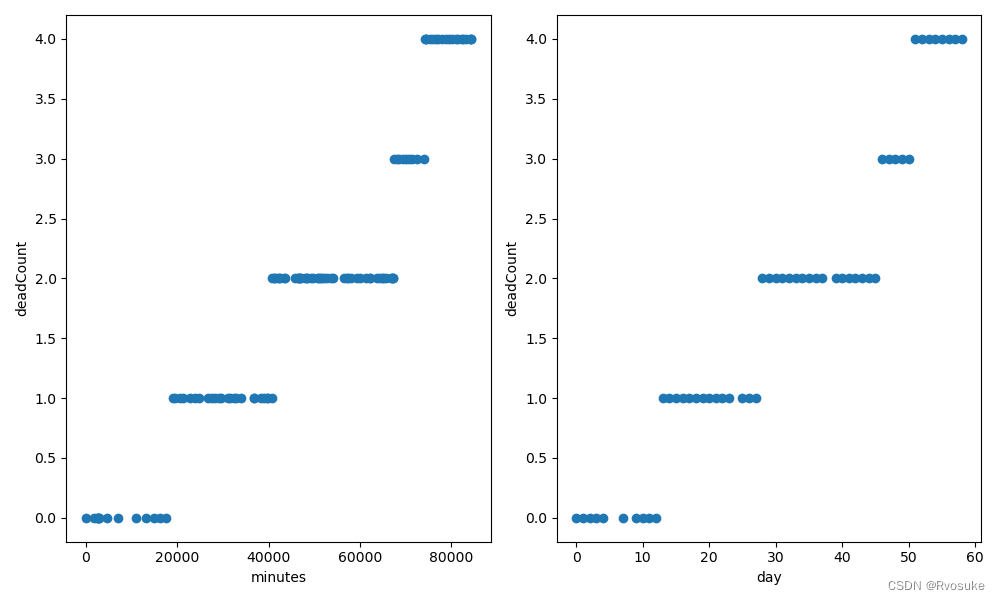
模型拟合
lr = Riger(alpha = 1000)
lr.fit(X_data, Y_data)
# 绘制拟合曲线
plt.figure(figsize=(10, 6))
plt.scatter(X_data, Y_data, color='blue', label='data')
Y_pred = lr.predict(X_data)
plt.plot(X_data, Y_pred, color='green', label='linear regression')
plt.show()
模型拟合
lr = Riger(alpha = 1000)
lr.fit(X_data, Y_data)
# 绘制拟合曲线
plt.figure(figsize=(10, 6))
plt.scatter(X_data, Y_data, color='blue', label='data')
Y_pred = lr.predict(X_data)
plt.plot(X_data, Y_pred, color='green', label='linear regression')
plt.show()