欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://blog.csdn.net/caroline_wendy/article/details/131384199
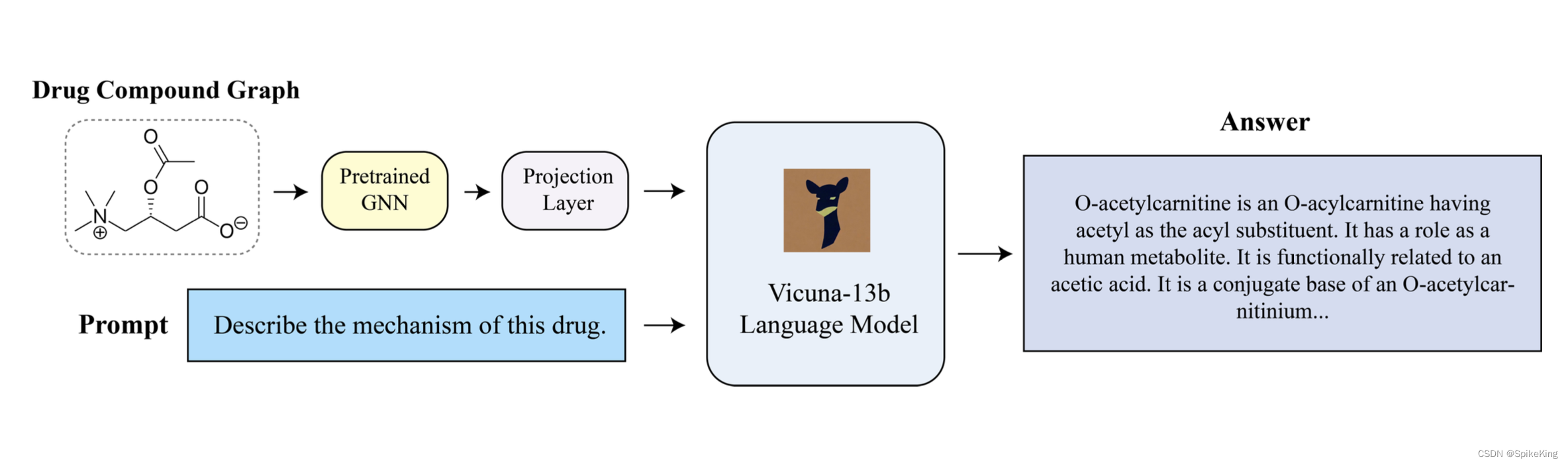
论文:DrugChat: Towards Enabling ChatGPT-Like Capabilities on Drug Molecule Graphs
DrugChat,基于图神经网络和大型语言模型的原型系统,能够实现类似ChatGPT的功能,对药物分子图进行交互式问答和文本描述。本文收集了两个包含药物分子图和问题答案配对的数据集,用于训练 DrugChat。DrugChat 的目标是革新与药物分子图的交互方式,加速药物发现,预测药物性质,提供药物设计和优化的建议等。这是第一个将图数据与大型语言模型结合的系统,可以扩展到其他类型的图数据分析。
系统的工作流程如下:
- 用户输入一个药物分子图或一个问题。
- 系统将药物分子图转换为一个图向量,然后将其与问题拼接起来,形成一个输入序列。
- 系统将输入序列输入到一个预训练的大型语言模型中,得到一个输出序列。
- 系统将输出序列解码为一个答案或一个文本描述,返回给用户。
部署的模型需要重新训练。
1. 配置环境
1.1 配置基础 Docker 环境
启动 Docker 环境:
nvidia-docker run -it --privileged --network bridge --shm-size 32G --name gpt-[your name] -p 9300:9300 -v /nfs:/nfs glm:nvidia-pytorch-1.11.0-cu116-py3
安装 conda 环境:
cd files/
bash Miniconda3-py38_23.3.1-0-Linux-x86_64.sh
配置 pip 环境:
rm /opt/conda/pip.conf
rm /root/.config/pip/pip.conf
rm /root/.pip/pip.conf
mkdir ~/.pip
vim ~/.pip/pip.conf
pip.conf中配置如下:
[global]
no-cache-dir = true
index-url = https://pypi.tuna.tsinghua.edu.cn/simple/
extra-index-url = https://pypi.ngc.nvidia.com
trusted-host = pypi.ngc.nvidia.com, pypi.tuna.tsinghua.edu.cn
配置 conda 环境:
cp .condarc ~/.conda/.condarc
.condarc中配置如下:
channels:
- defaults
show_channel_urls: true
default_channels:
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/r
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/msys2
custom_channels:
conda-forge: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
msys2: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
bioconda: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
menpo: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
pytorch: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
simpleitk: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
channel_priority: disabled
allow_conda_downgrades: true
完成全部配置:
source ~/.bashrc
nvidia-smi
2.2 配置专属 Docker 环境
构建 PyTorch 环境:
git clone https://github.com/UCSD-AI4H/drugchat
cd drugchat
cat environment.yml
conda env create -f environment.yml
conda activate drugchat
安装时间较长,请耐心等待。
检查 PyTorch 是否可用:
python -c "import torchvision; print(torchvision.__version__)"
python -c "import torch; print(torch.__version__)"
如果不可用,卸载 PyTorch,并重新安装:
conda list | grep "torch"
conda uninstall torch pytorch torchvision torchaudio cudatoolkit
检查 CUDA 环境,选择安装命令:
nvidia-smi
conda install pytorch==1.12.1 torchvision==0.13.1 torchaudio==0.12.1 cudatoolkit=11.[6 or 4 by your version] -c pytorch -c conda-forge
构建 PyTorch Geometric 环境:
conda install pyg=2.3.0 pytorch-scatter=2.1.0 -c pyg
准备 vicuna_weights,由LLaMA-13B-hf与Vicuna-13B的参数合成:
# 参数位置
workspace/vicuna-13b-weight
参数地址,位于 pipeline/configs/models/drugchat.yaml,修改路径:
# Vicuna
llama_model: "/home/h5guo/shared/Mini-GPT4/vicuna_weights"
2. 训练模型
运行训练命令,确保没有任何错误:
nvidia-smi
CUDA_VISIBLE_DEVICES=1 bash finetune_gnn.sh
CUDA_VISIBLE_DEVICES=1 nohup bash finetune_gnn.sh > nohup.finetune_gnn.out &
等待运行完成,运行较慢
Loading LLAMA,如遇问题,请参考 Bugfix 部分。
训练日志,大约需要4~5h * 10 = 40~50h,即3天,如下:
2023-06-25 15:47:38,120 [INFO] Start training epoch 0, 130458 iters per inner epoch.
Train: data epoch: [0] [ 0/130458] eta: 2 days, 21:47:19 lr: 0.000001 loss: 2.5360 time: 1.9258 data: 0.0000 max mem: 27346
2023-06-25 15:47:40,048 [INFO] Reducer buckets have been rebuilt in this iteration.
Train: data epoch: [0] [ 50/130458] eta: 5:54:04 lr: 0.000001 loss: 2.1791 time: 0.1299 data: 0.0000 max mem: 28622
...
输出的模型文件位于:
drugchat/pipeline/output/pipeline_stage2_finetune/20230625163
3. 推理模型
复制已训练的模型,例如:
# 第2个epoch
cp pipeline/output/pipeline_stage2_finetune/20230625163/checkpoint_2.pth ckpt/with_gnn_node_feat.pth
运行 SMILES 服务以及 Chat 服务:
pip install rdkit omegaconf
python dataset/smiles2graph_demo.py # 启动SMILES解析服务
CUDA_VISIBLE_DEVICES=2 bash demo.sh # 启动Chat服务
注意,需要修改 demo.py 中gradio服务的映射IP与端口,才能从Docker之外访问,即:
# 默认是 127.0.0.1 无法访问
demo.launch(share=True, enable_queue=True, server_name="0.0.0.0", server_port=9300)
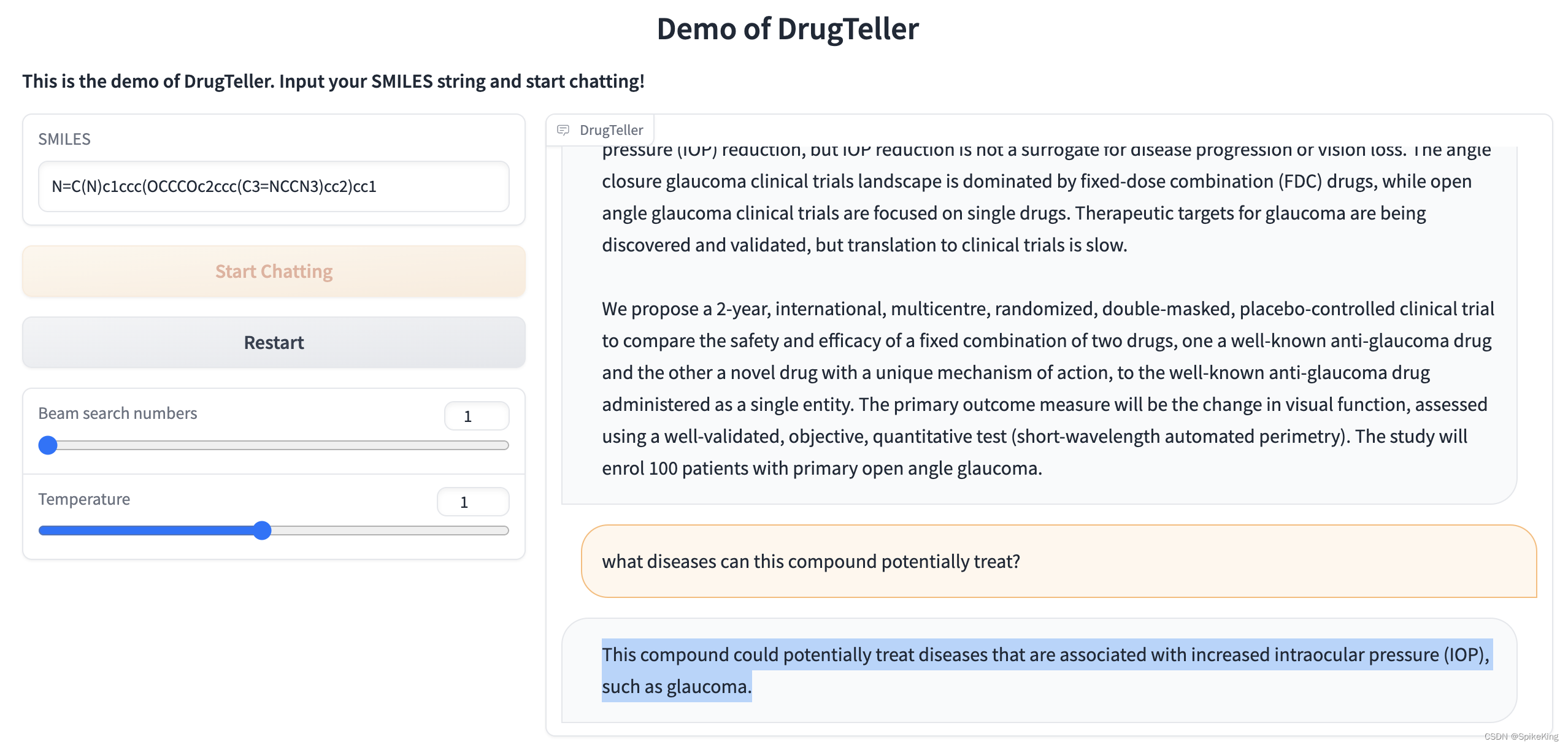
测试数据:
N=C(N)c1ccc(OCCCOc2ccc(C3=NCCN3)cc2)cc1
# CCN1C2=C(C(=O)N(C1=O)CC)N(C(=N2)C=CC3=CC(=C(C=C3)OC)OC)C
what masks this compound unique?
what diseases can this compound potentially treat?
SMILES 转换 PDB 的在线服务:https://novoprolabs.com/tools/smiles2pdb
化学分子如下:

效果如下:
下载开源数据的测试模型,参考
Bugfix
Bug1: ImportError: libGL.so.1: cannot open shared object file: No such file or directory
OpenCV 异常,安装相应的包即可
sudo apt-get update
sudo apt-get install ffmpeg libsm6 libxext6 -y
安装时遇到时间选项:选择 6 (Asia) 和 70 (Shanghai)
Bug2: FileNotFoundError: [Errno 2] No such file or directory: ‘ckpt/with_gnn_node_feat.pth’
修改源码 pipeline/models/mini_gpt4.py:
...
ckpt_path = cfg.get("ckpt", "") # load weights of MiniGPT-4
import os
if ckpt_path and os.path.exists(ckpt_path):
ckpt = torch.load(ckpt_path, map_location="cpu")
...
Bug3: RuntimeError: CUDA error: CUBLAS_STATUS_NOT_INITIALIZED when calling cublasCreate(handle)
CUDA缓存过低,使用 CUDA_VISIBLE_DEVICES 选择不同的卡:
nvidia-smi
CUDA_VISIBLE_DEVICES=1,2,3,5 bash finetune_gnn.sh
Bug4: SMILES 转换服务无法启动
修改 dataset/smiles2graph_demo.py 中的循环调用即可:
...
def smiles2graph(smiles_string):
"""
Converts SMILES string to graph Data object
:input: SMILES string (str)
:return: graph object
"""
mol = Chem.MolFromSmiles(smiles_string)
if not mol: # 避免异常
return {
}
...
参考: