一、R、Rtools下载
1.R下载
进入官网,点击Download下的CRAN或者downloadR
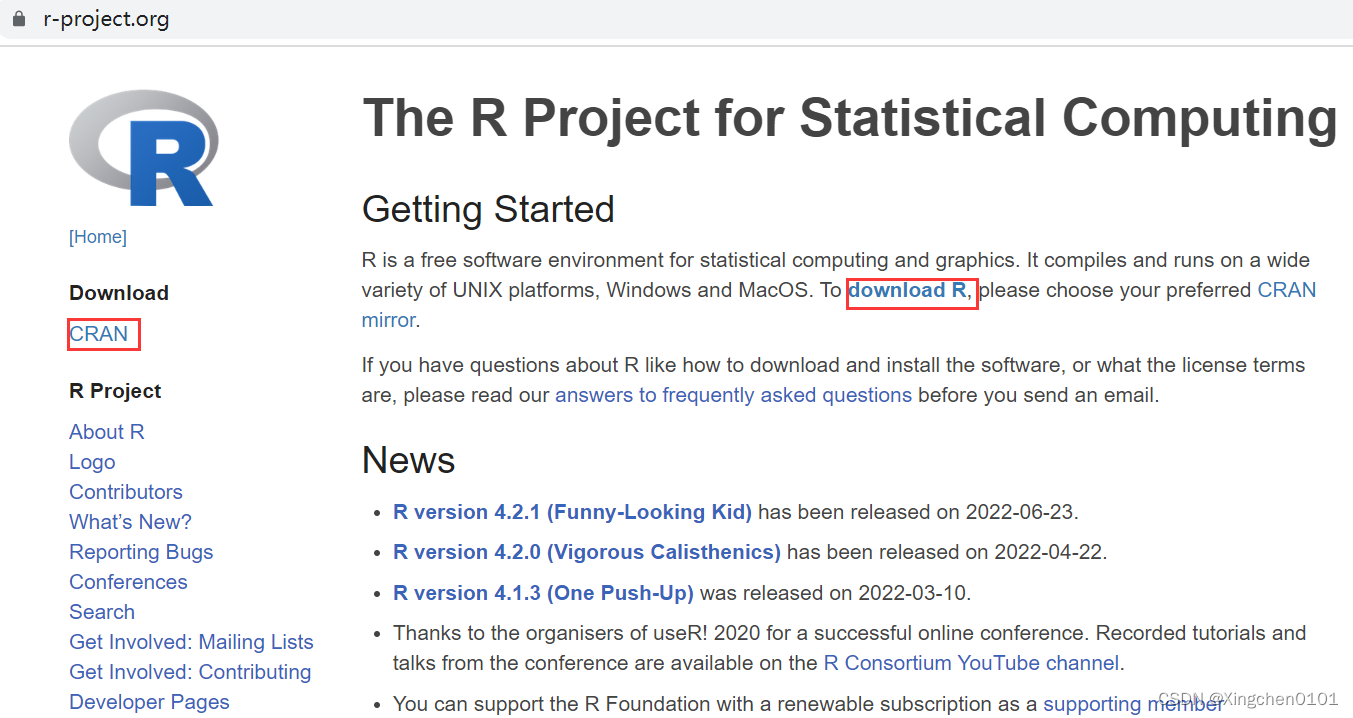
点击清华大学镜像地址
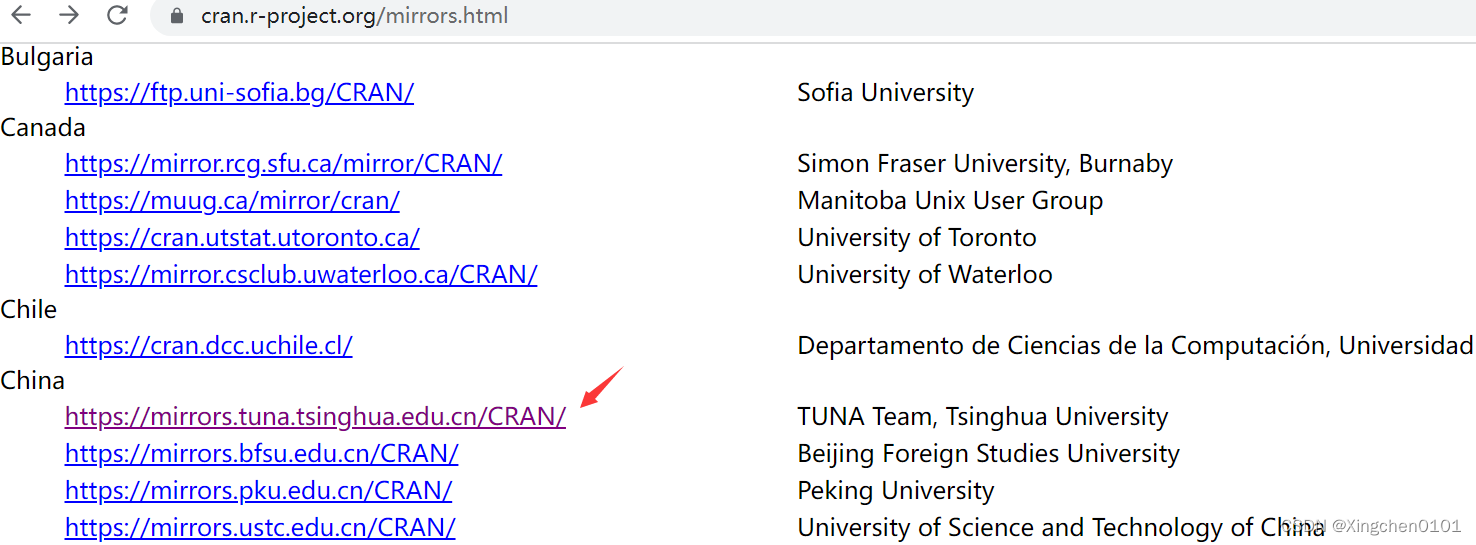
选择相应系统
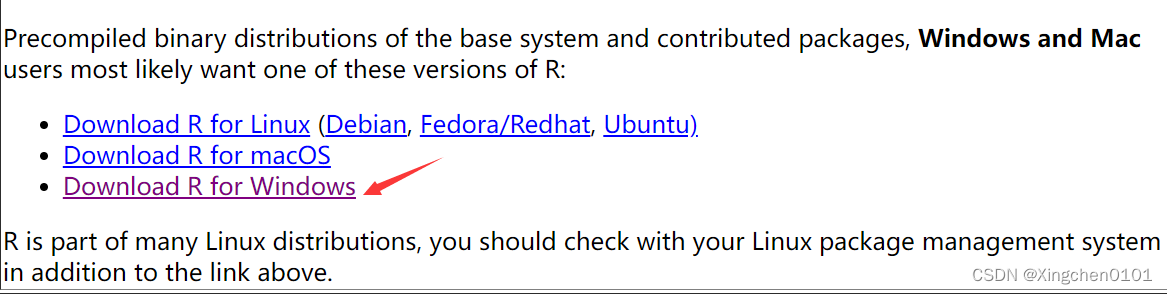
点击base和Rtools会分别进入R基本下载和Rtools工具下载页面

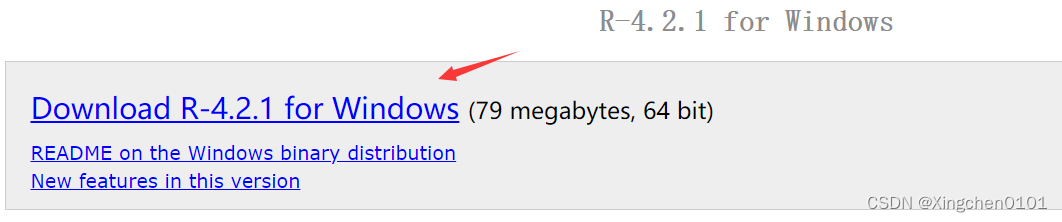
2.Rtools下载



二、Rsudio下载
点击DOWNLOAD
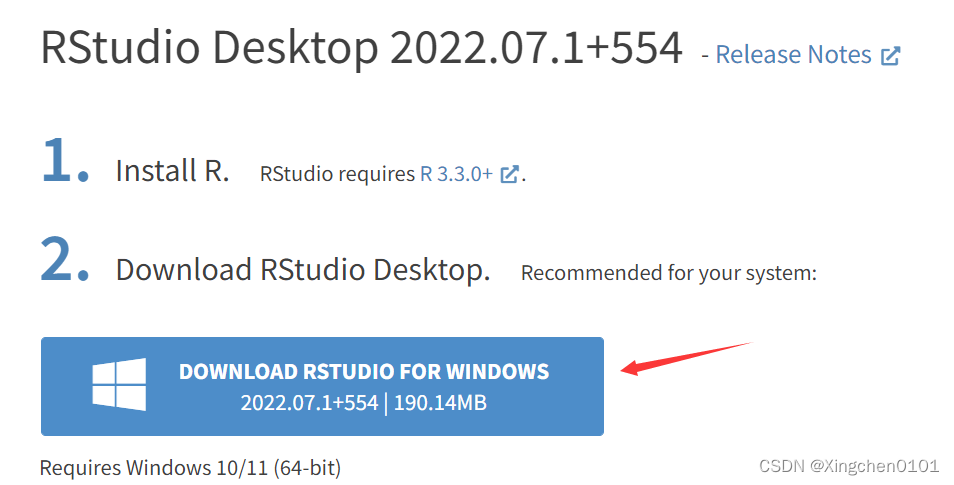
选择FREE版即可
三、配置
1.配置Rtools路径
(1)创建路径配置文件.Renviron
进入R Studio里,运行以下代码
writeLines('PATH="${RTOOLS40_HOME}\\usr\\bin;${PATH}"', con = "~/.Renviron")
没有出错就直接看步骤(2)
(2)关闭R Studio,重新打开
(3) 测试路径配置是否成功
运行 Sys.which("make") 这段代码,看输出是不是类似"C:\rtools40\usr\bin\make.exe",具体因rtools安装路径而异,只要输出不是空字符串就行,表明路径配置成功。
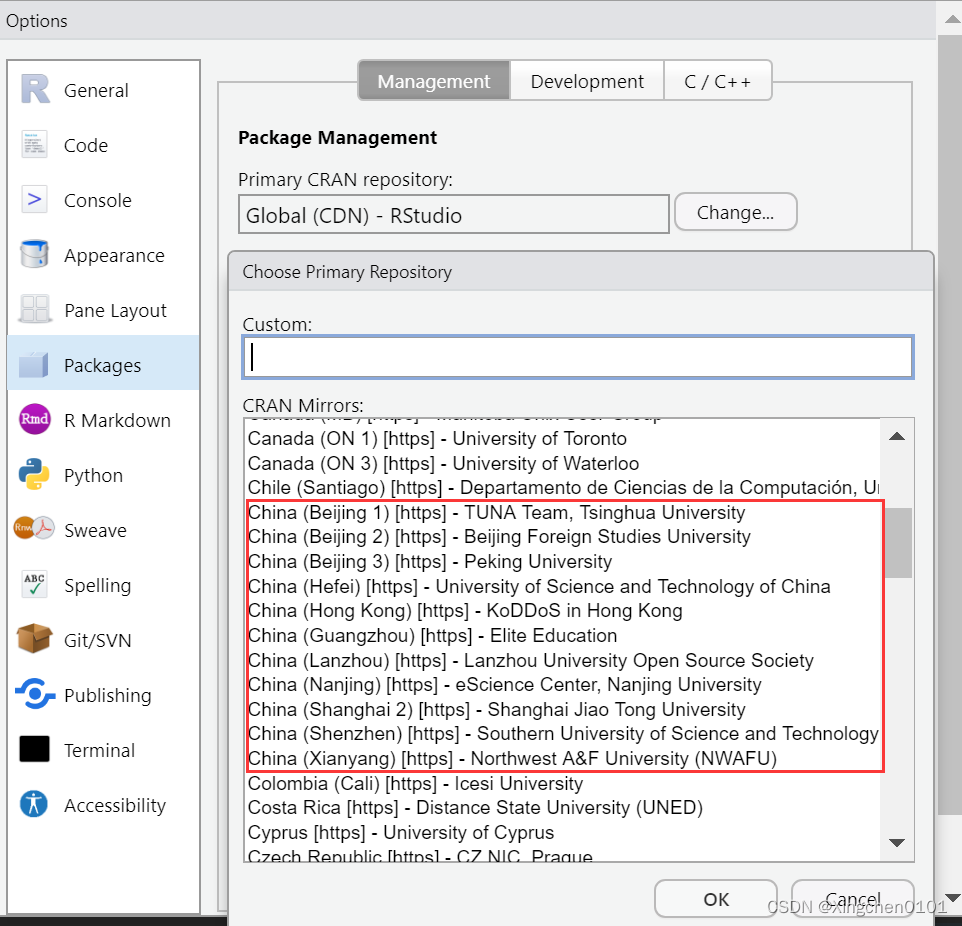
2.镜像配置
在Rstudio里点击Tools——Global Options——Packages——Change,选择一个国内的镜像即可。
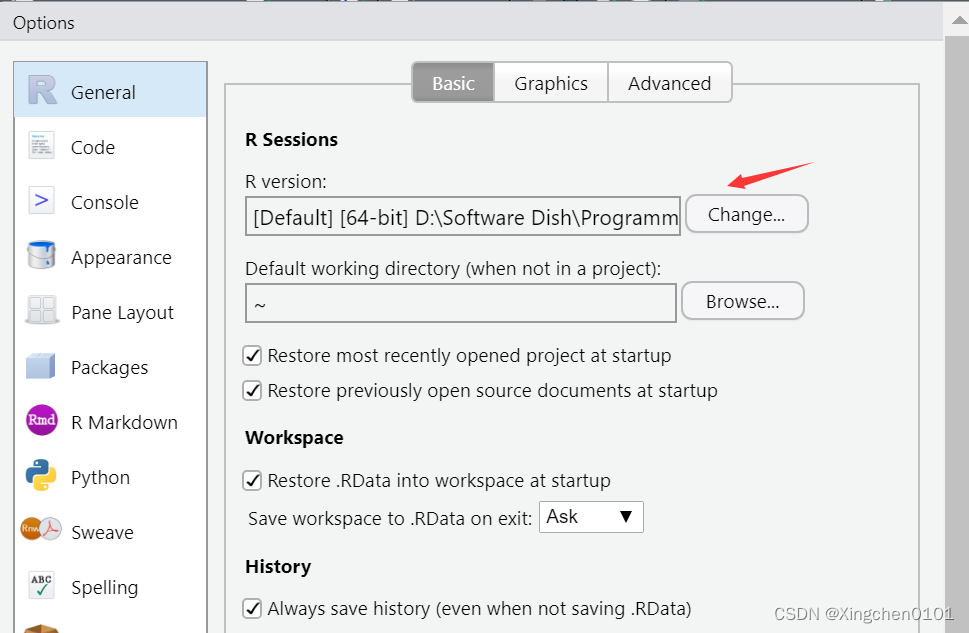
3.R版本选择
在Rstudio里点击Tools——Global Options——General——Change,选择已下载的需要使用的R版本。
四、常用R包的下载
安装R包
update.packages()
if(!require("xlsx")) install.packages("xlsx")
if(!require("tidyr")) install.packages("tidyr")
if(!require("dplyr")) install.packages("dplyr")
if(!require("ggplot2")) install.packages("ggplot2")
if(!require("data.table")) install.packages("data.table")
if(!require("ggrepel")) install.packages("ggrepel")
if(!require("devtools")) install.packages("devtools")
if(!require("BiocManager")) install.packages("BiocManager")
安装Bioconductor上的R包
BiocManager::install()
if(!require("DESeq2")) BiocManager::install("DESeq2")
if(!require("clusterProfiler")) BiocManager::install("clusterProfiler")