元素循环是生物地球化学循环的重要环节,主要涉及碳、氮、磷、硫等元素的循环过程。凌恩生物强势推出基于宏基因组的氮循环研究方案,构建了完整的氮循环循环模式图,对宏基因组数据进行深入挖掘,各部分结果图可直接用于文章发表,充分满足您对元素循环的各类个性化分析要求!

氮是所有生物的必要组成成分,同时也是限制地球上所有生物生长的主要营养素。目前来讲,人类可以自由地获取氮气作为氮源,它存在于大气中。然而,大多数生物依赖于生物可利用的氮形式,例如铵盐和硝酸盐,以满足其生长需求。这些氮形式的利用性取决于不同的氮转化反应。氮循环指的是氮元素在地球大气圈、生物圈、土壤圈和水圈之间的迁移、转化和周转循环过程。
全球最大的可自由获取氮储量是氮气(N2),其次是有机氮、硝酸盐(NO3-)和一氧化二氮(N2O)。通常,生物地球化学中的氮循环可归纳为六种不同的氮转化过程,包括同化、氨化、硝化、反硝化、厌氧氨氧化(anammox)和固氮。这些氮转化过程之间的通量差异很大,其中氨和有机氮的相互转化是氮通量最大的,另一个重要的氮通量是硝化作用。
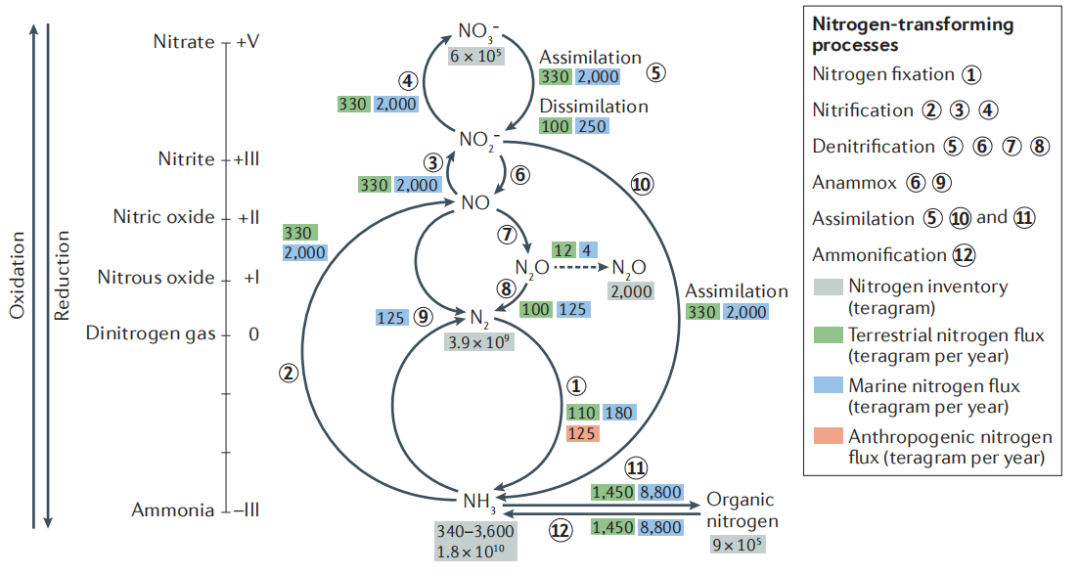
图 全球氮转化过程、氮储存及氮通量[1]。
图片来源:www.nature.com/nrmicro
目前,对于环境中氮元素循环挖掘主要是基于功能基因扩增、宏基因组数据分析等。
凌恩生物全新升级宏基因组氮循环分析方案:
功能基因扩增
1、引物特异性,覆盖不够全面;
2、只能获得部分元素循环相关基因。
宏基因组分析
1、对于元素循环代谢通路不够全面,且循环路径分类不够明确;
2、不能满足目前高水平科研论文研究需求
凌恩生物新流程
1、基于宏基因组标准分析得到的KEGG注释结果;
2、根据文献整合得到的碳、氮、硫和磷循环路径及详细基因丰度计算方式;
3、计算每个样本中碳、氮、硫和磷循环各路径的基因丰度,进而进行一系列样本间比较。
凌恩生物全新升级宏基因组氮循环方案,首先基于宏基因组标准分析得到的KEGG注释结果,通过查阅大量基于宏基因组测序分析环境微生物群落元素循环的相关文件,建立了完整的氮循环循环模式图。
通过建立完整的氮循环过程模式图,确定不同代谢过程所涉及的功能基因及其丰度计算方法,基于宏基因组测序数据的基因注释及丰度定量结果,对环境样品微生物群落氮循环能力进行分析及比较。构建了包含51个关键基因,涵盖7个N循环子路径的完整氮循环分析方案,基于该方案可对识别到的相关功能基因进行物种分类学注释,识别环境样本中参与氮循环的主要功能微生物种属,并快速准确地分析氮循环代谢的微生物群落的结构和功能,为研究微生物驱动的氮循环及其与其他元素的耦合机制提供保障。并可综合分析不同生境下氮元素循环的整体水平及其影响因素,为环境健康和影响评价提供基因水平上的有力证据。
氮循环路径
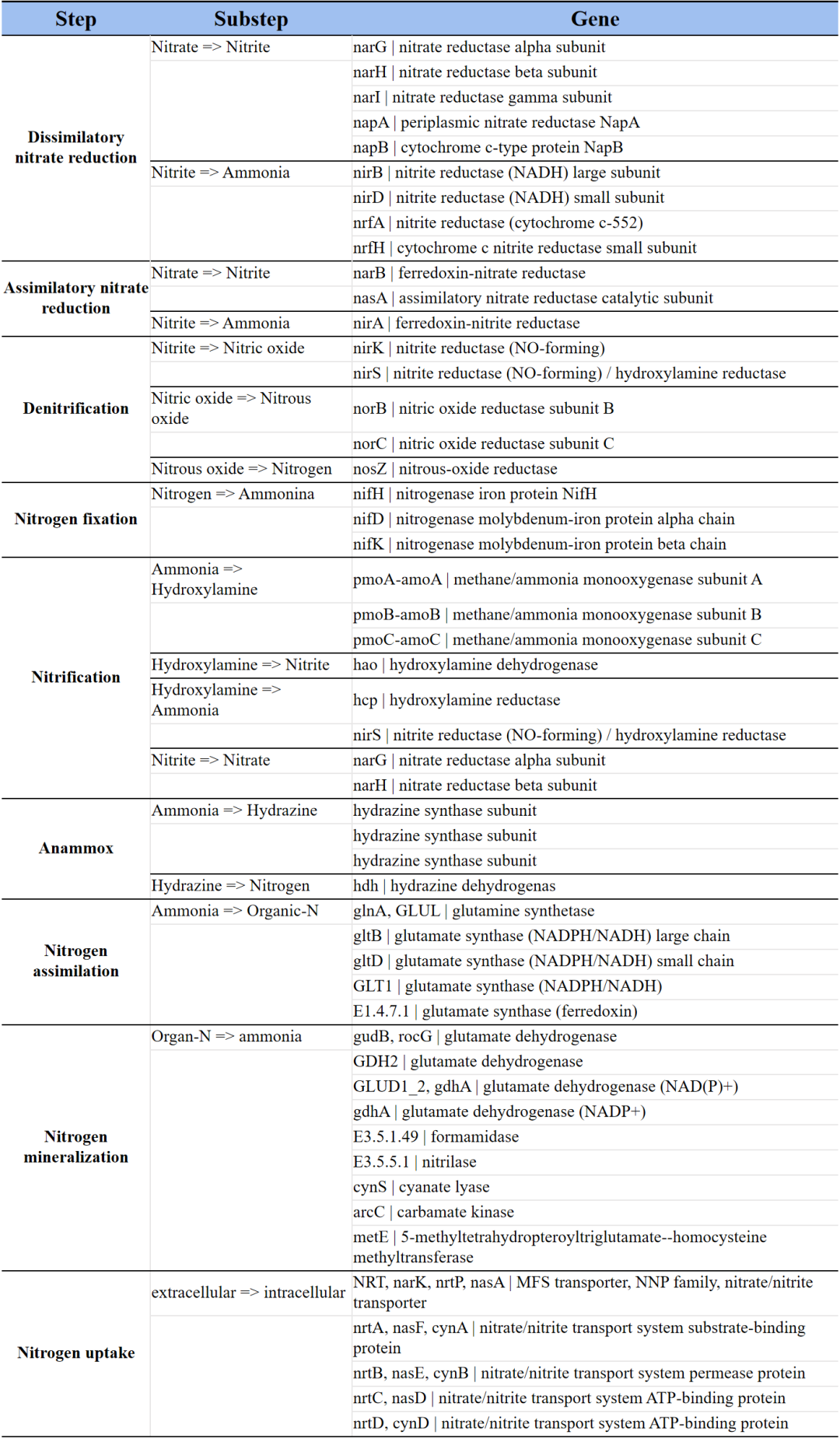
新升级的宏基因组氮循环
分析内容包含四大分析模块:
氮循环功能基因丰度定量及比较分析
氮循环功能基因路径图
样本间/组间Beta多样性比较分析
宿主微生物识别及与功能基因的联合分析
新升级的宏基因组氮循环分析对结果的展示进行了细致的优化,在保证分析结果清晰展示的前提下,对分析结果图的各项细节参数进行了优化调整,分析结果无需任何调整即可直接达到文章发表要求。
凌恩生物氮循环分析结果展示图
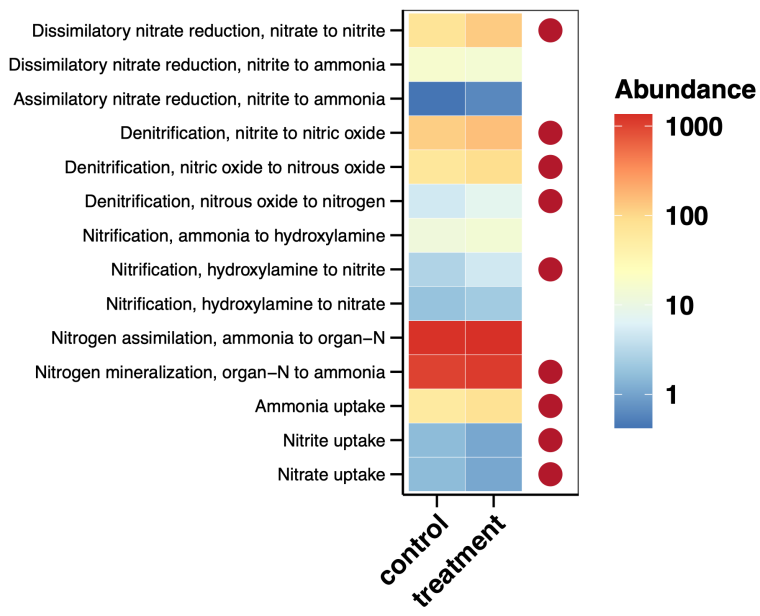

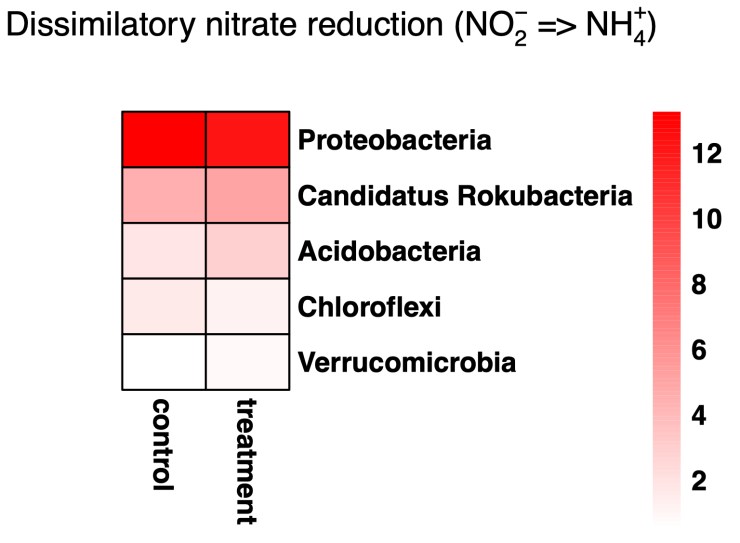
氮循环功能基因丰度热图
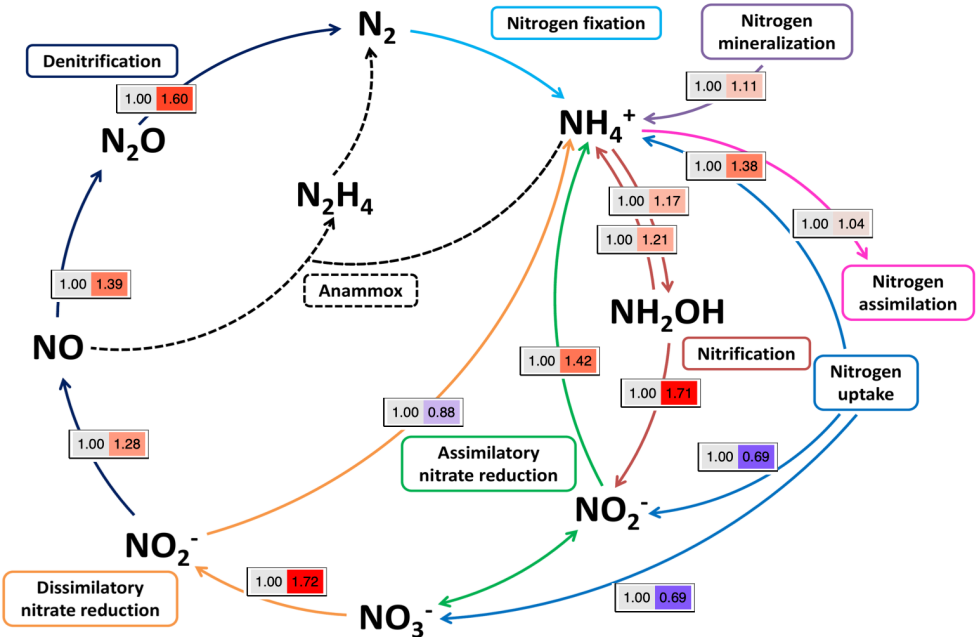
氮循环功能基因路径循环图
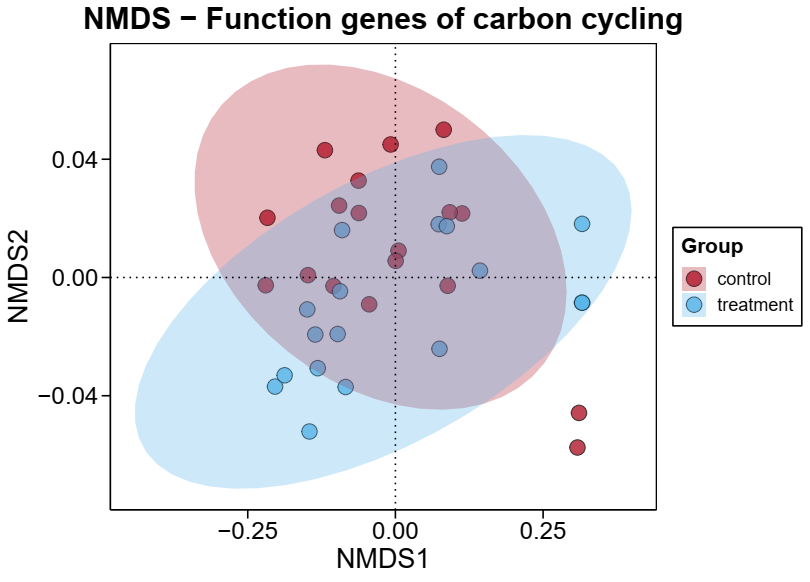
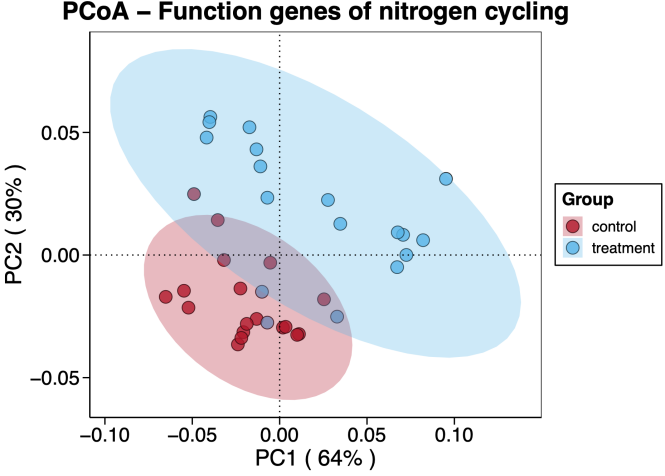
样本间/组间比较分析
宿主微生物组识别(子路径)
经典文献案例

题目:深海冷泉沉积物中存在系统进化和催化能力多样的固氮微生物
期刊:Nature communications
影响因子:17.694
DOI:10.1038/s41467-022-32503-w
在以碳为主的冷泉环境中,微生物介导的氮循环仍然知之甚少。研究利用宏基因组和宏转录组数据对冷泉生态系统中的生物固氮过程进行研究,报道了冷泉固氮菌度对全球氮平衡的重大贡献。
研究团队分析了61个宏基因组、1428个宏基因组组装基因组和6个宏转录组,这些数据来自于全球11个不同的冷泉位点。研究结果表明,冷泉沉积物含有系统发育多样的固氮酶基因,对应于重氮营养谱系的扩展多样性;冷泉中不同固氮菌群之间的固氮基因被推断为具有遗传流动性并受到纯化选择。研究将冷泉固氮菌的能力扩展到五个候选门(Altarchaeia、Omnitrophota、FCPU426、Caldatribacteriota和UBA6262),并表明冷泉固氮菌可能对全球氮平衡做出重大贡献。
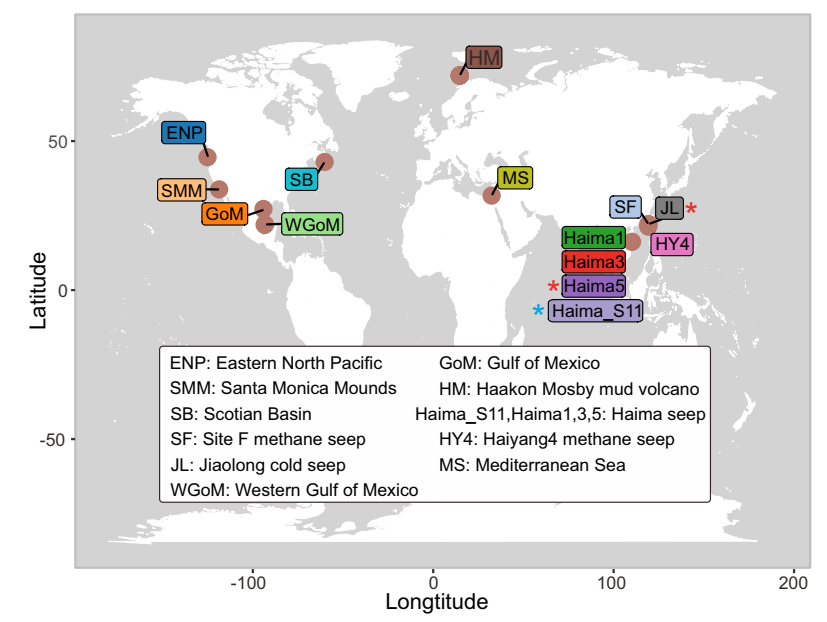
图1 收集宏基因组和宏转录组数据的11个冷泉地点的地理分布
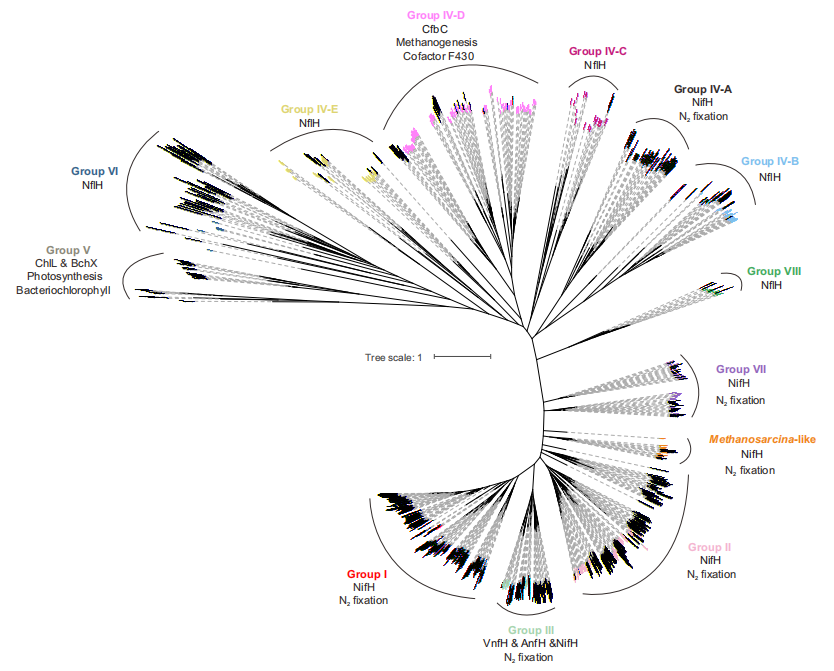
图2 从冷泉宏基因中鉴定的非冗余氮酶亚基NifH的系统发育进化树
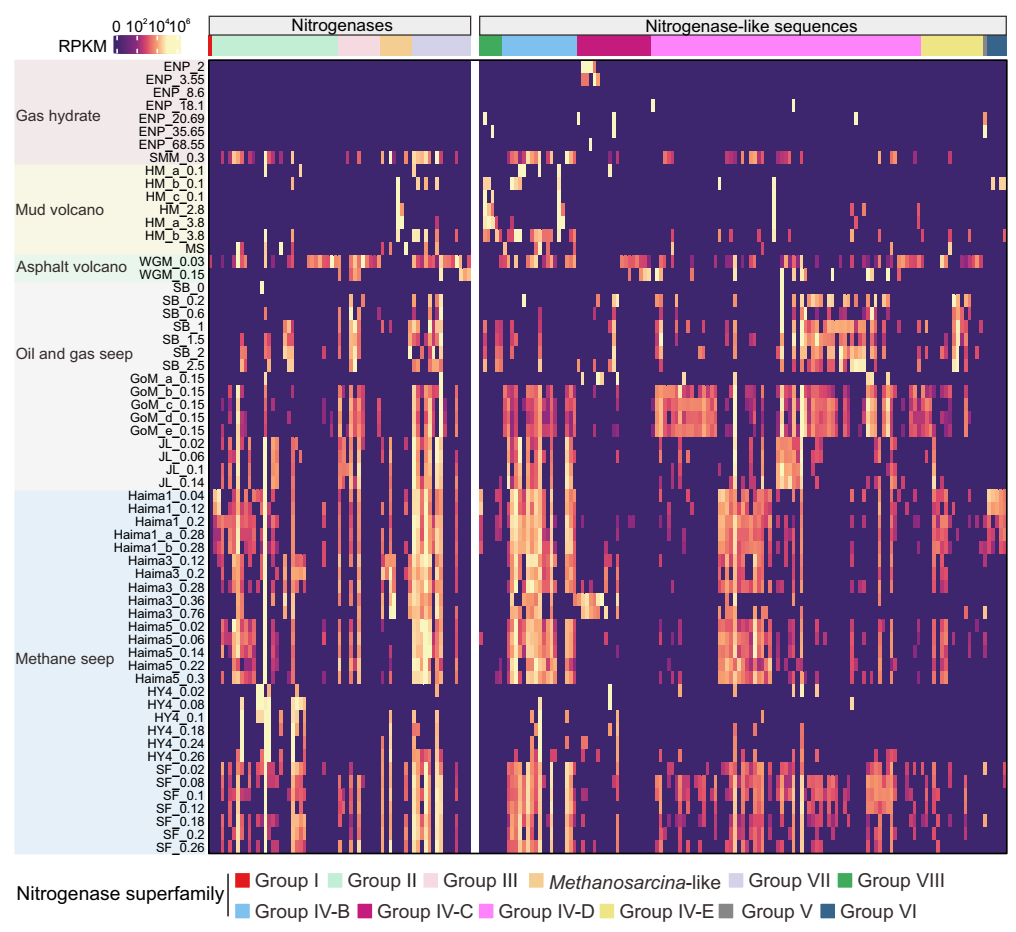
图3 来自不同沉积物的202个nifH基因的相对丰度热图
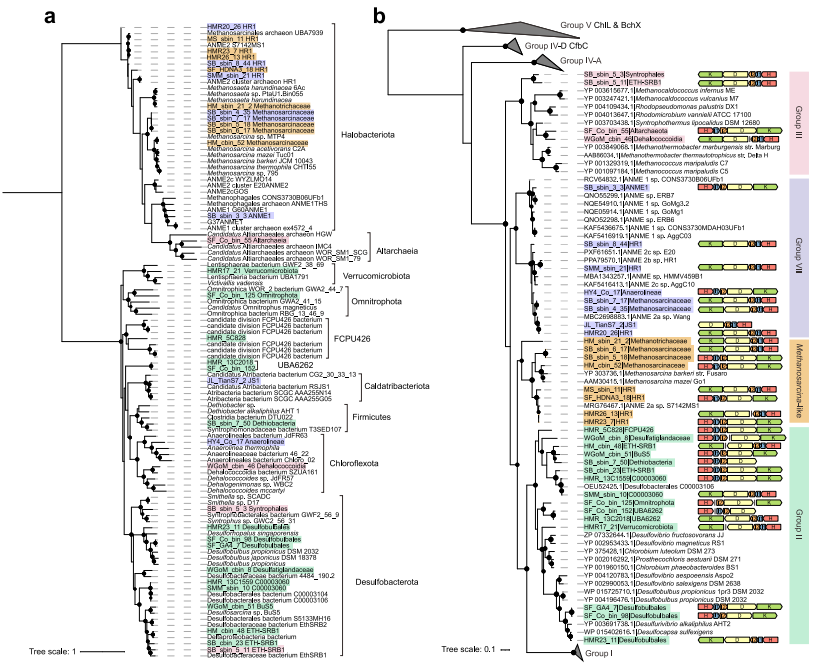
图4 具有固氮基因的相同35个MAG中鉴定的NifH蛋白序列和相应nif基因的基因组背景的系统发育分析
参考文献
[1] The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018.
[2] Phylogenetically and catabolically diverse diazotrophs reside in deep-sea cold seep sediments. Nature communications, 2022.