ENZYMES数据集是在GNN领域是非常经典的数据集,它是一个根据生物分子蛋白质结构而构建的Graph数据集合,总共600个图,也就是对应600个样本(蛋白质分子) ,共有六种结构
这个数据集是一个用于图级分类任务的数据,该数据集中共有 600张图,含有 19580个节点, 174564条边,每个节点的 特征维度为3。
1、读取ENZYMES数据集
在PyG中的 TUDataset 这个包可以导入很多图数据集,如果是第一次导入,需要进行下载,如果是因为网络问题,可以先从其它网站下载到本地,然后再使用该函数进行读取
from torch_geometric.datasets import TUDataset
dataset = TUDataset(root='./data/ENZYMES/', name='ENZYMES')
2、获取数据
data = dataset.data
print(data)
>>>Data(x=[19580, 3], edge_index=[2, 74564], y=[600])
从该结果可以看出这个数据集的节点特征矩阵的维度为【19580,3】,代表这个数据集(600张图一共)有19580个节点,每个节点的特征维度为3,共有74564条边,这些数据并不是一个图的,而是ENZYMES600张图加起来一共的。
3、获取第一张图的属性
如果我们需要获取第一张图,可以通过索引的方式,代码如下:
graph1 = dataset[0]
print(graph1)
>>>Data(edge_index=[2, 168], x=[37, 3], y=[1])
该结果可以看出第一张图,也就是第一个样本共有37个节点,168条边,对应一个类别
5、获取ENZYMES常见属性信息
print(dataset.num_classes)
>>>6
print(dataset.num_edge_attributes)
>>>0
print(dataset.num_edge_features)
>>>0
print(dataset.num_edge_labels)
>>>0
print(dataset.num_features)
>>>3
print(dataset.num_node_attributes)
>>>18
print(dataset.num_node_features)
>>>3
print(dataset.num_node_labels)
>>>3
6、可视化ENZYMES图数据
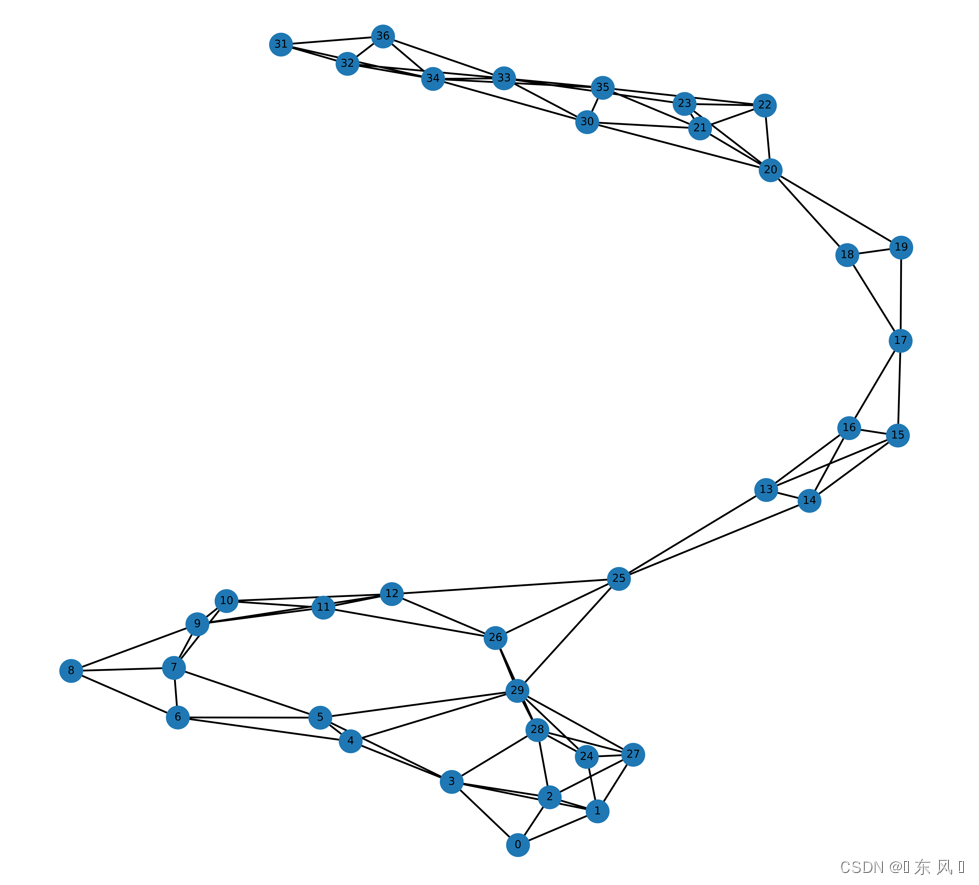
# 可视化图结构
graph = nx.Graph() # 创建一个空图
edge_index = dataset[0].edge_index # 获取第一张图
source = edge_index[0] # 起点
target = edge_index[1] # 终点
for src, tge in zip(source, target):
graph.add_edge(src.item(), tge.item())
plt.rcParams['figure.dpi'] = 300 # 设置分辨率
fig , ax1 = plt.subplots(figsize=(10,10))
nx.draw_networkx(G=graph , ax=ax1 , font_size=6 , node_size=150)
plt.show()