点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
127篇原创内容
公众号
桓峰基因公众号推出基于R语言绘图教程并配有视频在线教程,目前整理出来的教程目录如下:
FigDraw 1. SCI 文章的灵魂 之 简约优雅的图表配色
FigDraw 2. SCI 文章绘图必备 R 语言基础
FigDraw 3. SCI 文章绘图必备 R 数据转换
FigDraw 4. SCI 文章绘图之散点图 (Scatter)
FigDraw 5. SCI 文章绘图之柱状图 (Barplot)
FigDraw 6. SCI 文章绘图之箱线图 (Boxplot)
FigDraw 7. SCI 文章绘图之折线图 (Lineplot)
FigDraw 8. SCI 文章绘图之饼图 (Pieplot)
FigDraw 9. SCI 文章绘图之韦恩图 (Vennplot)
FigDraw 10. SCI 文章绘图之直方图 (HistogramPlot)
FigDraw 11. SCI 文章绘图之小提琴图 (ViolinPlot)
FigDraw 12. SCI 文章绘图之相关性矩阵图(Correlation Matrix)
FigDraw 13. SCI 文章绘图之桑葚图及文章复现(Sankey)
前言
桑基图(Sankey diagram),即桑基能量分流图,也叫桑基能量平衡图。它是一种特定类型的流程图,图中延伸的分支的宽度对应数据流量的大小,比较适用于用户流量等数据的可视化分析。因1898年Matthew Henry Phineas Riall Sankey绘制的“蒸汽机的能源效率图”而闻名,此后便以其名字命名为“桑基图”。
https://gestaltenlernen.ch/infografik/formen-von-diagramme/6-sankey-diagramm/
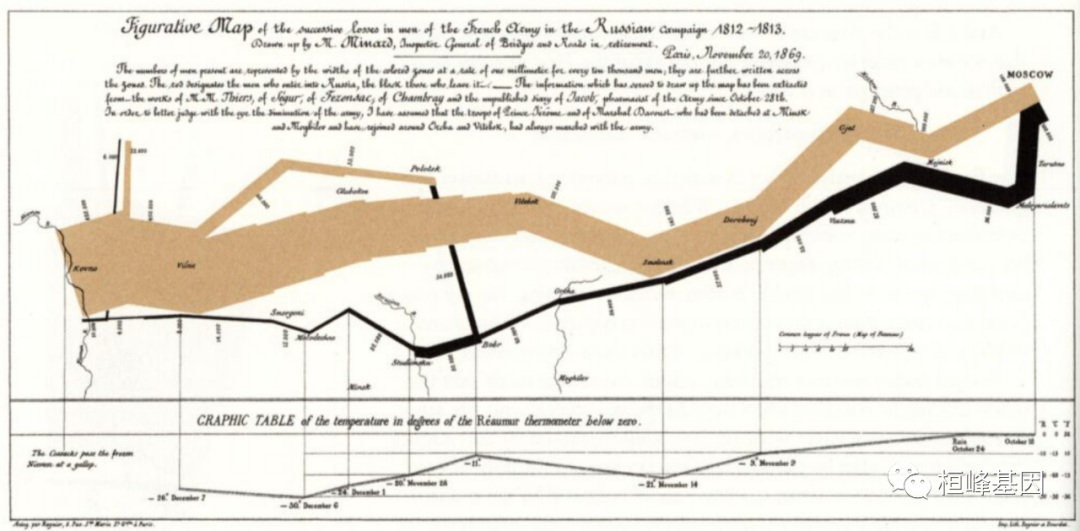
桑基图最明显的特征就是,始末端的分支宽度总和相等,即所有主支宽度的总和应与所有分出去的分支宽度的总和相等,保持能量的平衡。

现如今的可视化软件行业如此发达,制作此类桑基图已绝非难事,从最高端的JS库(D3、Ecgarts、highlight)到主流的数据科学编程工具(R、Python等)亦或者人人都能上手的自助式BI工具(PowerBI、Tableau等)都可以胜任此项工作。
软件安装
本文将着重分享使用R语言中的ggalluvial、networkD3和 easyalluvial 工具包可视化来制作桑基图的大致过程。
if (!require(ggalluvial)) install.packages("ggalluvial")
if (!require(networkD3)) install.packages("networkD3")
if (!require(riverplot)) install.packages("riverplot")
if (!require(LaCroixColoR)) devtools::install_github("johannesbjork/LaCroixColoR")
if (!require(easyalluvial)) devtools::install_github("erblast/easyalluvial")
if (!require(parcats)) devtools::install_github("erblast/parcats")
数据读取
桑基图的数据结构很简单,只有三列数据信息:
-
起点:
-
终点:
-
权重:
虽然只有三列数据,但是桑基图可以做出多级节点,在数据整合上,我们需要事前现将所有节点对应的起点、终点和权重值都顺序的纵向合并为三列字段。
例子实操
1. geom_alluvium {ggalluvial}
library(ggalluvial)
library(tidyverse)
library(ggtext)
library(hrbrthemes)
library(wesanderson)
library(LaCroixColoR)
head(as.data.frame(UCBAdmissions))
## Admit Gender Dept Freq
## 1 Admitted Male A 512
## 2 Rejected Male A 313
## 3 Admitted Female A 89
## 4 Rejected Female A 19
## 5 Admitted Male B 353
## 6 Rejected Male B 207
is_alluvia_form(as.data.frame(UCBAdmissions), axes = 1:3, silent = TRUE)
## [1] TRUE
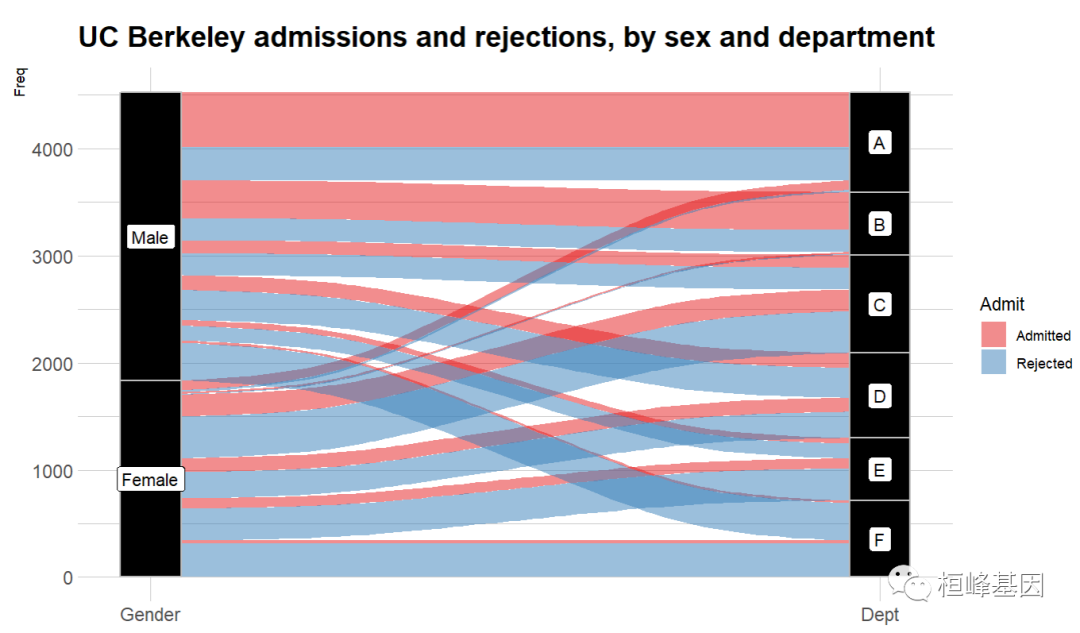
”宽数据“可视化绘制
ggalluvial可为两种数据绘制桑基图,其中一种就是宽数据,可以看到,这就是宽数据了,通常在使用ggplot2绘图,我们都需要将其转换成长数据绘图,这里使用官网提供的例子来看一下宽数据绘图过程:
ggplot(as.data.frame(UCBAdmissions), aes(y = Freq, axis1 = Gender, axis2 = Dept)) +
geom_alluvium(aes(fill = Admit), width = 1/12) + geom_stratum(width = 1/12, fill = "black",
color = "grey") + geom_label(stat = "stratum", aes(label = after_stat(stratum))) +
scale_x_discrete(limits = c("Gender", "Dept"), expand = c(0.05, 0.05)) + scale_fill_brewer(type = "qual",
palette = "Set1") + hrbrthemes::theme_ipsum(base_family = "Roboto Condensed") +
ggtitle("UC Berkeley admissions and rejections, by sex and department")
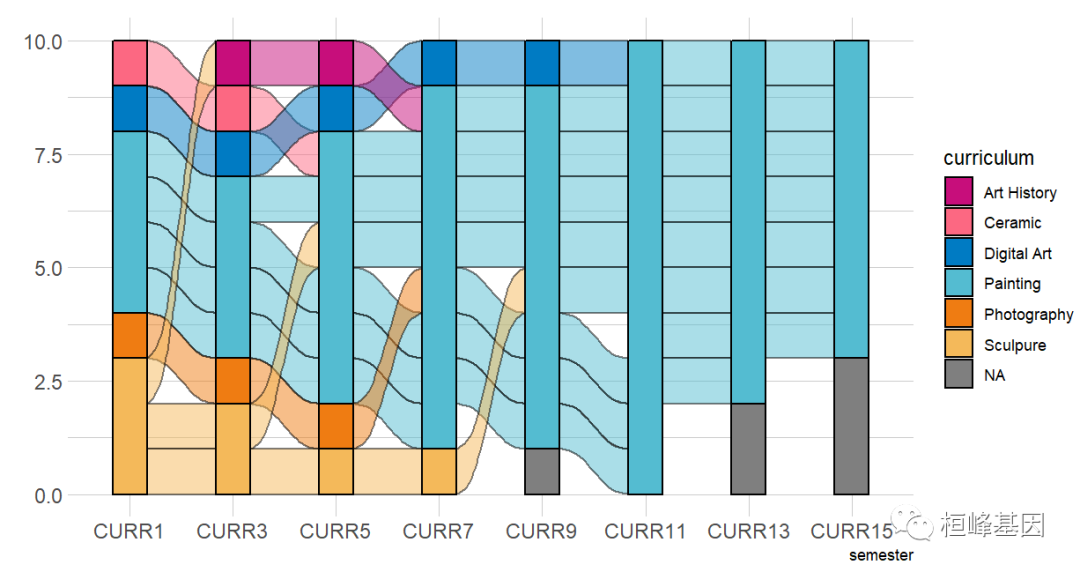
”长数据“可视化绘制
宽数据绘制虽然比较容易理解,但对其定制化修改则有些麻烦,这时候我们可以使用 长数据 进行绘制。接下来的绘图则与通常的ggplot2绘制相似,如下:
library(ggalluvial)
library(ggplot2)
data(majors)
head(majors)
## student semester curriculum
## 1 1 CURR1 Painting
## 2 2 CURR1 Painting
## 3 6 CURR1 Sculpure
## 4 8 CURR1 Painting
## 5 9 CURR1 Sculpure
## 6 10 CURR1 Painting
majors$curriculum <- as.factor(majors$curriculum)
ggplot(majors, aes(x = semester, stratum = curriculum, alluvium = student, fill = curriculum,
label = curriculum)) + scale_fill_manual(values = lacroix_palette(type = "paired")) +
ggalluvial::geom_flow(stat = "alluvium", lode.guidance = "frontback", color = "black") +
geom_stratum() + hrbrthemes::theme_ipsum(base_family = "Roboto Condensed")
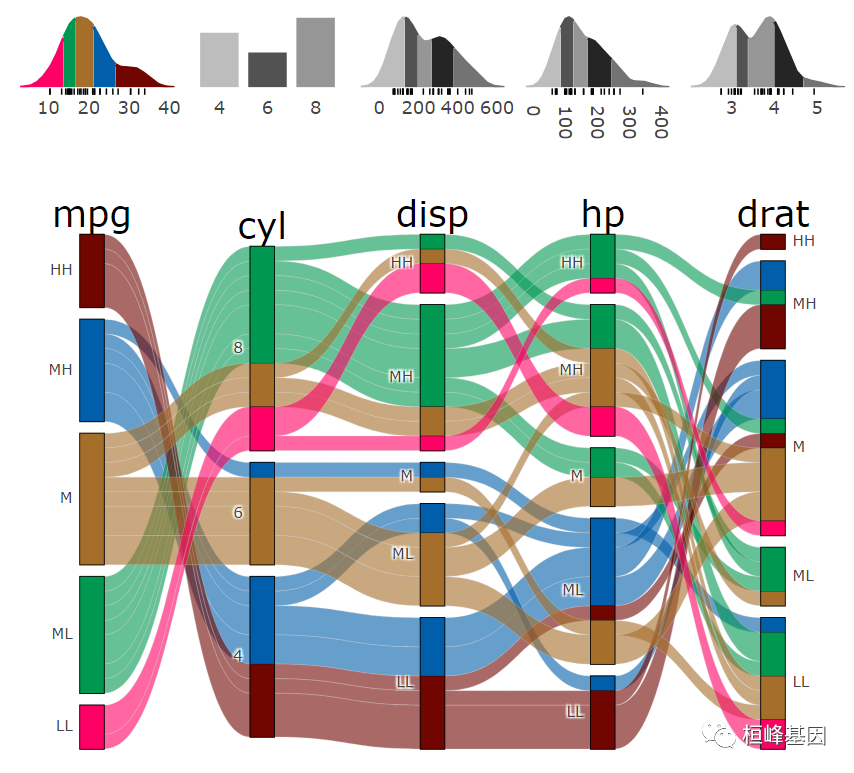
2. alluvial_wide {easyalluvial}
Description plots a dataframe as an alluvial plot. All numerical variables are scaled, centered and YeoJohnson transformed before binning. Plots all variables in the sequence as they appear in the dataframe until maximum number of values is reached.
suppressPackageStartupMessages(require(tidyverse))
suppressPackageStartupMessages(require(parcats))
suppressPackageStartupMessages(require(easyalluvial))
head(mtcars2)
## # A tibble: 6 x 12
## mpg cyl disp hp drat wt qsec vs am gear carb ids
## <dbl> <ord> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <fct> <ord> <ord> <chr>
## 1 21 6 160 110 3.9 2.62 16.5 V manual 4 4 Mazda R~
## 2 21 6 160 110 3.9 2.88 17.0 V manual 4 4 Mazda R~
## 3 22.8 4 108 93 3.85 2.32 18.6 S manual 4 1 Datsun ~
## 4 21.4 6 258 110 3.08 3.22 19.4 S automatic 3 1 Hornet ~
## 5 18.7 8 360 175 3.15 3.44 17.0 V automatic 3 2 Hornet ~
## 6 18.1 6 225 105 2.76 3.46 20.2 S automatic 3 1 Valiant
p = alluvial_wide(mtcars2, max_variables = 5)
parcats(p, marginal_histograms = TRUE, data_input = mtcars2)
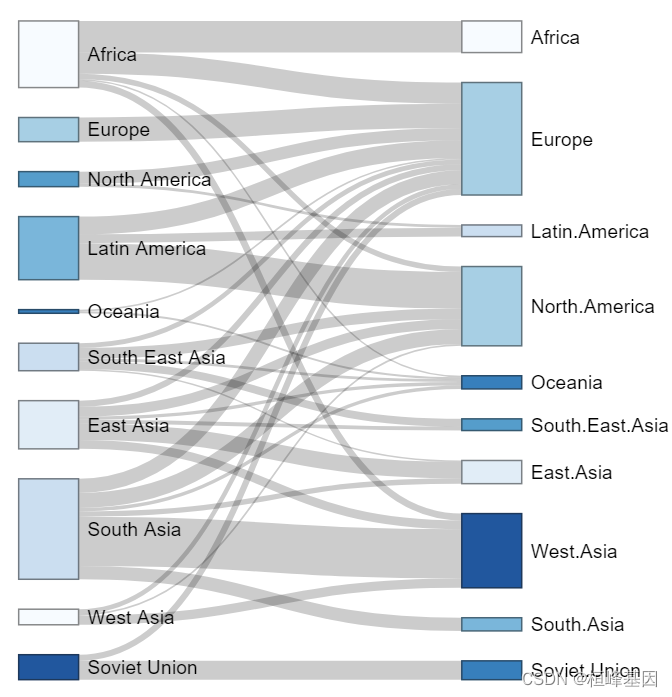
3. sankeyNetwork {networkD3}
可交互式的桑葚图,做法也同也非常简单。
Create a D3 JavaScript Sankey diagram Description Create a D3 JavaScript Sankey diagram
library(tidyverse)
library(viridis)
library(patchwork)
library(networkD3)
data <- read.table("./Example_dataset/13_AdjacencyDirectedWeighted.csv", header=TRUE)
data_long <- data %>%
rownames_to_column %>% #将行名变为普通的列
gather(key = 'key', value = 'value', -rowname) %>% #矩阵转化为作图需要的格式
filter(value > 0) #筛选value即移民数大于0的
colnames(data_long) <- c("source", "target", "value")#改变列名
data_long$target <- paste(data_long$target, " ", sep="")#加上空格以便于区分target和source
nodes <- data.frame(name=c(as.character(data_long$source), as.character(data_long$target)) %>% unique())#制作nodes
data_long$IDsource=match(data_long$source, nodes$name)-1
data_long$IDtarget=match(data_long$target, nodes$name)-1
ColourScal='d3.scaleOrdinal() .range(["#F7FBFF", "#DEEBF7", "#C6DBEF", "#9ECAE1" ,"#6BAED6", "#4292C6" ,"#2171B5" ,"#084594"])'
sankeyNetwork(Links = data_long, Nodes = nodes,
Source = "IDsource", Target = "IDtarget",
Value = "value", NodeID = "name",
sinksRight=FALSE, colourScale=ColourScal, nodeWidth=40, fontSize=13, nodePadding=20)
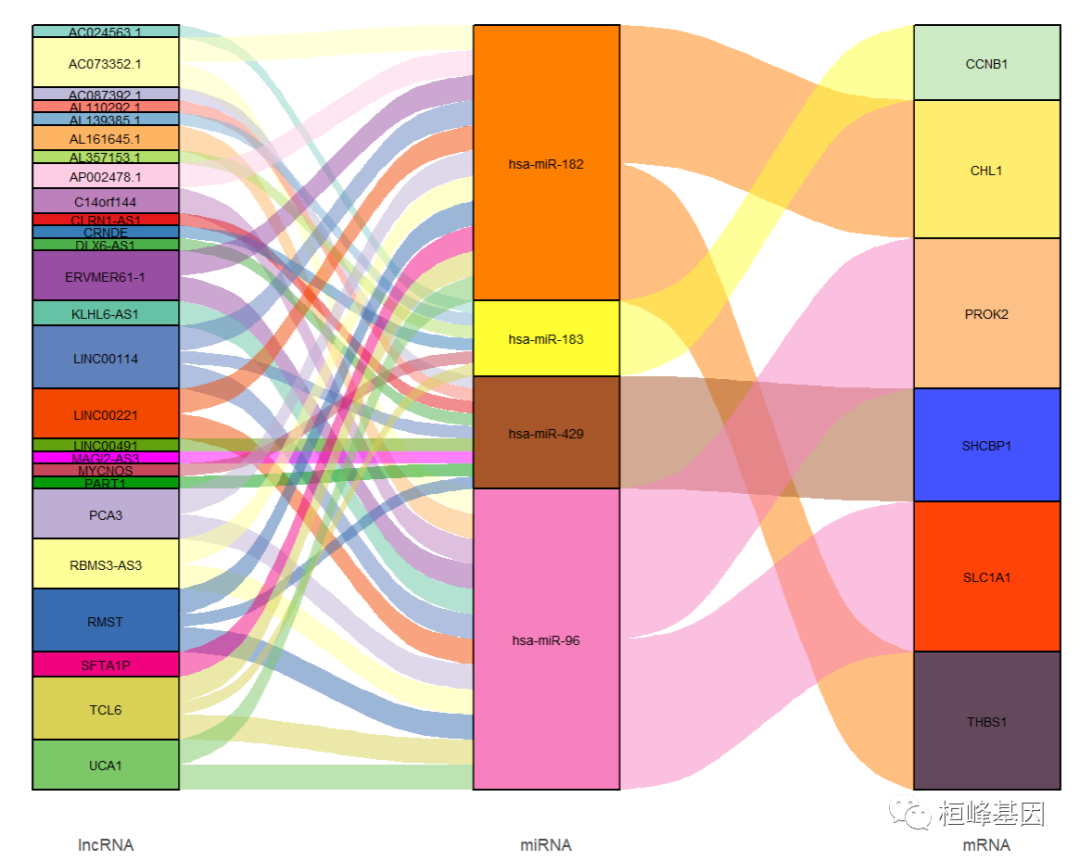
文章复现ceRNA
在ceRNA相关的研究中,例如circRNA-miRNA-mRNA,或者lncRNA-miRNA-mRNA的靶向关系图谱,一般通过网络图呈现。首先可以肯定的是,网络图是一种清晰展示ceRNA网络结构的有效方法。我们选择一篇文章:
肝癌中Fig.2 ceRNA网络的桑基图。每个矩形代表一个基因,根据矩形的大小将每个基因的连接程度可视化,如下
准备数据
在原文献附录中,作者提供了上图lncRNA-miRNA-mRNA靶向关系的原始数据,可以直接在原文中获取,下载链接:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6458652/bin/12935_2019_817_MOESM4_ESM.docx
为了方便大家操作,我们已经下载了数据做了本地整理,如下:
ceRNA <- read.delim('lncRNA_mRNA.txt', sep = '\t', stringsAsFactors = FALSE)
ceRNA=ceRNA[,c(2,1,3)]
dim(ceRNA)
## [1] 61 3
ceRNA$link <- 1
ceRNA <- reshape::melt(ceRNA, id = 'link')
variable <- summary(ceRNA$variable)
ceRNA$flow <- rep(1:variable[1], length(variable))
head(ceRNA)
## link variable value flow
## 1 1 miRNA hsa-miR-429 1
## 2 1 miRNA hsa-miR-183 2
## 3 1 miRNA hsa-miR-182 3
## 4 1 miRNA hsa-miR-183 4
## 5 1 miRNA hsa-miR-96 5
## 6 1 miRNA hsa-miR-182 6
mycol <- c('#8DD3C7', '#FFFFB3', '#BEBADA', '#FB8072', '#80B1D3', '#FDB462',
'#B3DE69', '#FCCDE5', '#BC80BD', '#CCEBC5', '#FFED6F', '#E41A1C', '#377EB8',
'#4DAF4A', '#984EA3', '#FF7F00', '#FFFF33', '#A65628', '#F781BF', '#66C2A5',
'#6181BD', '#F34800', '#64A10E', '#FF00FF', '#c7475b', '#049a0b', '#BEAED4',
'#FDC086', '#FFFF99', '#386CB0', '#F0027F', '#4253ff', '#ff4308', '#D8D155',
'#64495D', '#7CC767')
library(ggalluvial)
ggplot(ceRNA, aes(x = variable, y = link,
stratum = value, alluvium = flow, fill = value)) +
geom_stratum() + #冲击图中的堆叠柱形图
geom_flow(aes.flow = 'forward') + #冲击图连线绘制
scale_fill_manual(values = mycol) + #颜色赋值
geom_text(stat = 'stratum', infer.label = TRUE, size = 2.5) + #添加 lncRNA、miRNA 和 mRNA 标签
scale_x_discrete(limits = c('lncRNA', 'miRNA', 'mRNA')) + #定义 lncRNA、miRNA 和 mRNA 列的展示顺序
labs(x = '', y = '') + #去除 x 轴和 y 轴标题
theme(legend.position = 'none', panel.background = element_blank(),
line = element_blank(), axis.text.y = element_blank()) #去除背景和图例
通过这期教程,您是不是觉得高大上的桑葚图也没有那么难了,跟着桓峰基因学绘图教程,简单轻松,快来关注吧!
桓峰基因,铸造成功的您!
References:
-
Long J, Bai Y, Yang X, et al. Construction and comprehensive analysis of a ceRNA network to reveal potential prognostic biomarkers for hepatocellular carcinoma. Cancer Cell Int. 2019;19:90. Published 2019 Apr 11.
-
Rosvall M, Bergstrom CT (2010) Mapping Change in Large Networks. PLoS ONE 5(1): e8694.