
点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
130篇原创内容
公众号
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.
桓峰基因公众号推出转录组分析和临床预测模型教程,有需要生信的老师可以联系我们!
首选看下转录分析教程整理如下:
RNA 1. 基因表达那些事–基于 GEO
RNA 2. SCI文章中基于GEO的差异表达基因之 limma
RNA 3. SCI 文章中基于T CGA 差异表达基因之 DESeq2
RNA 4. SCI 文章中基于TCGA 差异表达之 edgeR
RNA 5. SCI 文章中差异基因表达之 MA 图
RNA 6. 差异基因表达之-- 火山图 (volcano)
RNA 7. SCI 文章中的基因表达——主成分分析 (PCA)
RNA 8. SCI文章中差异基因表达–热图 (heatmap)
RNA 9. SCI 文章中基因表达之 GO 注释
RNA 10. SCI 文章中基因表达富集之–KEGG
RNA 11. SCI 文章中基因表达富集之 GSEA
RNA 12. SCI 文章中肿瘤免疫浸润计算方法之 CIBERSORT
RNA 13. SCI 文章中差异表达基因之 WGCNA
RNA 14. SCI 文章中差异表达基因之 蛋白互作网络 (PPI)
RNA 15. SCI 文章中的融合基因之 FusionGDB2
RNA 16. SCI 文章中的融合基因之可视化
RNA 17. SCI 文章中的筛选 Hub 基因 (Hub genes)
RNA 18. SCI 文章中基因集变异分析 GSVA
RNA 19. SCI 文章中无监督聚类法 (ConsensusClusterPlus)
RNA 20. SCI 文章中单样本免疫浸润分析 (ssGSEA)
RNA 21. SCI 文章中单基因富集分析
RNA 22. SCI 文章中基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(ESTIMATE) 原创 已修改
RNA 23. SCI文章中表达基因模型的风险因子关联图(ggrisk)
临床预测模型整理如下:
Topic 1. _临床_标志物生信分析常规思路
Topic 2. 生存分析之 Kaplan-Meier
Topic 3. SCI文章第一张表格–基线表格
Topic 4. _临床_预测模型构建 Logistic 回归
Topic 5. 样本量确定及分割
Topic 6 计数变量泊松回归
Topic 7. _临床_预测模型–Cox回归
Topic 8. _临床_预测模型-Lasso回归
Topic 9. SCI 文章第二张表—单因素回归分析表
Topic 10. 单因素 Logistic 回归分析—单因素分析表格
Topic 11. SCI中多元变量筛选—单/多因素表
Topic 12 _临床_预测模型—列线表 (Nomogram)
Topic 13. _临床_预测模型—一致性指数 (C-index)
Topic 14. _临床_预测模型之校准曲线 (Calibration curve)
Topic 15. _临床_预测模型之决策曲线 (DCA)
Topic 16. _临床_预测模型之接收者操作特征曲线 (ROC)
Topic 17. 临床预测模型之缺失值识别及可视化
Topic 18. 临床预测模型之缺失值插补方法
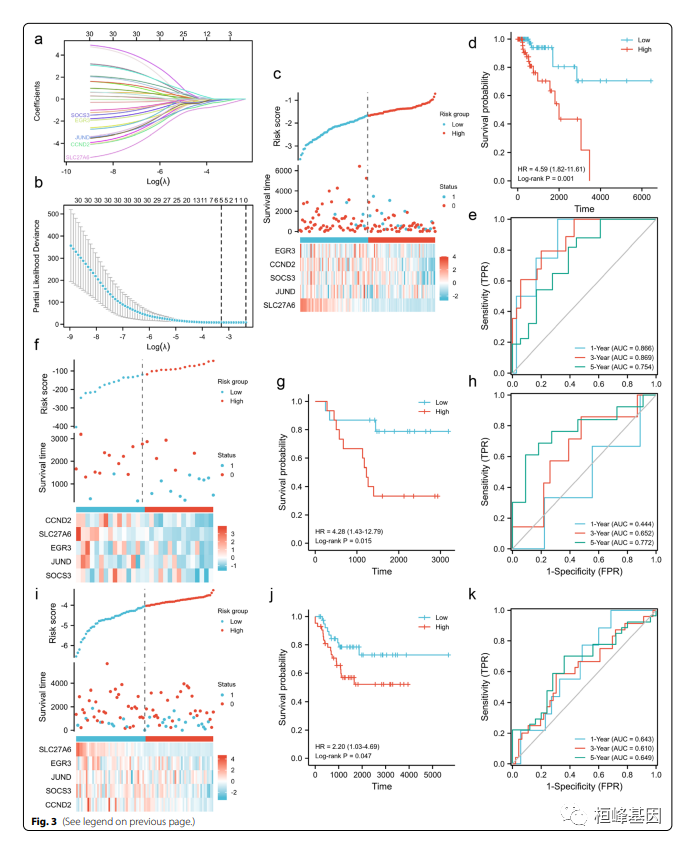
昨天我分享了一篇文章IF:4+ 铁代谢和免疫相关基因标记预测三阴性乳腺癌的临床结局和分子特征,有老师提出问题,说文章中的Fig.3 c,f,i的风险因子关联图该怎么绘制?其实这个现在有现成的软件包,超级简单,我们来看看吧!!!
前言
风险图可能是最常用的图形之一肿瘤基因资料分析。我们可以得出以下两点结论:将模型的预测结果与实际生存情况进行比较观察高危组生存率是否低于低危组,生存时间是否短于低危组。另一种是比较热图和散点图,看看预测因子和结果之间的相关性。风险因子关联图常见于cox风险比例模型,常见于下图这种三图联合的展示形式,展示的是由cox风险模型区分出的高风险和低风险人群在人群比例、生存时间(生/死)和关注的基因表达(通常是参与模型构建的基因)上分布上的差异。当然在模型不涉及基因表达时,往往风险因子关联图仅显示上两部分。
软件安装
在没有出现这个ggrisk包之前,绘图还是需要每个图都分开绘制在组图,现在有了ggrisk软件包就非常方便了,读入数据就可以出结果了,而且ABC图片编号都有了,开不开心?
if (!require(ggrisk)) install.packages("ggrisk")
library(ggrisk)
library(survival)
library(survminer)
数据读取
我们使用ggrisk包内置数据集LIRI来自日本的肝癌ICGC数据库,包含时间、事件和四个基因进行测试。
ICGC Liver Data from Japan Description This data is a liver cancer data from Japan Data released in ICGC database (Link). It cantains time, event and four genes. An object of class data.frame with 232 rows and 6 columns.
data(LIRI)
head(LIRI)
## time status ANLN CENPA GPR182 BCO2
## 1 3.0410959 1 6.821354 3.0366550 0.00000000 0.2248344
## 2 2.5479452 0 1.073527 0.4654169 0.17895040 5.8924860
## 3 4.0273973 0 2.579530 0.7732644 0.06809686 3.5994330
## 4 0.1643836 1 14.183630 7.7239000 0.03749626 1.1194870
## 5 0.8219178 0 3.588320 2.3237710 0.16762610 2.6660850
## 6 2.8767123 0 6.079665 3.6674980 0.21788230 0.7691067
# 使用四个基因构建多因素cox回归模型:
实例操作
1. 构建COX回归
我们使用四个基因构建多因素cox回归模型:
library(rms)
fit <- cph(Surv(time, status) ~ ANLN + CENPA + GPR182 + BCO2, LIRI)
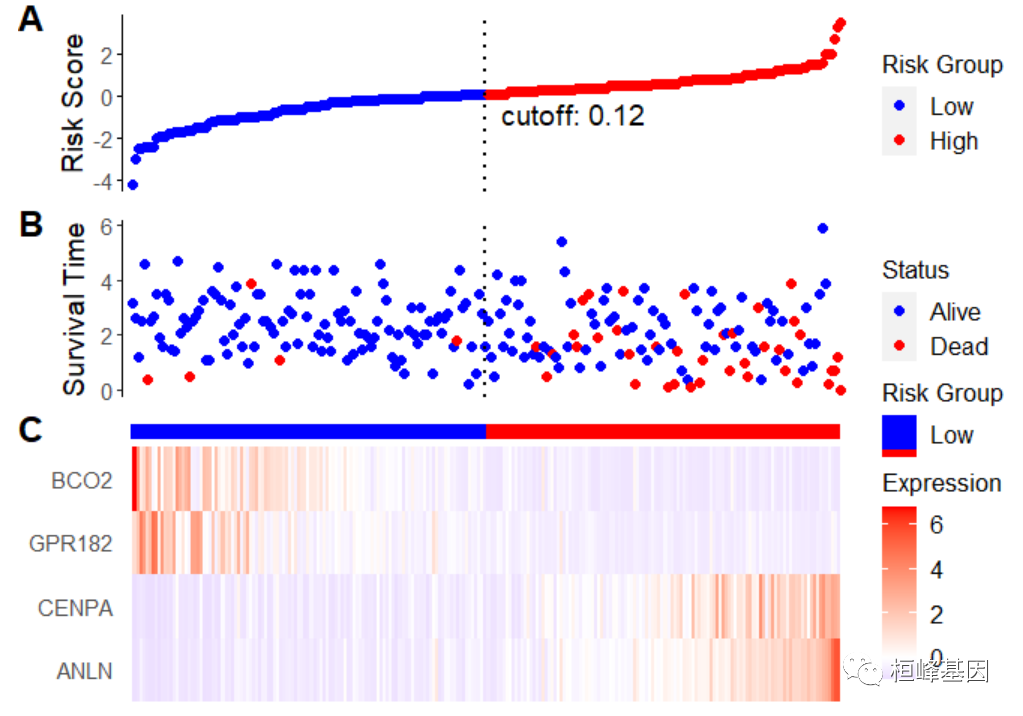
2. 风险因子联动图绘制
ggrisk(fit, cutoff.value = "median", cutoff.x = 145, cutoff.y = -0.8)
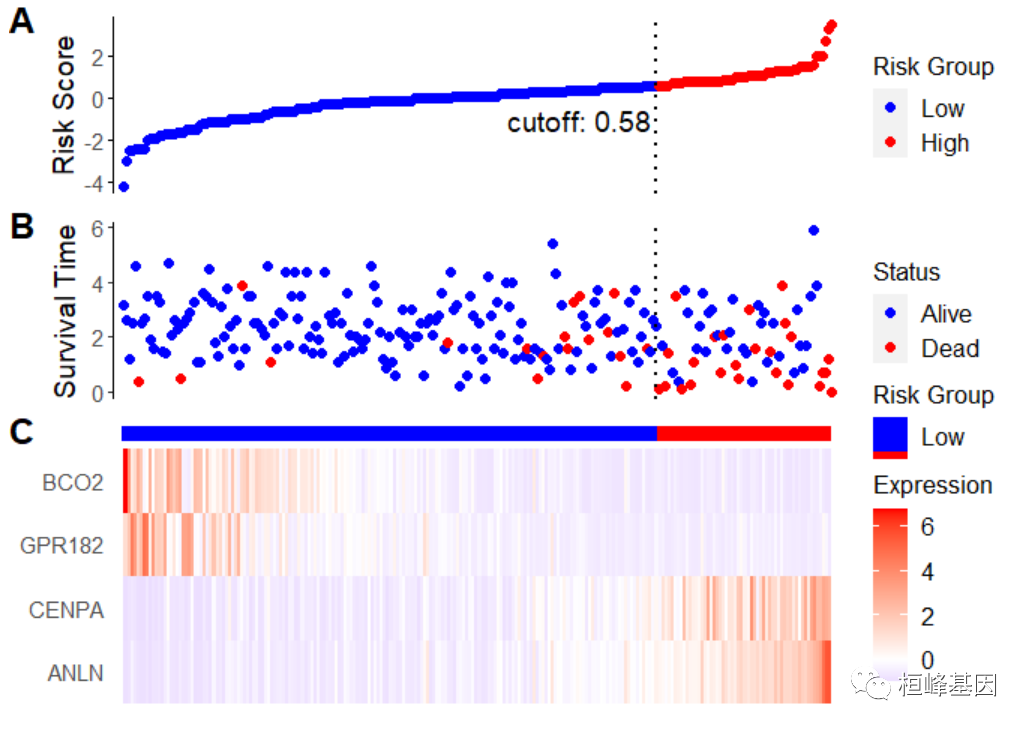
3.调整风险得分的cutoff以及位置
ggrisk(fit,
cutoff.value='cutoff', #可选‘median’, ’roc’ or ’cutoff’
cutoff.x = 150, #“cutoff”文本的水平位置
cutoff.y = -1#“cutoff”文本的垂直位置
)
## --
## ==
## Combination: 185
## Combination: 43
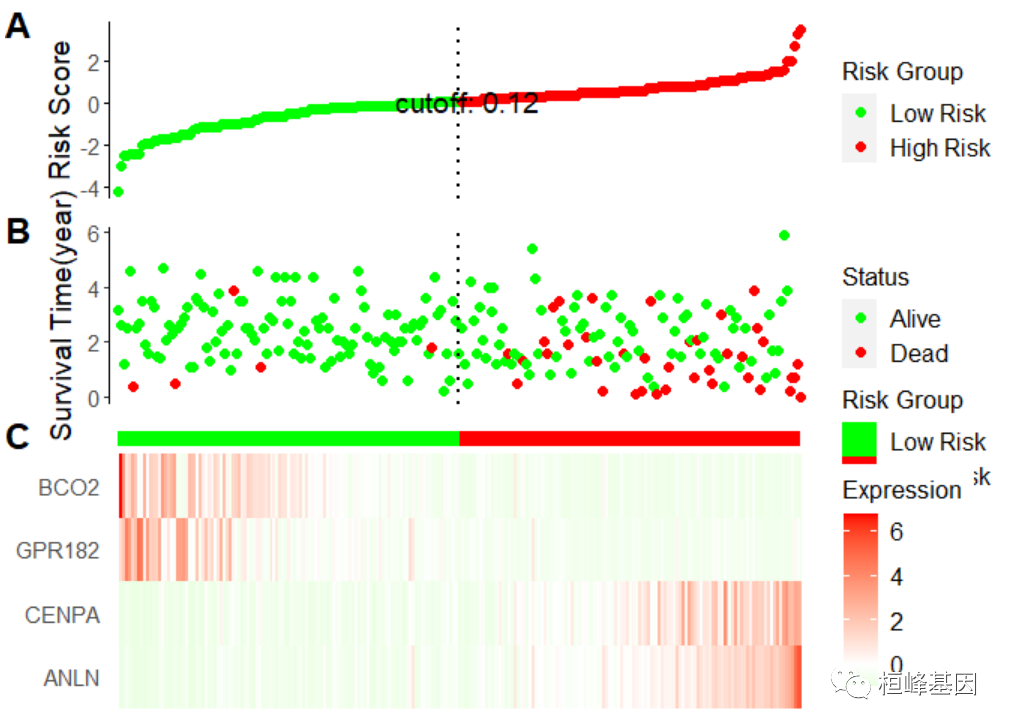
4. 其他参数设置
其他参数设置包括颜色,标签,图例等绘图元素的修改,具体说明可直接使用?ggrisk查看用法。
ggrisk(fit,
code.highrisk = 'High Risk',#高风险标签,默认为 ’High’
code.lowrisk = 'Low Risk', #低风险标签,默认为 ’Low’
title.A.ylab='Risk Score', #A图 y轴名称
title.B.ylab='Survival Time(year)', #B图 y轴名称,注意区分year month day
title.A.legend='Risk Group', #A图图例名称
title.B.legend='Status', #B图图例名称
title.C.legend='Expression', #C图图例名称
relative_heights=c(0.1,0.1,0.01,0.15), #A、B、热图注释和热图C的相对高度
color.A=c(low='green',high='red'),#A图中点的颜色
color.B=c(code.0='green',code.1='red'), #B图中点的颜色
color.C=c(low='green',median='white',high='red'), #C图中热图颜色
vjust.A.ylab=1, #A图中y轴标签到y坐标轴的距离,默认是1
vjust.B.ylab=2#B图中y轴标签到y坐标轴的距离,默认是2
)
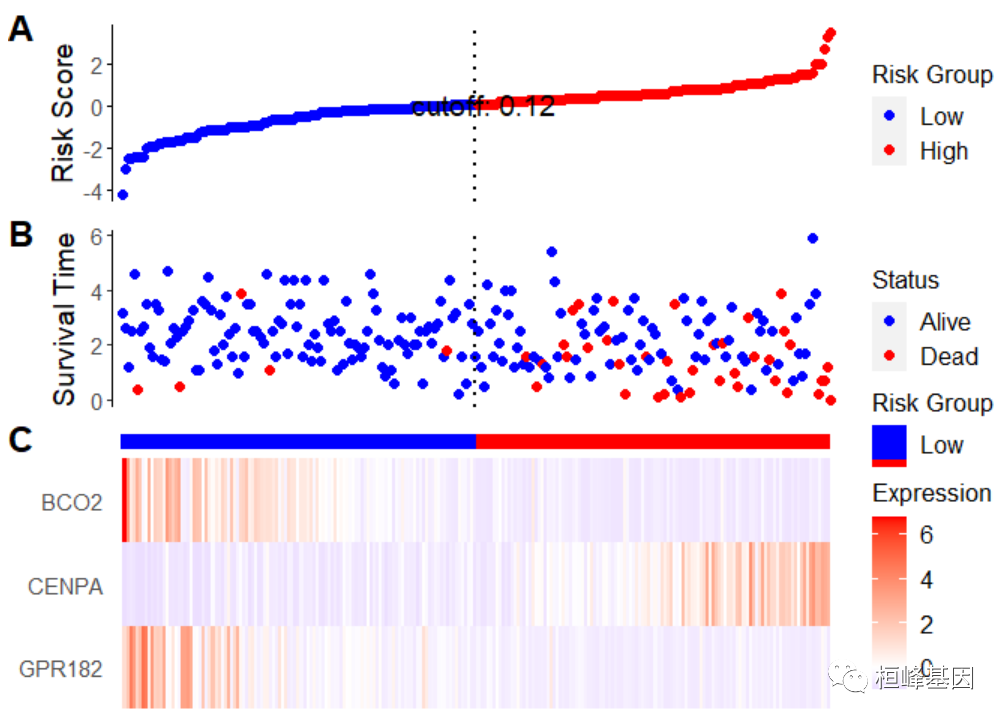
5. 指定热图展示基因
我们也可以指定哪几个基因进行热图的展示,如下:
ggrisk(fit, heatmap.genes = c("GPR182", "CENPA", "BCO2"))
6. 不展示热图
只展示散点图,不展示热图,直接使用two_scatter()函数,如下:
two_scatter(fit, cutoff.value = "median", cutoff.x = 142, cutoff.y = -0.5)
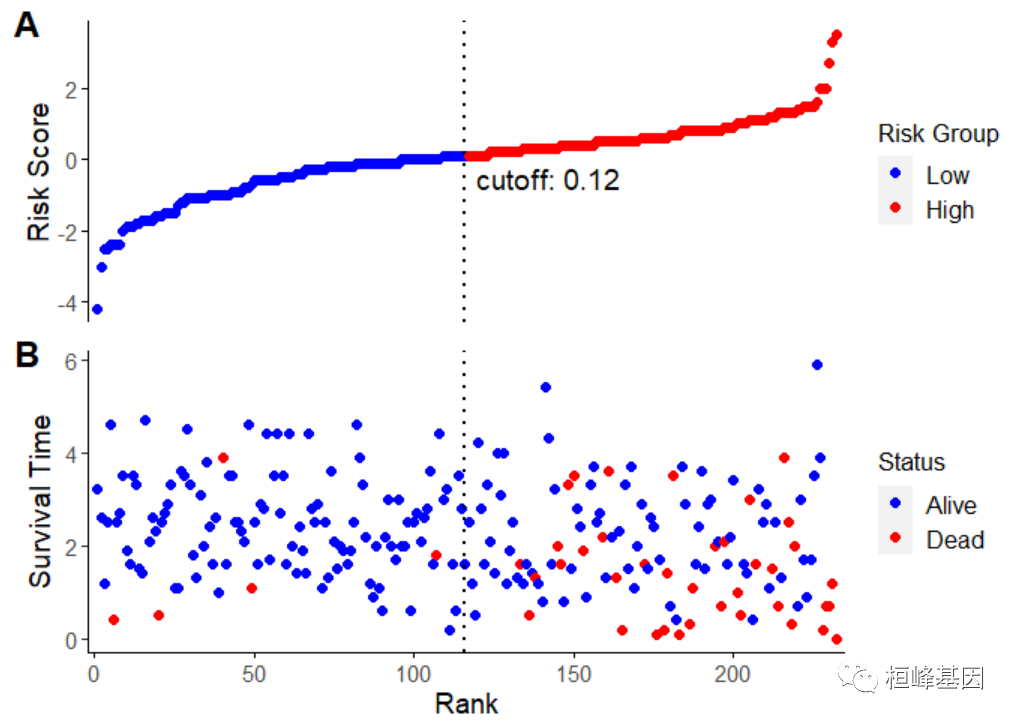
这个是不是非常简单,跟着桓峰基因教程学习,铸造成功的您,记得关注我们哦,如果觉得自己搞生信有难度,快来联系桓峰基因吧!!!
References:
- Li XF, Fu WF, Zhang J, Song CG. An iron metabolism and immune related gene signature for the prediction of clinical outcome and molecular characteristics of triple-negative breast cancer. BMC Cancer. 2022;22(1):619. Published 2022 Jun 7. doi:10.1186/s12885-022-09679-x