论文
De novo assembly, annotation, and comparative analysis of 26 diverse maize genomes

部分数据和代码是公开的,我们今天试着重复一下论文补充材料里的 Figure S29
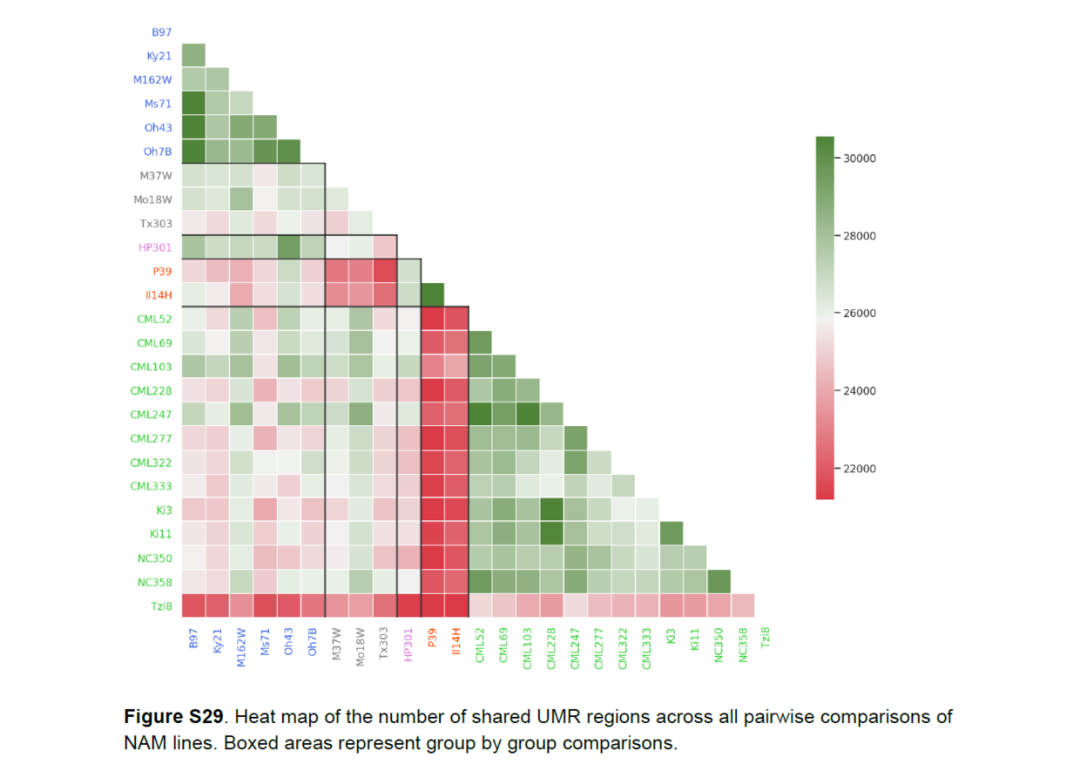
这个热图是用python中的seaborn模块画的,下面介绍画图代码
导入需要用到的模块
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
读入数据集
部分数据截图如下
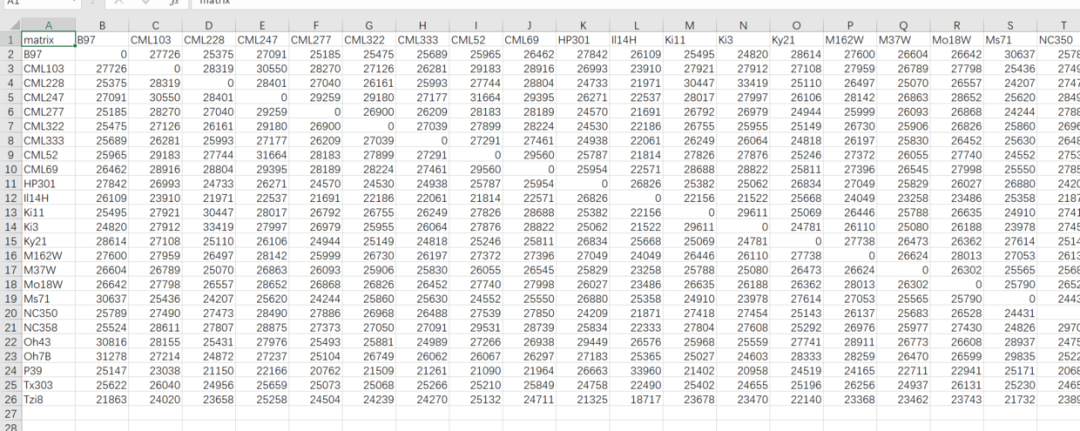
file = "matrix-b73-ref.csv"
b73Ref = pd.read_csv(file, index_col=0).reindex(["B97", "Ky21", "M162W",
"Ms71", "Oh43", "Oh7B", "M37W", "Mo18W", "Tx303", "HP301", "P39",
"Il14H", "CML52", "CML69", "CML103", "CML228", "CML247", "CML277",
"CML322", "CML333", "Ki3", "Ki11", "NC350", "NC358", "Tzi8"])
b73Ref = b73Ref[["B97", "Ky21", "M162W",
"Ms71", "Oh43", "Oh7B", "M37W", "Mo18W", "Tx303", "HP301", "P39",
"Il14H", "CML52", "CML69", "CML103", "CML228", "CML247", "CML277",
"CML322", "CML333", "Ki3", "Ki11", "NC350", "NC358", "Tzi8"]]
这里 index_col=0是用数据集中的第一列来做行名
reindx()函数是将行按照自己制定的内容排序
[[]]是把列按照指定的内容排序
查看数据集的前5行
b73Ref.head(5)
最基本的热图
sns.heatmap(b73Ref)

只保留下三角
这里直接读取的数据集的数据类型是整数型,我们需要把数据转换为浮点型。论文中提供的代码是没有转换数据类型的,如果完全按照他的代码运行可能会遇到报错,这里可能是因为python的版本不同吧,我现在用的python是3.8.3
colnames = ["B97", "Ky21", "M162W",
"Ms71", "Oh43", "Oh7B", "M37W", "Mo18W", "Tx303", "HP301", "P39",
"Il14H", "CML52", "CML69", "CML103", "CML228", "CML247", "CML277",
"CML322", "CML333", "Ki3", "Ki11", "NC350", "NC358", "Tzi8"]
dtype = {}
for colname in colnames:
dtype[colname] = np.float64
df = b73Ref.astype(dtype)
mask = np.triu(np.ones_like(df,dtype=bool))
sns.heatmap(df,mask=mask)
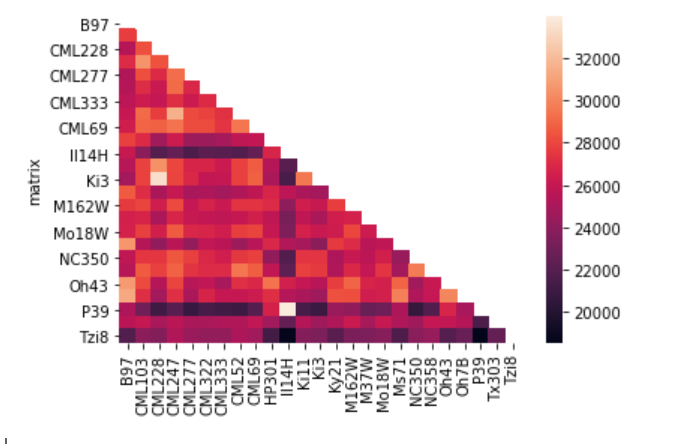
更改配色
cmap = sns.diverging_palette(370, 120, n=80, as_cmap=True)
sns.heatmap(df, mask=mask, cmap=cmap, robust=True,
square=True, linewidths=.5, cbar_kws={"shrink": .5})
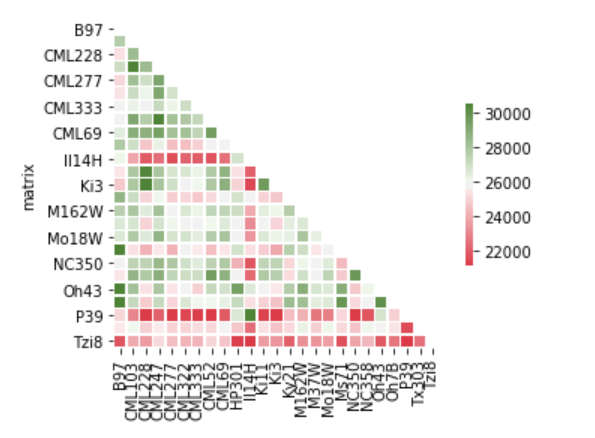
添加辅助线,去掉y轴标题
f, ax = plt.subplots(figsize=(14, 14))
cmap = sns.diverging_palette(370, 120, n=80, as_cmap=True)
sns.heatmap(df, mask=mask, cmap=cmap, robust=True,
square=True, linewidths=.5, cbar_kws={"shrink": .5})
plt.ylabel('')
ax.axvline(x=6, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.76)
ax.axvline(x=9, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.64)
ax.axvline(x=10, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.6)
ax.axvline(x=12, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.52)
ax.axhline(y=6, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.24)
ax.axhline(y=9, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.36)
ax.axhline(y=10, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.4)
ax.axhline(y=12, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.48)
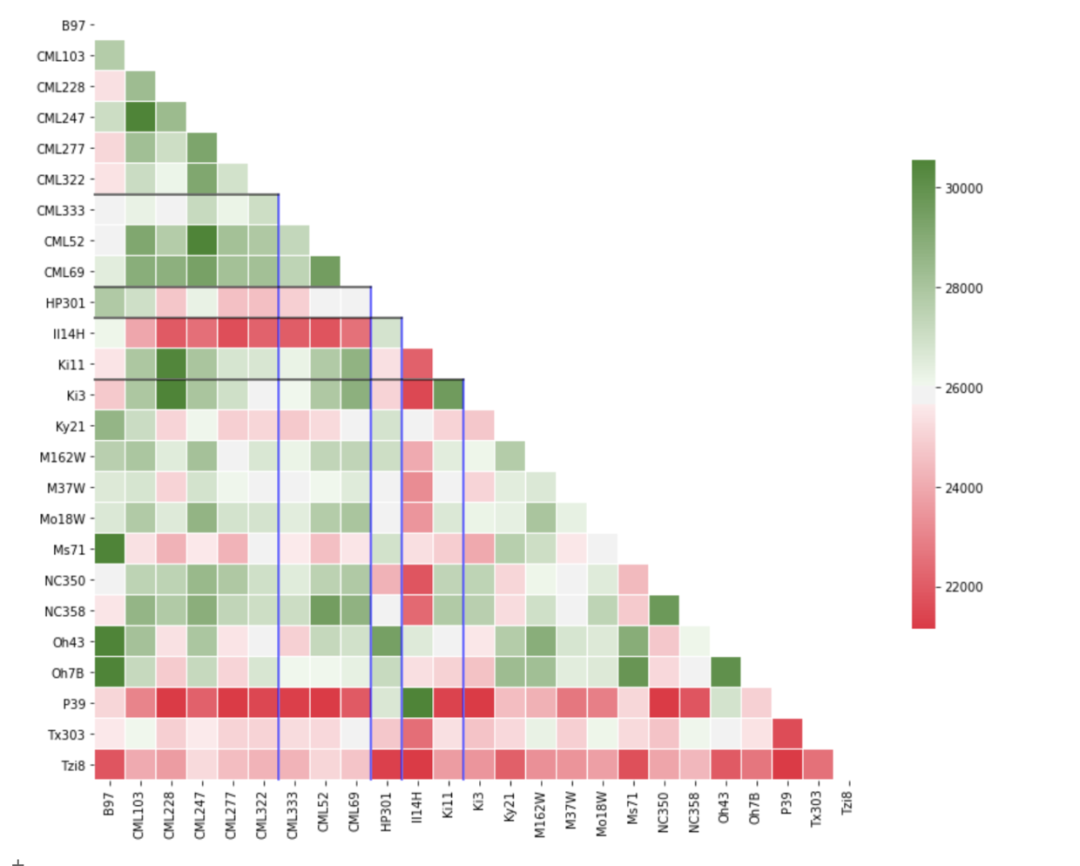
给坐标轴的标签赋予颜色
f, ax = plt.subplots(figsize=(14, 14))
cmap = sns.diverging_palette(370, 120, n=80, as_cmap=True)
sns.heatmap(df, mask=mask, cmap=cmap, robust=True,
square=True, linewidths=.5, cbar_kws={"shrink": .5})
plt.ylabel('')
ax.axvline(x=6, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.76)
ax.axvline(x=9, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.64)
ax.axvline(x=10, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.6)
ax.axvline(x=12, color ='blue', lw = 1.5, alpha = 0.75, ymax = 0.52)
ax.axhline(y=6, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.24)
ax.axhline(y=9, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.36)
ax.axhline(y=10, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.4)
ax.axhline(y=12, color ='black', lw = 1.5, alpha = 0.75, xmax = 0.48)
mycol = ["#4169E1", "#4169E1", "#4169E1", "#4169E1", "#4169E1", "#4169E1", "#787878", "#787878", "#787878", "#DA70D6", "#FF4500", "#FF4500", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32", "#32CD32"]
for tick, color in zip(ax.get_xticklabels(), mycol): tick.set_color(color)
for tick, color in zip(ax.get_yticklabels(), mycol): tick.set_color(color)
plt.savefig("1.pdf")
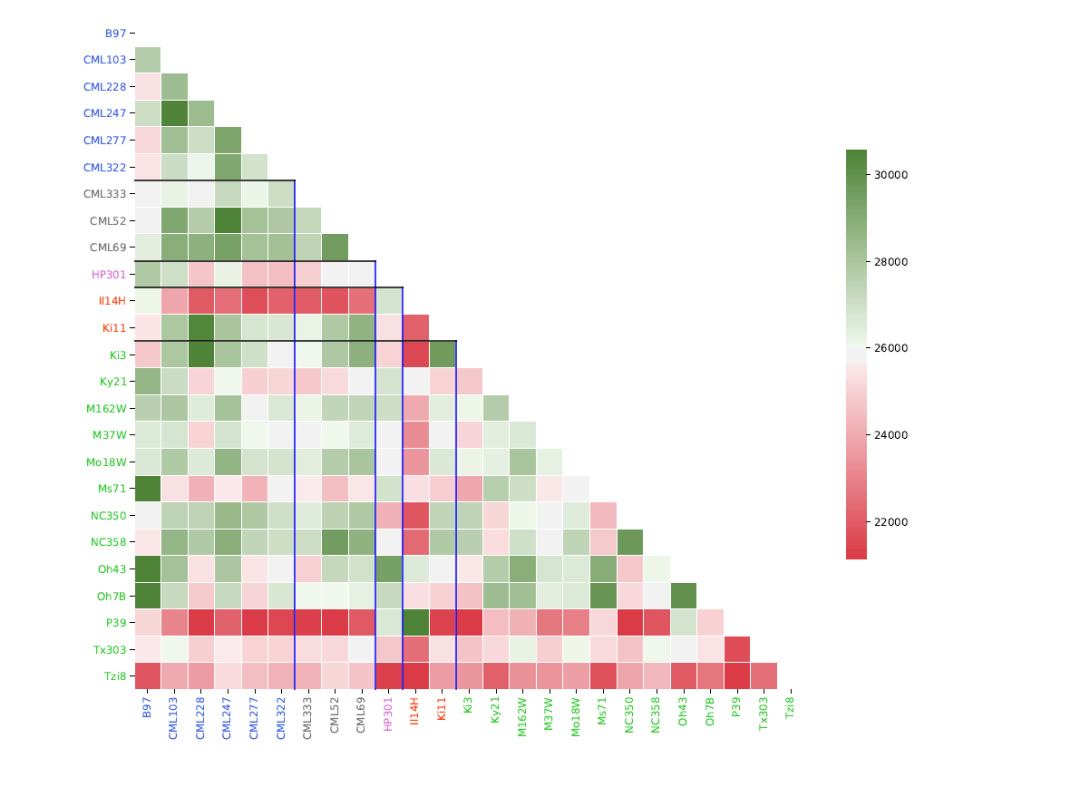
这个是最终的结果
欢迎大家关注我的公众号
小明的数据分析笔记本
小明的数据分析笔记本 公众号 主要分享:1、R语言和python做数据分析和数据可视化的简单小例子;2、园艺植物相关转录组学、基因组学、群体遗传学文献阅读笔记;3、生物信息学入门学习资料及自己的学习笔记!
本文分享自微信公众号 - 小明的数据分析笔记本(gh_0c8895f349d3)。
如有侵权,请联系 [email protected] 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
{{o.name}}
{{m.name}}