问题描述
一个商品推销员要去若干个城市推销商品,该推销员从一个城市出发,需要经过所有城市后,回到出发地。应如何选择行进路线,以使总的行程最短。
对于n个城市的TSP,本文利用python分别实现混合粒子群算法对该问题的求解。
混合粒子群
混合粒子群算法的基本运算过程如下
- 初始化编码:设置最大进化代数T_max、随机生成m个染色体的群体编码
- 适应度函数:对每一个染色体,都有其个体适应度值
- 交叉:将每个个体与该个体的个体极值和当前群体的群体极值进行交叉操作更新,只有交叉后的新个体比旧个体的适应度更好,才替换更改。
- 变异:对单个染色体随机交换两个点的位置,如果变异后的个体比旧个体的适应度更好,就替换更改。
流程图如图

代码
语言:python
第一步初始化参数
import numpy as np
import matplotlib.pyplot as plt
class Hybrid_POS_TSP(object):
def __init__(self, data, num_pop=200):
self.num_pop = num_pop # 群体个数
self.data = data # 城市坐标
self.num = len(data) # 城市个数
# 群体的初始化和路径的初始化
self.chrom = np.array([0] * self.num_pop * self.num).reshape(self.num_pop, self.num)#shape(num_pop,num)
self.fitness = [0] * self.num_pop#一个个体一个适应度函数
# 路径矩阵,函数matrix_dis同遗传算法
self.matrix_distance = self.matrix_dis()
距离函数,加入类Hybrid_POS_TSP
# 计算城市间的距离函数 n*n, 第[i,j]个元素表示城市i到j距离
def matrix_dis(self):
res = np.zeros((self.num, self.num))
for i in range(self.num):
for j in range(i + 1, self.num):
res[i, j] = np.linalg.norm(self.data[i, :] - self.data[j, :]) # 求二阶范数 就是距离公式
res[j, i] = res[i, j]
return res
随机产生初始化群体函数,加入类Hybrid_POS_TSP
# 随机产生初始化群体函数
def rand_chrom(self):
rand_ch = np.array(range(self.num)) ## num 城市个数 对应染色体长度 城市=14
for i in range(self.num_pop): # num_pop # 群体个数 200
np.random.shuffle(rand_ch) # 打乱城市染色体编码
self.chrom[i, :] = rand_ch#.chrom 父代
self.fitness[i] = self.comp_fit(rand_ch)
# 计算单个染色体的路径距离值,可利用该函数更新fittness
def comp_fit(self, one_path):
res = 0
for i in range(self.num - 1):
res += self.matrix_distance[one_path[i], one_path[i + 1]] # matrix_distance n*n, 第[i,j]个元素表示城市i到j距离
res += self.matrix_distance[one_path[-1], one_path[0]] # 最后一个城市 到起点距离
return res
路径可视化函数,加入类Hybrid_POS_TSP
def out_path(self, one_path):
res = str(one_path[0] + 1) + '-->'
for i in range(1, self.num):
res += str(one_path[i] + 1) + '-->'
res += str(one_path[0] + 1) + '\n'
print(res)
两条路径的交叉函数:将每个个体与该个体的个体极值和当前群体的群体极值进行交叉操作。代码加入类Hybrid_POS_TSP
#两条路径的交叉函数:将每个个体与该个体的个体极值和当前群体的群体极值进行交叉操作
def cross_1(self, path, best_path):#path为 个体最优 ,best_path为群体最优
r1 = np.random.randint(self.num)#随机产生小于num的整数
r2 = np.random.randint(self.num)#随机产生小于num的整数
while r2 == r1:#如果两者相等
r2 = np.random.randint(self.num)#重新产生r2
left, right = min(r1, r2), max(r1, r2)#left 为(r1,r2)小者,right 为(r1,r2)大者
cross = best_path[left:right + 1]#交叉片段为 群体最优中的(r1:r2)片段
#下面的for 循环是为了确保 个体染色体(0:(num-(right-left+1)) 片段 不含有 cross中的元素。
for i in range(right - left + 1):#有(r2-r1)次遍历
for k in range(self.num):#遍历个体染色体中的每一个值
if path[k] == cross[i]:#如果当前个体染色体元素path[k] 是cross之内
path[k:self.num - 1] = path[k + 1:self.num] #则修改path[k:num - 1] 片段,同时末尾补0 (也就是说把不属于cross的个体染色体元素 往前赶
path[-1] = 0
path[self.num - right + left - 1:self.num] = cross#把个体染色体(num-(right-left+1):num ) 片段 和群体交叉
return path
变异函数:对单个染色体随机交换两个点的位置。代码加入类Hybrid_POS_TSP
def mutation(self, path):#path 个体染色体
r1 = np.random.randint(self.num)#随机生成小于num的整数
r2 = np.random.randint(self.num)#随机生成小于num的整数
while r2 == r1:#如果r1==r2
r2 = np.random.randint(self.num)#则r2再重新生成
path[r1], path[r2] = path[r2], path[r1]#交换片段
return path
主函数
#data = np.random.rand(20, 2) * 10 # 随机产生20个城市坐标
def main(data, max_n=200, num_pop=200):#迭代次数max_n200 群体个数num_pop=200
Path_short = Hybrid_POS_TSP(data, num_pop=num_pop) # 混合粒子群算法类
Path_short.rand_chrom() # 初始化种群
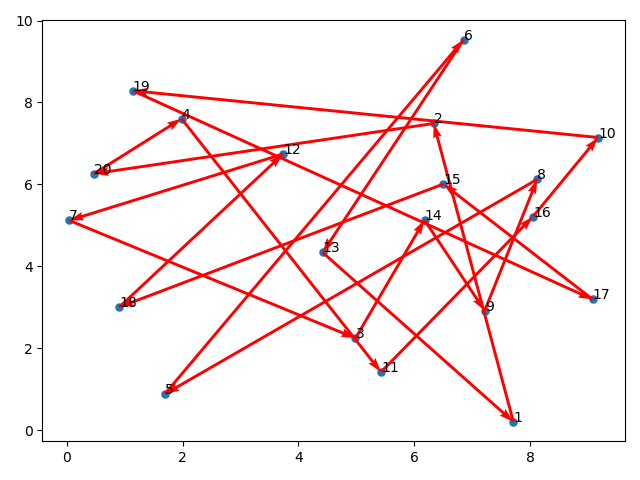
# 初始化路径绘图
fig, ax = plt.subplots()
x = data[:, 0]
y = data[:, 1]
ax.scatter(x, y, linewidths=0.1)
for i, txt in enumerate(range(1, len(data) + 1)):
ax.annotate(txt, (x[i], y[i]))
res0 = Path_short.chrom[0]
x0 = x[res0]
y0 = y[res0]
for i in range(len(data) - 1):
plt.quiver(x0[i], y0[i], x0[i + 1] - x0[i], y0[i + 1] - y0[i], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.quiver(x0[-1], y0[-1], x0[0] - x0[-1], y0[0] - y0[-1], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.show()
print('初始染色体的路程: ' + str(Path_short.fitness[0]))
#定义6个容器 分别存放个体极值,个体染色体,群体最优极值,群体最优染色体,每一次迭代后的最优极值,每一次迭代后最优个体染色体 (容器定义的样子要和染色体个数等一致)
# 存储个体极值的路径和距离
best_P_chrom = Path_short.chrom.copy()# 个体极值 路径 (个体染色体)
best_P_fit = Path_short.fitness.copy()#个体极值 距离
min_index = np.argmin(Path_short.fitness)#最优个体的index序号
#存储当前种群极值的路径和距离
best_G_chrom = Path_short.chrom[min_index, :]#存储当前种群极值的路径(群体最优染色体)
best_G_fit = Path_short.fitness[min_index]#存储当前种群极值的距离(群体最优极值)
# 存储每一步迭代后的最优路径和距离
best_chrom = [best_G_chrom]#当代最优个体
best_fit = [best_G_fit]#当代最优极值
# 复制当前群体进行交叉变异
x_new = Path_short.chrom.copy()
# 进入迭代 更新个体极值,个体染色体,群体最优极值,群体最优个体,存放当代最优个体,存放当代最优极值
for i in range(max_n):#遍历迭代
# 更新当前的个体极值 和路径 #
for j in range(num_pop):#遍历每一个个体
if Path_short.fitness[j] < best_P_fit[j]:#
best_P_fit[j] = Path_short.fitness[j]#更新个体极值
best_P_chrom[j, :] = Path_short.chrom[j, :]#更新个体极值路径
# 更新当前种群的群体极值
min_index = np.argmin(Path_short.fitness)
best_G_chrom = Path_short.chrom[min_index, :]#群体极值 路径
best_G_fit = Path_short.fitness[min_index]#群体极值
# 添加 每一次迭代后的 当代全局最优个体和解极值
if best_G_fit < best_fit[-1]:#best_G_fit 全局极值 best_fit每一步迭代后的最优极值
best_fit.append(best_G_fit)#
best_chrom.append(best_G_chrom)
else:
best_fit.append(best_fit[-1])
best_chrom.append(best_chrom[-1])
#遍历每一个个体,将个体与当代最优个体进行有条件交叉;个体有条件自我变异。条件都为(个体适应度是否更好,如果是则交叉变异
# 将每个个体与个体极值和当前的群体极值进行交叉
for j in range(num_pop):#遍历每一个个体
# 与当代极值交叉
x_new[j, :] = Path_short.cross_1(x_new[j, :], best_G_chrom)
fit = Path_short.comp_fit(x_new[j, :])
if fit < Path_short.fitness[j]:
Path_short.chrom[j, :] = x_new[j, :]
Path_short.fitness[j] = fit
# 变异
x_new[j, :] = Path_short.mutation(x_new[j, :])
fit = Path_short.comp_fit(x_new[j, :])
if fit <= Path_short.fitness[j]:
Path_short.chrom[j] = x_new[j, :]
Path_short.fitness[j] = fit
if (i + 1) % 20 == 0:
print('第' + str(i + 1) + '步后的最短的路程: ' + str(Path_short.fitness[min_index]))
print('第' + str(i + 1) + '步后的最优路径:')
Path_short.out_path(Path_short.chrom[min_index, :]) # 显示每一步的最优路径
Path_short.best_chrom = best_chrom
Path_short.best_fit = best_fit
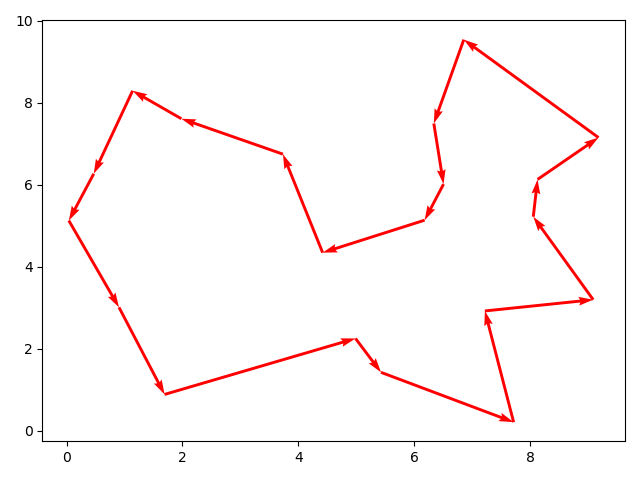
#画出最优路径
res1 = Path_short.best_chrom[-1]
x0 = x[res1]
y0 = y[res1]
for i in range(len(data) - 1):
plt.quiver(x0[i], y0[i], x0[i + 1] - x0[i], y0[i + 1] - y0[i], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.quiver(x0[-1], y0[-1], x0[0] - x0[-1], y0[0] - y0[-1], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.show()
return Path_short # 返回结果类
if __name__ == '__main__':
# 路径坐标
np.random.seed(10)
data = np.random.rand(20, 2) * 10 # 随机产生20个城市坐标
main(data,)
结果:
开始路径
结果路径
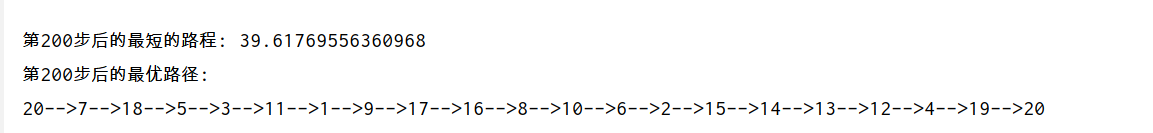

作者:电气余登武。写作不容易,点个赞再走。

