Squeeze-and-Excitation Normalization for Automated Delineation of Head and Neck Primary Tumors in Combined PET and CT Images
挤压激发归一化在PET和CT联合图像中自动勾画头颈部原发肿瘤的研究
摘要
开发健壮、准确、全自动的医学图像分割方法在临床实践和放射组学研究中至关重要。在这项工作中,我们在MICCAI2020头颈部肿瘤分割挑战(HECKTOR)的背景下,提出了一种在正电子发射断层扫描/计算机断层扫描(PET/CT)图像中自动分割头颈部(H&N)原发肿瘤的方法。我们的模型是在具有残留层的U-Net结构上设计的,并补充了压缩和激发归一化。所描述的方法在不同中心的交叉验证(DSC 0.745,精确度0.760,召回率0.789)以及测试集(DSC 0.759,精确度0.833,召回率0.740)上取得了有竞争力的结果,使我们在21个参赛团队中获得了HECKTOR挑战赛的一等奖。基于pytorch的完整实现和经过训练的模型可以在github上获得。
介绍
材料和方法
SE Normalization
我们模型的关键元素是SE归一化层[6],这是最近在脑瘤分割挑战(BRATS 2020)[3]的背景下提出的。类似于实例归一化[4],对于具有N个通道的输入X=(x1,x2,…,xN),SE范数层首先使用平均值和标准差来归一化一批中每个示例的所有通道
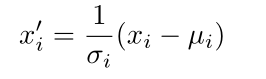
(上式得到的就是归一化值)
其中,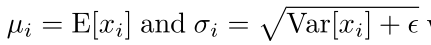
根号下加号后的是一个小常量,以防止被零除。之后,将一对参数γi、βi应用于每个通道,以缩放和移位归一化值
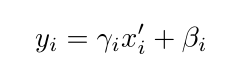
在实例归一化的情况下,在训练过程中拟合的两个参数γi和βi在推理过程中保持不变,并且与输入X无关。不一样的是,我们建议通过SE块来模拟输入X函数的参数γi,βi,即
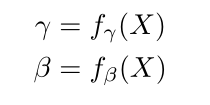
其中,γ=(γ1,γ2,…,γN)和β=(β1,β2,…,βN)-所有通道的比例和移位参数,fγ-具有sigmoid的原始SE块,以及fβ被建模为具有tanh激活功能的SE块以启用负移位(参见图1A)。这两个SE块都首先应用全局平均池(GAP)来将每个通道压缩到单个描述符中。然后,两个全连接(FC)层旨在捕获非线性跨通道依赖关系。第一FC层以缩减率r实现,以形成控制模型复杂性的瓶颈。在本文中,我们使用固定折减比r=2的SE范数层。(应该就是普通的SE block,但是一个的激活函数是sigmoid,一个的激活函数是tanh)

具有SE归一化的层:(A)SE范数层,(B)具有快捷连接的残留层,以及©具有非线性投影的残留层。输出尺寸用斜体表示
网络架构
我们的模型建立在一个开创性的U-Net体系结构[7,8]上,使用了SE归一化层[6]。形成模型解码器的卷积块是3×3×3卷积和ReLU激活的堆栈,随后是SE归一化层。编码器中的残差块由具有快捷连接的卷积块组成(参见图1B)。如果残差块中的输入/输出通道数不同,则通过将1×1×1卷积块添加到快捷方式来执行非线性投影,以便匹配尺寸(参见图1C)。
在编码器中,采用核大小为2×2×2的最大值池化进行下采样;对于解码器中的线性上采样特征映射,采用了3×3×3的转置卷积。此外,通过应用1×1×1卷积块来减少通道数,并利用三线性插值来增加特征地图的空间大小(参见图2,黄色块),我们为解码器补充了三条上采样路径(哪三条,只发现了两条),以进一步传输模型中的低分辨率特征。

具有SE归一化层的模型体系结构。输入由144×144×144体素的PET/CT图像块组成。编码器由带有**标识(实心箭头)和投影(虚线箭头)**快捷键的残差块组成(==实心箭头是图1中的B和虚线箭头是图1中的C ==)。解码器由卷积块构成。附加的上采样路径被添加以在解码器中进一步传输低分辨率特征。在每个块中描述了输出通道的内核大小和数量。(在线彩色插图)
数据预处理和采样
PET和CT图像均采用三线性插值法重采样至1×1×1mm3的共同分辨率。每个训练样本是从整个PET/CT图像中随机提取的144×144×144个体素的补丁,而验证样本是从组织者提供的包围盒中接收的。提取的训练块包含90%的肿瘤,以便于模型训练。
CT强度在[−1024,1024]Hounsfield单位范围内修剪,然后映射到[−1,1]。使用Z-Score归一化对PET图像进行独立变换,并在每个patch上执行。(检查CT强度先裁剪到-1024到1024,然后映射到-1到1,Z-Score通过(x-μ)/σ将两组或多组数据转化为无单位的Z-Score分值,使得数据标准统一化,提高了数据可比性,削弱了数据解释性。)
训练过程
该模型在两个GPU NVIDIA GeForce GTX 1080Ti(11 GB)上使用ADAM优化器训练了800个时期,批次大小为2(每个工人一个样本)。采用余弦退火法,在每25个周期内将学习速率从1e−3降至1e−6。
损失函数
利用soft dice loss[9]和focal loss[10]的未加权和来训练模型。基于[9],一个训练示例的soft dice loss可以写为
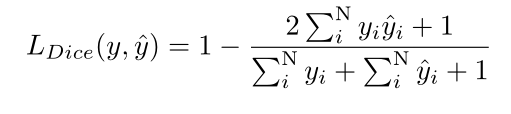
Focal loss定义如下:
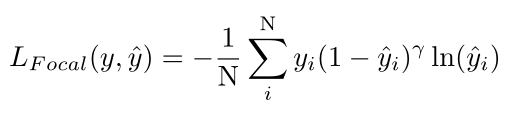
在这两个定义中,yi∈{0,1}-第i个体素的标签,ˆyi∈[0,1]-第i个体素的预测概率,以及N-体素的总数。此外,我们在soft dice loss中的分子和分母上加+1,以避免在训练patch中没有肿瘤类别的情况下的零分割。将focal loss 中的参数γ设置为2。
整体分析
我们在测试集上的结果是使用8个模型组成的集成来产生的,这些模型在不同的训练集分割上进行了训练和验证。采用留一中心交叉验证的方法建立了四个模型,即三个中心的数据用于训练,第四个中心的数据用于验证。在整个数据集的随机训练/验证分割上拟合了其他四个模型。测试集上的预测是通过对各个模型的预测进行平均并应用阈值运算(其值等于0.5)来产生的。
结果和讨论
表1总结了我们在HECKTOR挑战背景下的验证结果。就所有评估指标而言,‘CHGJ’中心有55名患者获得了最好的结果。该模型显示,在整个数据集中表现最差的“CHMR”中心的性能最差。在所有评估指标上,与其他两个中心的差异都很小。所有中心和平均结果之间的小差距意味着模型预测是稳健的,不需要任何特定中心的数据标准化。留单中心剔除和随机分裂交叉验证的平均结果没有显著差异,这一发现得到了支持。表2给出了由来自CHUV中心的53名患者组成的测试集上的集成结果。在之前未见过的数据上,由8个模型组成的集成在21个参赛团队中取得了最高的结果,Dice得分为75.9%,准确率为83.3%,召回率为74%。

不同的交叉验证拆分会产生不同的性能结果。在留单中心剔除交叉验证(前四行)中,为所有中心的每个评估指标提供平均结果(行“Average”)。在相应验证中心的所有数据样本上计算每个度量的平均值和标准偏差。行‘Average(Rs)’表示四个随机数据分割的平均结果。