The Head and Neck Tumor Segmentation Using nnU-Net with Spatial and Channel ‘Squeeze & Excitation’ Blocks
使用带有空间和通道“挤压和激发”模块的nnU-Net进行的头颈部肿瘤分割
Abstract
头颈部(H&N)癌症是癌症死亡的第八大最常见原因。放射疗法是最有效的疗法之一,但它在很大程度上依赖于医学图像上肿瘤体积的轮廓。在本文中,3D nnU-Net首先应用于在氟脱氧葡萄糖正电子发射断层扫描(FDG-PET)和计算机断层扫描(CT)图像中分割H&N肿瘤。此外,我们通过将3D nnU-Net集成到空间和通道的“挤压和激发”(scSE)块中来进行改进,以增强这些有意义的特征,同时抑制较弱的特征。我们将高级3D nnU-Net命名为3D scSE nnU-Net。通过将其分为训练和验证子集,在HECKTOR 2020训练数据上测试其性能,例如训练子集中有160张图像,验证子集中包含41张图像。验证图像上的实验结果表明,在此分割任务的DSC(Dice相似系数)度量方面,拟议的3D scSE nnU-Net优于原始3D nnU-Net 1.4%。我们的3D scSE nnU-Net在HECKTOR测试数据上的DSC为0.735。它在此HECKTOR挑战赛中排名第三。
Introduction
Data and Methods
本节首先简要介绍HECKTOR 2020的数据。然后,它详细描述了我们的方法。我们描述了本文中使用的数据预处理方法。我们还总结了[5]中发布的3D nnU-Net。然后,我们介绍我们提出的3D scSE nnU-Net。接下来,我们描述了我们在实验中使用的训练方案。主要思想是我们采用[5]中的3D nnU-Net作为基线,然后将其推进到3D scSE nnU-Net,从而提高HECKTOR 2020上3D nnU-Net的性能。
Data
本文实验中使用的数据来自HECKTOR2020。训练数据包括来自CHGJ,CHMR,CHUM和CHUS四个中心的201例。测试数据包括来自CHUV另一个中心的53个案例。参加挑战赛时,我们可以获得培训数据。每个训练案例包括NIfTI格式的CT,PET和GTVt(主要肿瘤总体积,即每个案例的标签),以及CSV格式文件中的边界框位置和患者信息。测试数据不包含GTVt信息。
在保留训练数据原始分布的情况下,我们将HECKTOR 2020训练数据分为训练和验证子集。训练子集包含来自201个训练案例的160个训练图像。验证子集包括来自201个训练案例的41个验证图像。训练图像用于训练模型,而验证图像用于估计模型的性能。
Data Preprocessing
为了减少计算量,我们首先将训练数据中的所有图像裁剪到非零值区域。即,如果在CT和PET图像中边缘区域的值均为零,则将裁剪一个患者的图像的相同边缘区域。其次,由于不同的扫描仪或不同的采集协议可能导致数据具有不同的体素间距,因此我们将所有情况重新采样为所有CT图像的中值体素间距,例如0.9765625x0.9765625x3.0 mm,以使HECKTOR训练数据的中间图像形状为147x147x48个体素。最后,为了在训练时加速神经网络收敛,对图像强度进行了归一化。对于CT图像,将收集HECKTOR训练数据中所有CT图像的前景体素,并根据这些值的0.5%和99.5%进行强度值的自动水平窗口式裁剪[5]。对于PET,我们使用z-score归一化对强度值进行归一化。
Baseline:3D nnU-net
3D nnU-Net的体系结构如图1所示。3DnnU-Net的输入图像大小为2x48x 160x160.如果裁剪后的图像的大小小于此输入图像的大小,则边框将填充为0。3DnnU-Net在编码器和解码器的下采样和上采样之间使用两个conv-instnorm-leaky ReLU块。下采样通过跨步卷积完成,而上采样则通过转置卷积完成。该体系结构最初使用30个特征图,对于编码器中的每个下采样操作,该特征图将被加倍(最多320个特征图),而对于解码器中的每个转置卷积则减半(图5)。解码器的末尾后跟1x1x1个卷积将通道数变为2和一个softmax函数,最后输出的特征图具有与输入相同的空间大小。

3D U-Net [7]由于其良好的性能已成为医学图像分割的骨干网络。但是,上采样过程涉及空间信息的恢复,这在不考虑全局信息的情况下很难实现[8]。 scSE块能够自适应地重新校准学习到的特征图的方向,以增强有意义的特征并抑制较弱的特征。因此,我们遵循其编码器和解码器块将scSE块集成到3D nnU-Net中。图2显示了新提出的3D scSE nnU-Net体系结构。

这个scSE块包含两个块,分别是cSE和sSE。对于cSE块,我们将中间输入特征图U = [u1,u2 , uc]表示为通道um∈R^(ZxHxW) 的组合,其中Z,H和W为深度,高度和宽度分别为空间压缩由全局平均池化层完成,以产生向量z∈R^(Cx1x1x1)。其元素在(1)中计算。
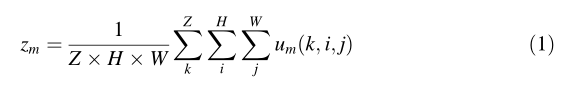
其中um(k,i,j)对应于U在空间位置(k,i,j)的第m个通道的值,其中k∈{1,2,……,Z},i∈{1 ,2,……,H}和j∈{1,2,……,W}

(主要介绍了cSE部分的内容,首先z是经过全局平均池化后的向量,W1、W2分别是两个卷积层的参数,两个卷积层之间有一个ReLU函数,最后的结果经过一个Sigmoid函数,所得向量指示第m通道的重要性,可以按比例放大或缩小,并用于将U重新校准为公式2中的UcSE^)
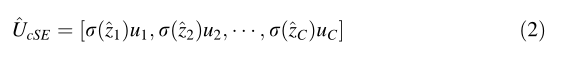
对于sSE块,我们考虑输入特征图U = [u(1,1,1), u(1,1,2),…u(k,i,j),…,u(Z, H, W)] 其中 u(k, i, j)∈R^(Cx1x1x1)。空间压缩操作是通过q = WsqU完成的,其中Wsq∈R(1xCx1x1x1),从而生成投影张量q∈R(ZxHxW)。投影的每个q(k,i,j)表示空间位置(k,i,j)的所有通道C的线性组合表示。然后,q穿过sigmoid()以重新调整激活范围,使其降至[0,1],该值用于将U在空间上重新校准为(3)中的^ UsSE。
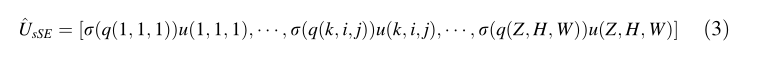
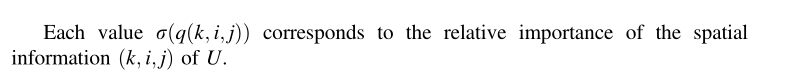
每一个值sigmoid(q(k,i,j))表示了U中空间位置(k,i,j)的相对重要性。(sigmoid(q(k,i,j))和sigmoid(^z1)都是一个比例,还要乘以对应位置的图像值。)
Training Scheme
我们训练我们的网络的组合损失,即Dice损失和加权交叉熵损失在(4)。

这种双重损失受益于交叉熵损失的平滑和有界梯度,以及用于评价的显式优化Dice得分及其对类不平衡的鲁棒性。这里使用的Dice损失是[9]中提出的多类变异损失。计算方法为(5)。
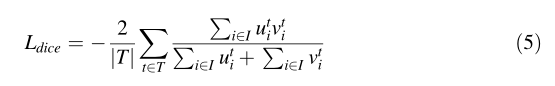
式中,u为网络的softmax输出,v为ground truth的热编码。u和v都有IxT的形状,i∈i, t∈t。其中I为training patch/batch中体素的数量,T为classes的数量。
加权交叉熵损失:LWCE计算如下。在我们的实验中,我们设w = 3.0。
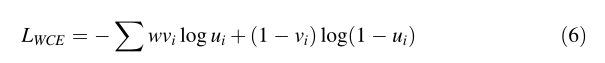
我们使用随机梯度下降优化器来训练我们的网络。初始学习速率是1e-3.在(7)中更新了学习速率。

其中e是epoch,Ne是epoch总数。我们在实验中将Ne设置为1000。我们使用2的批处理大小。模型在250个epoch后收敛。我们还使用权重为5e-5的L2范数正则化对卷积核参数进行正则化。
此外,为了防止在使用有限的训练数据训练大型神经网络时过度拟合,我们采用了以下增强技术,例如随机旋转,随机缩放,随机弹性变形,gamma校正增强和镜像,以在训练期间扩大训练子集数据培训过程。
我们在Pytorch中实施我们的网络,并在具有11 GB内存的单个GeForce RTX 2080Ti GPU上对其进行了培训。单个网络的训练大约需要10个小时。
Results
本部分显示了我们获得的所有实验结果。由于HECKTOR 2020竞赛使用骰子相似性系数(DSC)作为评估指标,因此我们将其作为评估我们训练模型的性能的指标。定量结果总结在表1中,以DSC表示。表2比较了使用我们的3D scSE nnU-Net和原始3D nnU-Net的验证子集中的验证图像的平均推断时间。表3显示了针对HECKTOR 2020挑战赛的竞赛团队测试数据的前5名结果。为了进一步证明我们提出的3D scSE nnU-Net的有效性,我们将其训练损失函数曲线与图3中的3D nnU-Net进行了比较,并在3D scSE nnU-Net的验证数据上显示了一些分割结果以及图4中原始的3D nnU-Net。



关于验证子集和相应DSC值的分割结果的示例。绿线代表地面事实。紫线和黄线分别表示3D nnUnet和3D scSE nnUnet的分割结果。顶行是预测结果良好的示例,底行是预测结果不佳的示例。(在线彩色插图)
Discussions
表1中的结果表明,在DSC度量方面,我们的3D scSE nnUnet的性能优于3D nnUnet。表2中的结果表明,3D scSE nnUnet的平均推理时间比原始3D nnUnet的平均推理时间稍长。这意味着我们要承担推理时间上的损失,以获得更好的目标检测。我们总是在效率和性能之间进行权衡,这通常是一个事实。
图3中的结果表明,在训练模型时,我们的3D scSE nnUnet比原来的3D nnUnet收敛得更快。图4中的结果表明,我们提出的3D csSE nnUnet检测到的目标比3D nnUnet更接近实际目标。我们的3D scSE nnUnet的DSC值比原来的3D nnUnet的相应值要高得多。
图3中的结果表明,在训练模型时,我们的3D scSE nnUnet比原来的3D nnUnet收敛得更快。图4中的结果表明,我们提出的3D csSE nnUnet检测到的目标比3D nnUnet更接近实际目标。我们的3D scSE nnUnet的DSC值比原来的3D nnUnet的相应值要高得多。
Conclusion
在这项工作中,我们提出了一个语义分割网络,称为3DscSE nnUNet,用于从多模式3D图像中分割H&N肿瘤。该3D scSE NNU-Net推进了原始的3D nnU-Net。通过将scSE块集成到原始的3D nnUnet中,实现了H&N肿瘤的自动分割。虽然所提出的3D scSE nnUnet在从多模态3D图像中分割H&N肿瘤的任务上优于原始的scSE nnUnet,但与原始3D nnUnet相比,它需要更多的推理时间。我们将努力提高我们的3D scSE nnUnet在分割头颈部肿瘤时的性能,同时保留其参考时间。此外,我们还将研究不同的数据分区对模型性能的影响,例如留出一个中心作为测试数据。我们希望在HECKTOR 2020挑战之后,能够在头颈部肿瘤分割任务中进一步深入研究。