目录
1、摘要
人们普遍认为,训练一个成功的深度网络需要数以千计的标注好的训练样本。在本文中,提出了一个神经网络及其对应训练策略,其依赖于强数据增强以及对标注样本的更高效的利用。网络结构由一条用于捕获上下文信息的收缩路径和一条对称的用于精准预测的扩展路径组成。我们表明,这样的网络可以从很少的图像端到端训练,并在ISBI挑战中优于之前最好的方法(滑动窗口卷积网络)。使用该网络训练光学显微镜图像(相位对比度和DIC),我们在2015年ISBI细胞跟踪挑战赛的各个类别中以大幅度优势领先。此外,网络速度很快。在最近的GPU上分割512x512图像只需要不到一秒的时间。预训练模型:https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/。
2、网络结构以及相关知识点
总体来说,U-Net扩展了FCN,使得模型可以从很少的数据中学习,且无backbone,属于trained from scratch。
网络结构如图1所示:
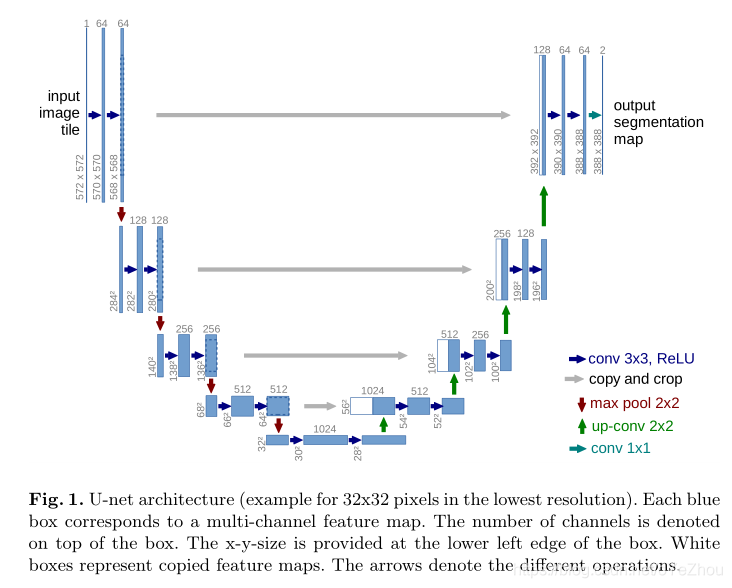
网络结构的细节:
- U-Net包含两个路径——a contracting path (left side) and an expansive path (right side),也即左边的收缩路径和右边的扩展路径;
- 在收缩路径上,每次池化下采样后,都会double feature channel;
- 在扩张路径上,每次上采样之后,都会从当前层和同级的收缩路径中的层中各取一半channel的feature map,crop到同尺寸,然后concat;
- 最后,使用1*1卷积将通道映射到与类别个数相同的数量;
- 相比FCN,U-Net在上采样路径上,使用了更多的feature channel,这使得网络能够将高层语义信息传播到高分辨率的层中;
- U-Net的卷积过程,没有使用padding,所以每次卷积后,featuremap的宽高都会减2。
3、一些策略
在数据增强方面,使用了elastic deformations策略:
elastic deformations:使用了弹性形变数据增强,这对医学图像中细胞之类的内容很有效,因为细胞形变是常有的事;但在其他领域可能就没有什么增强的效果了。
在训练时,U-Net倾向于使用大的输入图,而不使用大的batch,因此对于一张大图,直接训练会倒是GPU显存不够;作者提出了一个overlap-tile strategy来解决这个问题:
overlap-tile strategy:叠层策略,对于一个大图,分成若干小图,对每个小图,扩充其边界然后送入网络进行预测,这样可以利用其周围像素的信息来获取更精准的预测;对于边缘的图像,则使用镜像填充的方式来扩充。
同时,由于每个batch图像个数很少,因此使用了大动量(0.99)来平滑梯度的更新。
此外,为了解决相互接触的细胞的分割,作者提出使用加权损失,即相互接触的细胞之间的背景标签使用了较大的权重。