[论文解读] Non-local U-Net for Biomedical Image Segmentation
非局部Unet在生物医学图像分割中的应用
论文:https://arxiv.org/pdf/1812.04103
代码:https://github.com/divelab/Non-local-U-Nets
https://github.com/Whu-wxy/Non-local-U-Nets-2D-block
数据集(未开源):3D多模态婴儿脑部MR图像
发表:2020 AAAI
一、基本介绍
1.1历史背景
深度学习在各种生物医学图像分割任务中显示出巨大的前景。现有模型通常基于U-Net,并依赖于具有堆叠本地运算符的编码器-解码器架构来逐渐聚合远程信息。然而,仅使用本地操作符限制了效率和有效性。所以提出了用于生物医学图像分割的非局部神经网络,它配备了灵活的全局聚集块。这些块可以作为尺寸保持过程以及下采样和上采样层插入到U-Net中。
1.2 unet的不足
unet编码器均为局部操作,无法整合全局信息,同时下采样丢失空间信息。unet编码器通常会堆叠卷积层与下采样交叉在一起,逐渐减小特征图的空间尺寸。卷积操作、下采样操作都是局部操作,运用小卷积核进行特征提取。通过级联的方式叠加卷积和下采样操作产生较大卷积核,因此能够聚集较大范围信息。由于生物医学图像分割通常受益于广泛的上下文信息,因此大多数模型需要深层的编码器,即堆叠更多的局部操作。这样会引入大量训练参数,尤其是在需要更多下采样的时候,因为通常这样特征映射的通道数会加倍此外,下采样会丢失更多的空间信息,这些信息对于生物医学图像分割至关重要。
unet解码器同样为局部操作,上采样很难恢复全局信息,尤其是无法从编码器有效获取全局信息。unet解码器上采样如反卷积,反池化操作都是局部操作,上采样恢复细节信息需要全局信息。如果不考虑全局信息就很难做到这一点。
1.3基本内容
提出了一个新的U-Net模型,它的推理速度较之以往速度更快,精度更高,参数量更少。提出了一个新的上、下采样方法:全局聚合块,把self-attention和上、下采样相结合,在上/下采样的同时考虑全图信息。
二、网络结构
2.1 解决思路
从Transformer的self-attention入手
作者参考的是2018 cvpr的一篇论文non-local neural networks for video classification
(非局部神经网络在视频分类中的应用)
论文链接:https://arxiv.org/pdf/1711.07971v1.pdf
代码链接:https://github.com/facebookresearch/video-nonlocal-net
2.2结构详解
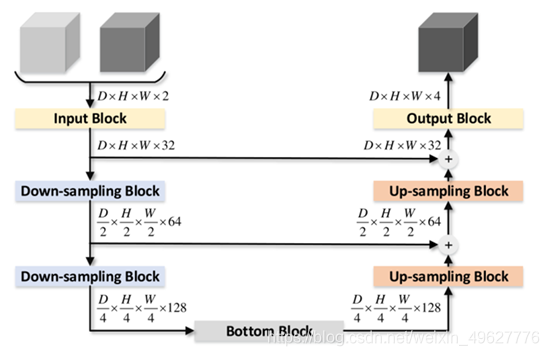
网络使用的3d结构,上图示例输入为两个通道,输出为4类,每一次下采样通道加倍,反之上采样通道减半,短连接采用相加操作。
输入将会首先通过输入块,将会提取出低维特征。两个下采样模块将会用来减少空间尺寸并且用来提取高维特征。在每次下采样过后通道的数量加倍,然后一个底部挡块(bottom block)用来汇聚全局信息并且产生编码器的输出。相对应的,解码器用两个上采样块来恢复空间信息,每次上采样过后,通道数量减半。
上采样部分,通过跳跃连接来将编码器部分的下采样的特征图复制到解码器部分。与其他模型不同的是,这里的复制的特征图通过加法与解码器部分的特征图相结合,而不是串联。采用加法有两点好处:
①.跳跃连接不会增加特征图的数量,从而减少了训练参数
②.相加的跳跃连接可以视作残差连接, 可以促进网络的训练
2.3 Residual Blocks(剩余块)
残差连接被证明可以促进深度学习模型的训练并表现得更好。unet网络中使用的加法连接相当于长范围的残差链接。为了更好地提升unet网络,提出了加入短程残差链接的想法,然而这些想法并没有被应用于下采样和上采样块的残差链接。带有残差链接的下采样模块在ResNet里面已经有所提及。我们研究了基于我们提出的上采样全局聚合块的上采样块。
在作者提出的模型中,用四种不同的残差块来形成一个完整的残差网络
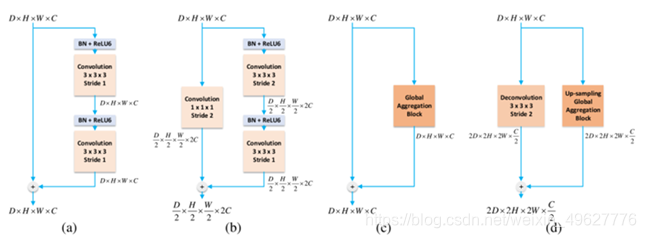
a.regular residual block
标准残差模块,在跳跃连接求和后插入这样的模块
b.down-sampling residual block
下采样残差模块,下采样时使用
c.bottom block
底部模块,使用global aggregation block
d.up-sampling residual block
上采样模块,上采样时使用
(a)展示了一个常见的有着两个连续卷积层的剩余块。在每个卷积层之前使用了BN和Relu6 激活函数。这个模块在我们的网络中作为输入块。输出块则是在该模块后面加入了一个333,步幅为1的卷积层。此外,我们在每个加法的跳跃连接过后也加入一个这个模块。
(b)是一个下采样的残差模块。使用一个步长为2的111的卷积来代替原先的残差链接,是为了调整特征图的空间尺寸。
(c)描述了我们的底部模块。主要采用全局聚合块。
(d)和下采样模块(b)相似,只不过换成了步长为2的333的反卷积,另外一个分支换成了上采样全局聚合块。我们将这个模块作为上采样块。
2.4 Global Aggregation Block(全局聚合块)
为了融合全局信息,输出的特征图的每个位置,都应该取决于输入的特征图的每个位置。这样的操作和像卷积和反卷积一样的局部操作不同,它们每个输出的位置都在输入上有一个局部的感受野。实际上,全连接网络有这样的全局性质,然而这很容易过拟合并且实际中表现并不好。Transformer通过考虑输入的每一个位置来计算输出的一个位置。研究都采用self-attention块来捕捉序列的长期依赖信息,因此我们指出图像特征图的全局信息可以通过self-attention 块来进行汇集。
基于这个观点,作者提出了全局聚合块可以从任意尺寸的特征图来获取全局信息。将其推广到下采样和上采样的过程中,让它变成一个在深度学习模型任意位置都可以使用的块。
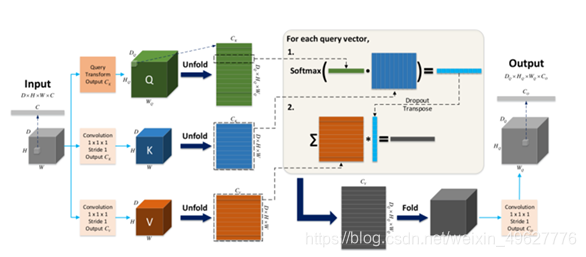
x,y: X来表示输入,Y表示输出
Conv_1N:步长为1 输出通道数为N的卷积
Unfold(·):将D × H × W × C的张量展开为(D ×H ×W)×C的矩阵
QueryTransformCK (·): 生成Ck个feature maps(特征图)的任何操作
CK, CV:是表示键和值的维度的超参数
K,V: 假设X的尺寸为D×H×W×C,则K和V的尺寸分别为(D×H×W)×Ck和(D×H×W)×Cv
第一步,生成query (Q), key (K) and value (V ) 矩阵。Unfold之前有Multi-Head操作,Multi-Head将通道拆分,Q的维度为(DQ × HQ × WQ) × Ck,
第二步,自注意机制。A为注意力矩阵,用Q点乘K的转置,除以根号Ck,再使用softmax,A的维度为(DQ × HQ × WQ) × (D × H × W)。O的维度为(DQ × HQ × WQ) × CV。
注意力权重矩阵 A 的维度是 (DQ × HQ × WQ) × (D × H × W) 输出矩阵 O 的维度是 (DQ × HQ × WQ) × CV . 为了展示它是如何运作的,我们从Q里面选择一个query vector来举例。在注意力机制中,query vector和所有的key vector相互作用,通过点乘的方式来产生value vector所对应的scalar weight。query vector的输出是一个有权重的value vector的总和,通过softmax来进行归一化。对所有的query vector都采用这个过程,最后生成 (DQ ×HQ ×WQ) Cv的向量。注意图中的dropout可以被用在A上来防止过拟合。
第三步,还原为原维度大小。
Fold的Unfold相对的操作,Co代表输出的维度。最终,Y的输出是 DQ×HQ×WQ×CO。值得注意的是,输出Y的空间大小是由Q矩阵来决定的,也就是通过QueryTransformCK这个操作,也因此全局聚合块可以灵活的适用于保持尺寸,下采样和上采样。在我们提出的non-local U-Nets中,我们令Ck = Cv= Co 并且采用两种不同的QueryTransformCK函数,对于(c )而言,QueryTransformCK是Conv_1Ck。对于上采样全局聚合块(d)而言,QueryTransformCK是一个步长为2的333的反卷积层。这些块的使用,缓解了通过单独的反卷积层来上采样会丢失信息的问题。通过考虑全局信息,上采样模块可以恢复更多的精确信息。
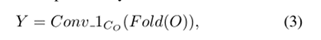
三、方法亮点
1.提出了全局聚合模块global aggregation block,汇聚全局信息得到更加精确的分割图
2.简化unet下采样到4,使其更加高效快速且减少很多参数
3.为了解决以上问题,作者基于self-attention提出了一个非局部的结构,全局聚合块用于在上/下采样时可以看到全图的信息,这样会使得到更精准的分割图。
4.简化U-Net,减少参数量,提高推理速度,上采样和下采样使用全局聚合块,使分割更准确。
四、网络模型主要应用及结果
4.1 实验中使用的图像分割数据集
数据集:
在三维多模态等强度婴儿脑磁共振图像分割任务上进行实验,以评估我们的非局部神经网络。任务是将磁共振图像自动分割成脑脊液、灰质和白质区域。我们首先介绍基线模型和实验中使用的评估方法。然后描述了训练和推理过程。我们提供了有效性和效率方面的比较结果,并进行消融研究,以证明我们的非局部U-Net中的每个全局聚合块如何提高性能。此外,我们探讨了基于不同重叠步长的推理速度和准确性之间的权衡,并分析了补丁大小的影响。
(将MR图像分割成CSF,GM,WM三种区域
Baseline:使用CC-3D-FCN作为基线。CC3D-FCN是一种三维全卷积网络(3D- fcn),它具有卷积和连接(CC)跳跃连接,是为三维多模等强度婴儿大脑图像分割而设计的。)
4.2“留一”交叉验证(K折交叉验证)
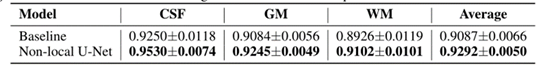
比较提出的模型和基线模型的分割性能。使用“留一”交叉验证。数值越大表示性能越好。
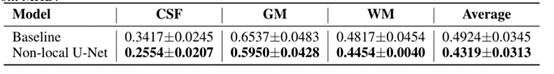
提出的模型与基线模型在3D-MHD分割性能的比较。使用了“留一”交叉验证。数值越小,性能越好。注意,3D-MHD给出的结果与MHD不同。(3D-MHD和MHD具体参考论文)
4.3在iSeg-2017中13个测试对象的分割性能比较
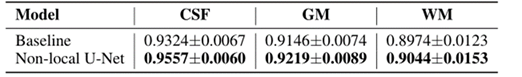
提出的模型与基线模型在iSeg-2017中13个测试对象的分割性能比较,数值越大,分割性能越好。
4.4参数比较

提出的模型和基线模型之间参数数量的比较。(与3DFCN相比,我们的模型减少了28%的参数,获得了更好的性能)
4.5推理时间
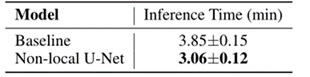
提出的模型和基线模型之间推理时间的比较
4.6分割结果可视化
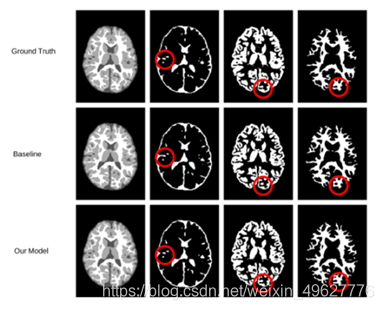
提出的模型和基线模型分割结果的可视化。
第一列显示原始分割图。第二、第三和第四列分别显示了CSF、GM和WM的二进制分割图。
4.7消融实验
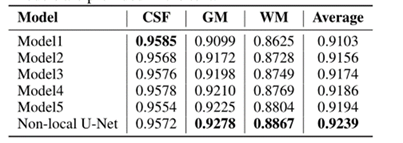
消融研究,以显示非局部Unet各部分的有效性
不同模型在分割性能进行消融研究。所有模型在前9名受试者中进行训练,在第10名受试者中进行评估。值越大表示性能越好。
模型的详细信息:1~5
3D U-Net 无短连接
3D U-Net 有短连接
用Residual Blocks(剩余块)中的d块(上采样块)代替模型2中的第一个上采样块。
用Residual Blocks(剩余块)中的d块(上采样块)代替模型2中的2个上采样块。
用Residual Blocks(剩余块)中的c块(底部模块)代替模型2中的底部块
六、个人思考总结
从transformer的self-attention中找到灵感,深度学习各领域其实是相互连接的,不同领域的优秀方法也值得我们去学习借鉴。
总结:
提出了用于生物医学图像分割的非局部U-网。如前所述,现有的基于U-Net的模型没有一种高效且有效的方法来通过仅使用堆叠的局部算子来聚集全局信息,这限制了它们的性能。为了解决这些问题,提出了一个全局聚合块,它可以灵活地用于U-Net中的大小保持、下采样和上采样过程。在3D多模态等强度婴儿脑磁共振图像分割任务上的实验表明,在全局聚集块的情况下,局部U网以更少的参数和更快的计算速度明显优于以前的模型。